猪13号染色体上拷贝数变异区内基因信息发掘及遗传规律分析
刘静, 王亚楠, 孙亚奇, 王洪洋, 汪超, 彭中镇, 刘榜
华中农业大学动物科技学院, 农业动物遗传育种与繁殖教育部重点实验室, 武汉 430070
猪13号染色体上拷贝数变异区内基因信息发掘及遗传规律分析
刘静, 王亚楠, 孙亚奇, 王洪洋, 汪超, 彭中镇, 刘榜
华中农业大学动物科技学院, 农业动物遗传育种与繁殖教育部重点实验室, 武汉 430070
拷贝数变异(Copy number variation, CNV)是染色体上发生的一种微结构变异, 已引起越来越多研究者的关注。本课题组前期已获得猪13号染色体上的32个CNV区域(CNV region, CNVR), 为了发掘CNVR内的基因信息, 文章在线检索了上述CNVR内的基因并进行基因本体(Gene Ontology)分析。结果共发现236个基因, 其中有注释基因169个, 主要参与蛋白质水解、细胞粘附、大分子降解等生物过程。为了探索这些基因拷贝数变异的遗传规律, 文章选择RCAN1(Regulators of calcineurin 1)基因为候选基因, 利用QPCR方法在莱芜猪群中检测了该基因的拷贝数, 并分析了CNV在莱芜猪3个家系中的遗传规律。结果表明, RCAN1基因在莱芜猪群体中存在拷贝数的缺失、重复现象, 其拷贝数变异的遗传规律符合孟德尔遗传方式。
拷贝数变异; 13号染色体; RCAN1基因; 遗传规律; 莱芜猪
拷贝数变异(Copy number variation, CNV)是指与生物正常的基因组序列(或参考基因组序列)相比,基因组中所发生的长度在1 kb至数Mb之间的变异,其形式包括重复、缺失、插入、易位及衍生出的染色体结构变异[1]。由于CNV长度跨度较大, 以致它可以包含一个基因或基因的一部分, 也可以跨越多个基因, 在基因组中覆盖的核苷酸总数远远超过单核苷酸多态性(Single nucleotide polymorphism, SNP)总数。研究发现, CNV可以通过剂量效应改变基因的表达量[2], 也可以影响转录调控从而影响基因的表达[3,4], 它对表型变异的潜在影响可能要比SNP的影响更大。本课题组余少波等[5]利用猪60K SNP芯片技术在中外9个猪品种和1个杂种群体(杜洛克猪×通城猪)中发现了大量拷贝数变异区域(Copy number variation region, CNVR), 绘制了中外猪品种的全基因组CNV图谱, 发现猪13号染色体(Sus scrofa chromosome 13, SSC13)上存在32个CNVR。其中莱芜猪尤其特殊, 在13号染色体上存在大范围的染色体缺失或重复区域, 但是未对SSC13上32个CNVR存在的真实性、它们的分类与长度分布、具有Ensembl ID基因与注释基因的个数及拷贝数变异的遗传规律进行探究。
本研究在课题组前期研究基础上, 分析计算SSC13上CNVR的分类及长度分布; 利用生物信息学方法检索SSC13上32个CNVR内的基因并分析其功能; 选择钙调神经磷酸酶调节因子1(Regulators of calcineurin 1, RCAN1)基因为候选基因, 利用QPCR方法在 38头莱芜猪中检测该基因的拷贝数,并在该品种3个家系共15个个体中对该基因拷贝数变异进行遗传规律分析。
1 材料和方法
1.1 材料
检测RCAN1基因拷贝数所用DNA样品来自山东省莱芜市莱芜猪原种场38头莱芜猪的耳组织, 并用杜洛克猪作为参考基因组。
莱芜猪的A、B、C 3个家系用于分析RCAN1基因拷贝数变异的遗传规律。A、B家系均包含父本、母本及3个子代个体, C家系包含父本、母本及4个子代个体。B、C为同父异母的半同胞家系。
1.2 猪13号染色体上CNVR内基因的发掘与其功能预测方法
利用 Ensembl中的 BioMart工具(http://asia. ensembl.org/biomart/martview/)检索SSC13上CNVR内的基因信息。应用基因本体(Gene Ontology, GO)数据库(http://david.abcc.ncifcrf.gov/)将猪 CNVR内的基因与人的基因进行同源比对, 根据同源基因产物的生物学功能、所参与的生物学过程等信息, 预测SSC13上CNVR中的基因功能。
1.3 利用 QPCR方法检测莱芜猪 13号染色体上RCAN1基因的拷贝数
通过检索SSC13上CNVR内的基因, 发现RCAN1基因位于 CNVR内, 并且其功能与免疫相关, 但是猪中尚无该基因的研究报道, 本研究利用 QPCR方法在38头莱芜猪群体中检测RCAN1基因的拷贝数。
1.3.1 引物设计
从Ensembl数据库(http://asia.ensembl.org/index. html)中获取RCAN1基因(ENSSSCG00000012050)的全长序列, 用Primer5.0设计4对引物, 引物位于该基因的上游和下游(表1)。10 μL反应体系包括:1 μL DNA模板, 上下游引物各0.2 μL, Taq DNA聚合酶(Biotium公司)0.1 μL, dNTP混合物0.3 μL, PCR缓冲液1 μL, 加双蒸水至10 μL。PCR扩增程序:95℃ 5 min; 94℃ 30 s, 60℃ 30 s, 72℃ 15 s, 34个循环; 最后再72℃延伸5 min。取4 μL PCR扩增产物, 用2%琼脂糖凝胶电泳检测并回收目的片段, 通过测序验证引物的扩增准确性。
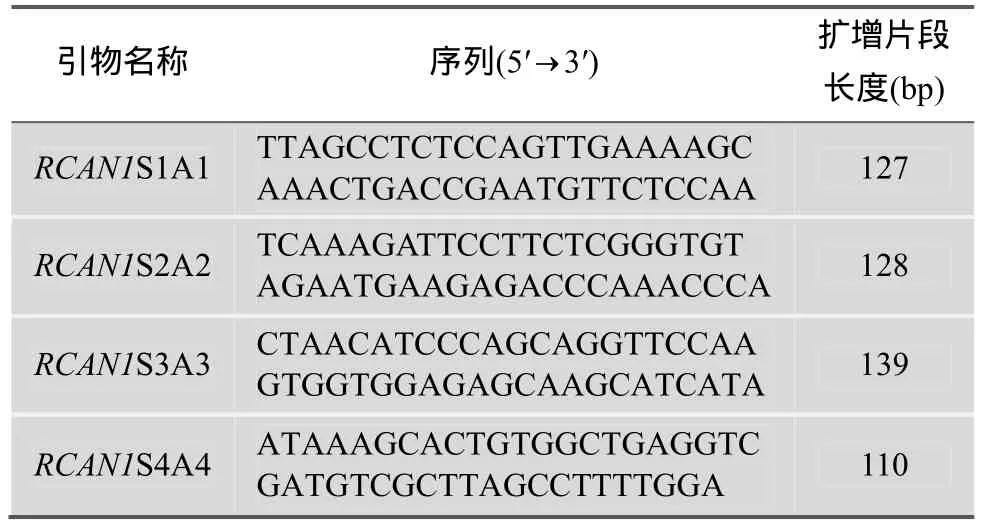
表1 RCAN1基因PCR扩增引物信息
1.3.2 QPCR扩增
QPCR采用SYBR Green相对定量的方法, 以莱芜猪基因组 DNA为模板, 以胰高血糖素(Glucagon, GCG)基因为内参基因[6], 用RCAN1S1A1扩增RCAN1基因, 每个个体 3次重复。反应体系按照 Real-time PCR Master Mix (TOYOBO, Japan)说明书进行, 反应体积为10 μL。扩增程序:95℃ 5 min; 94℃ 30 s, 60℃30 s, 72℃ 15 s, 34个循环; 最后再72℃延伸5 min。
1.3.3 RCAN1基因拷贝数的估计
以 2-ΔΔCt为基础, 利用公式:拷贝数=21-ΔΔCt对QPCR结果进行计算, 采用四舍五入方法对基因拷贝数进行估计。正常个体中, RCAN1基因拷贝数与内参基因相同, 即 2个拷贝; 具有 CNV的个体中,该基因拷贝数会发生增加或缺失。
1.4 RCAN1基因拷贝数变异的遗传规律分析
以CNVR内的RCAN1基因为候选基因, 以3个莱芜猪家系为材料, 利用 QPCR方法检测该基因拷贝数, 分析每条染色体上可能含有的拷贝数情况,判断RCAN1基因的拷贝数变异在3个莱芜猪家系中是否符合孟德尔遗传规律。
2 结果与分析
2.1 猪13号染色体上CNVR的分类与长度分布
本课题组前期研究获得了 SSC13上的 32个CNVR, 其中16个是重复区域, 10个为缺失区域, 6个同时存在重复和缺失的区域[5]。本研究计算结果表明:所有CNVR全长45 722 kb, 占该染色体总长的20.9%,平均每个CNVR长1428 kb, 最大的可达12 411 kb,最小的仅27.6 kb, CNVR的中值为250.1 kb。
2.2 猪13号染色体CNVR中的基因注释及其功能分析
使用Ensembl中的Biomart工具检索本课题组前期获得的SSC13上32个CNVR内的基因, 发现具有Ensembl ID的基因共236个, 其中注释基因169个(占71.6%)。CNVR中, 基因的密度为5.16/Mb。
将上述 236个基因与人类基因进行同源比对,发现其中的 180个基因在人基因组中进行过注释。GO分析发现这些基因主要参与蛋白质水解(Proteolysis)、细胞粘附(Cell adhesion)、大分子降解(Macromolecule catabolic process)等生物学过程, 并具有调节酶活性(Peptidase activity)、离子通道活性(Ion channel activity)的生物学作用(图1)。
2.3 猪13号染色体上RCAN1基因的拷贝数检测结果
经过BLAST(http://blast.ncbi.nlm.nih.gov/Blast.cgi)分析, 参考基因组上无其他与RCAN1基因高度同源序列, 因此推断该基因在每条染色体上各含有 1个拷贝。QPCR方法检测结果显示:该基因在10个个体中发生了拷贝数的增加, 在14个个体中发生了拷贝数的缺失(图 2), 说明在不同莱芜猪个体中, RCAN1基因存在拷贝数变异, 与SNP芯片检测结果一致, 从而进一步验证了CNVR内拷贝数变异的真实存在。
2.4 候选基因 RCAN1拷贝数变异的遗传规律分析结果
分析样本来自莱芜猪A、B、C 3个家系15个个体。利用QPCR方法所检测的3个家系亲子两代的 RCAN1基因的拷贝数及其拷贝数变异的遗传规律分析结果如图3所示。
从图3可看出:①A家系中父本含有1个拷贝,基因型为0/1, 母本含有3个拷贝, 基因型为 1/2, 3个子代个体分别有 1、3、2个拷贝, 基因型分别为0/1、1/2、1/1或 0/2; ②B、C家系为同一父本, 父本均有2个拷贝, 基因型均为0/2, 母本均有1个拷贝, 基因型均为0/1, 所有子代个体包含1、2、3 共3种拷贝数和0/1、0/2、1/2共3种基因型。③在同一个体的一条染色体上 RCAN1基因的拷贝数不同,有缺失(0个拷贝)也有重复(2个拷贝), 但是莱芜猪个体中不存在缺失纯合子(0/0)。
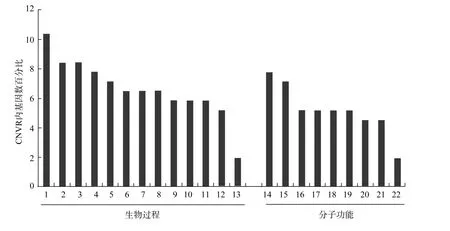
图1 GO方法预测猪13号染色体上CNVR内基因功能的结果1:蛋白质水解; 2:细胞粘附; 3:生物粘附; 4:大分子降解; :5:离子转运; 6:细胞外蛋白质代谢; 7:细胞内蛋白质代谢; 8:蛋白质分解; 9:阳离子运输; 10:修饰依赖的大分子代谢; 11:修饰依赖的蛋白质代谢; 12:细胞间粘附; 13:不依赖钙的细胞间粘附; 14:肽酶活性; 15:作用于亮氨酸的肽酶活性; 16:细胞内肽酶活性; 17:离子通道活性; 18:底物特异性通道活性; 19:通道活性; 20:碱金属离子结合; 21:门控通道活性; 22:清道夫受体活性。
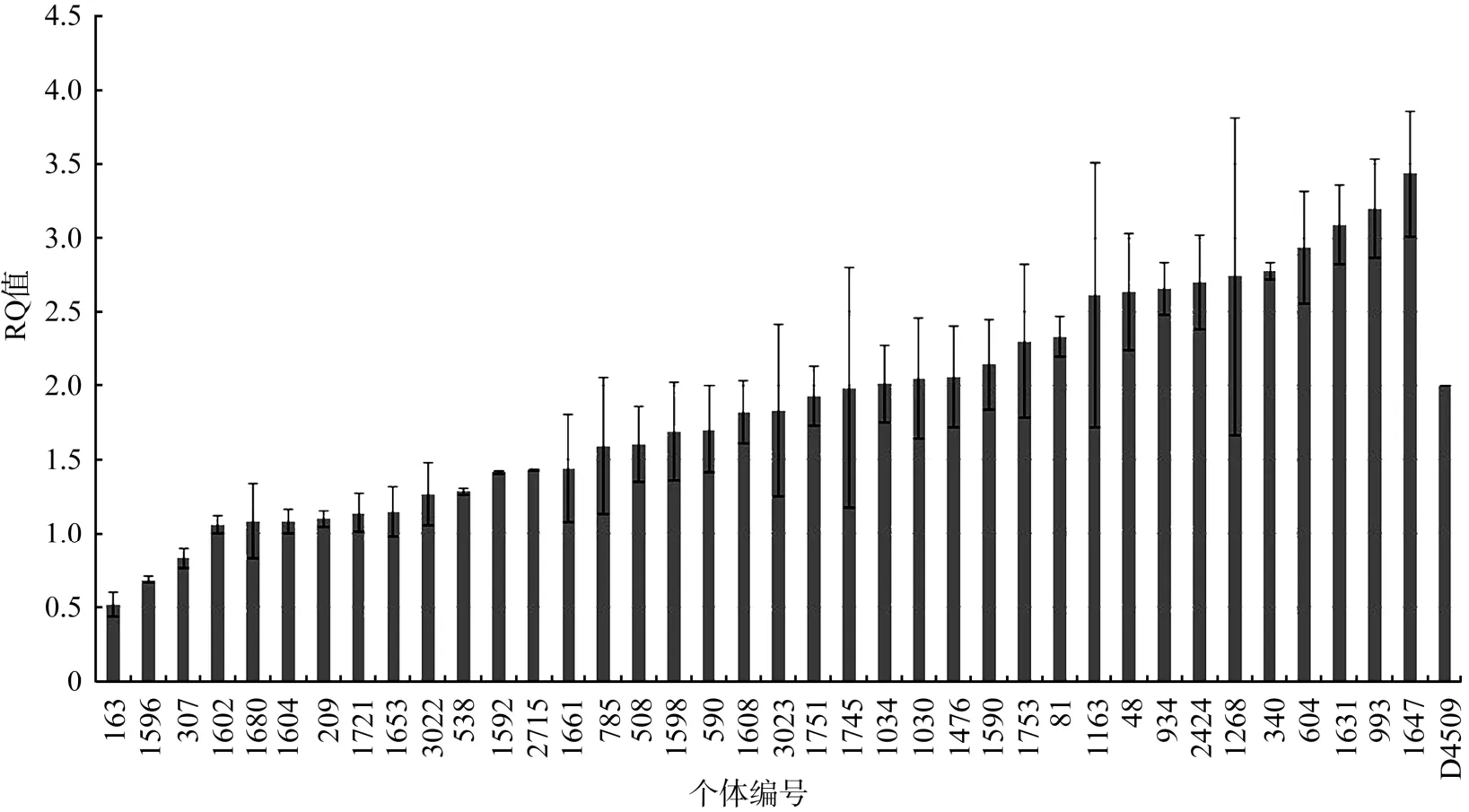
图2 QPCR方法检测莱芜猪13号染色体上RCAN1基因拷贝数的结果相对于参考个体D4509, 有10个个体(604、993、1163、48、1268、2424、934、340、1647和1631)发生了拷贝数的增加, 有14个个体(3022、1721、307、1661、1653、1602、1680、1604、1596、209、163、1592、2715和538)发生了拷贝数的缺失。
3 讨 论
3.1 猪13号染色体具有丰富的CNV及其在标记选择中的可能作用
根据Ensembl(http://asia.ensembl.org/index.html)提供的信息, 猪13号染色体全长约218 635 kb, 所有CNVR长度45 721 kb, 约占13号染色体的20.9%。Li等[7]发现在猪的不同染色体中, CNVR所占的比例介于 0.14%~1.87%之间; 在绵羊的不同染色体上, CNV可以覆盖0.46%~5.08%[8]; 牛的不同染色体中, CNVR约占 0.08%~3.5%[9]。然而本研究结果发现, SSC13上CNVR的比例远远高于在猪、绵羊、牛中已有的报道。另外, 本研究中猪 13号染色体上CNVR重复区域数(16个)要多于缺失区域数(10个),这与已有的报道中各染色体上缺失区域数多于重复区域数[10]的结果不一致。这说明本研究中SSC13上具有丰富的拷贝数变异。
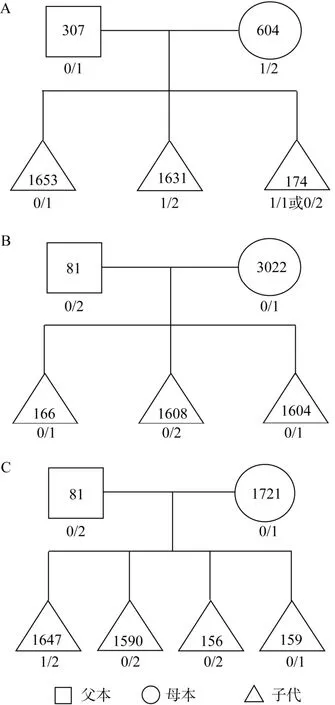
图3 RCAN1基因拷贝数变异的遗传规律分析结果
Ensembl数据库中, 猪13号染色体全长含编码基因1298个, 计算得出基因密度为5.95/Mb。Conrad等[11]和 Freeman等[12]在人类基因组的研究结果表明:CNV更多的趋向于发生在基因匮乏的区域。而本研究发现SSC13上CNVR内的基因密度与该染色体上的基因密度差异不显著, 这说明猪的13号染色体上CNV的分布比较均匀, 并不趋向于发生在基因匮乏或者基因富集的区域。
根据比较基因组学方法所得出的结果推断, SSC13上CNVR中的180个基因其生物学功能可能涉及参与蛋白质水解、细胞粘附过程, 可能影响机体能量代谢和免疫等生物学活动。课题组前期工作中发现莱芜猪尤其特殊, 在13号染色体上存在大范围的染色体缺失或重复区域, 而莱芜猪具有肌内脂肪含量高的种质特性[13], 是目前所报道的肌内脂肪含量最高的品种。CNV的存在是否与莱芜猪的高肌内脂肪含量有关, CNVR内的基因是否可作为莱芜猪的候选基因值得进一步研究。
3.2 关于QPCR验证结果及遗传规律分析
根据RCAN1基因在3个莱芜猪家系上下代中的拷贝数, 假设该基因存在 2个及以上拷贝时, 呈现串联重复且完全连锁, A家系中, 父本的基因型为0/1, 母本的基因型可能存在0/3或1/2两种情况, 如果母本为0/3, 后代会有0/0、0/1、0/3、1/3等4种基因型和0、1、3、4共4种拷贝数; 如果为1/2, 后代会有0/1、0/2、1/1、1/2这4种基因型和1、2、3等3种拷贝数, 由于子代中出现了RCAN1基因拷贝数为2的个体, 所以母本的基因型只能是1/2。B、C家系中, 母本基因型均为 0/1, 父本基因型可能存在1/1或0/2两种情况, 如果为1/1, 后代会有0/1、1/1两种基因型和1、2两种拷贝数; 如果为0/2, 后代会有0/0、0/1、0/2、1/2这4种基因型和0、1、2、3共4种拷贝数, 由于C家系子代中出现了RCAN1基因拷贝数为 3的个体, 那么父本的基因型只能为0/2。由此推断该基因的拷贝数变异在莱芜猪群体中符合孟德尔分离定律, 为CNV的遗传规律研究提供了素材和研究基础。
B、C家系中, 父本基因型为0/2, 母本基因型为0/1, 则可以产生0/0、0/1、0/2、1/2共4种基因型的后代, 但是本研究并没有发现基因型为 0/0的子代个体, 可能是因为本研究的家系含量小, 不能使所有的基因型在后代都能出现。另一方面, RCAN1基因可以调节钙调神经磷酸酶(Calcineurin, CN)的活性, Park等[14]的研究表明RCAN1基因的表达与唐氏综合征具有密切关系, Ermak等[15]也发现RCAN1基因在人脑中的高表达可以引起阿尔茨海默氏病(Alzheimer’s disease, AD), 说明RCAN1基因在一些疾病中发挥了重要作用。本研究在莱芜猪3个家系中未发现缺失纯合体, 这可能是因为RCAN1基因具有重要功能, 缺失纯合体生活力弱或者胚胎期已死亡。这种推断尚有待进一步在群体中进行验证或在细胞水平上进行研究。
[1] Sebat J, Lakshmi B, Troge J, Alexander J, Young J, Lundin P, Månér S, Massa H, Walker M, Chi MY, Navin N, Lucito R, Healy J, Hicks J, Ye K, Reiner A, Gilliam TC, Trask B, Patterson N, Zetterberg A, Wigler M. Large-scale copy number polymorphism in the human genome. Science, 2004, 305(5683): 525-528
[2] Lupski JR, Stankiewicz P. Genomic disorders: molecular mechanisms for rearrangements and conveyed phenotypes. PLoS Genet, 2005, 1(6): e49.
[3] McCarroll SA, Hadnott TN, Perry GH, Sabeti PC, Zody MC, Barrett JC, Dallaire S, Gabriel SB, Lee C, Daly MJ, Altshuler DM. Common deletion polymorphisms in the human genome. Nat Genet, 2006, 38(1): 86-92.
[4] Kleinjan DA, van Heyningen V. Long-range control of gene expression: emerging mechanisms and disruption in disease. Am J Hum Genet, 2005, 76(1): 8-32.
[5] 余少波. 猪全基因组 CNV发掘与 CNV图谱构建[学位论文]. 华中农业大学, 2011.
[6] Ballester M, Castelló A, Ibáñez E, Sánchez A, Folch JM. Real-time quantitative PCR-based system for determining transgene copy number in transgenic animals. Biotechniques, 2004, 37(4): 610-613.
[7] Li Y, Mei SQ, Zhang XY, Peng XW, Liu G, Tao H, Wu HY, Jiang SW, Xiong YZ, Li FG. Identification of genome-wide copy number variations among diverse pig breeds by array CGH. BMC Genomics, 2012, 13(1): 725.
[8] Liu JS, Zhang L, Xu LY, Ren HX, Lu J, Zhang XN, Zhang SF, Zhou XL, Wei CH, Zhao FP, Du LX. Analysis of copy number variations in the sheep genome using 50K SNP BeadChip array. BMC Genomics, 2013, 14(1): 229.
[9] Liu GE, Hou YL, Zhu B, Cardone MF, Jiang L, Cellamare A, Mitra A, Alexander LJ, Coutinho LL, Dell'Aquila ME, Gasbarre LC, Lacalandra G, Li RW, Matukumalli LK, Nonneman D, de A Regitano LC, Smith TPL, Song JZ, Sonstegard TS, Van Tassell CP, Ventura M, Eichler EE, McDaneld TG, Keele JW. Analysis of copy number variations among diverse cattle breeds. Genome Res, 2010, 20(5): 693-703.
[10] Wang JY, Jiang JC, Fu WX, Jiang L, Ding XD, Liu JF, Zhang Q. A genome-wide detection of copy number variations using SNP genotyping arrays in swine. BMC Genomics, 2012, 13(1): 273.
[11] Conrad DF, Andrews TD, Carter NP, Hurles ME, Pritchard JK. A high-resolution survey of deletion polymorphism in the human genome. Nat Genet, 2006, 38(1): 75-81.
[12] Freeman JL, Perry GH, Feuk L, Redon R, McCarroll SA, Altshuler DM, Aburatani H, Jones KW, Tyler-Smith C, Hurles ME, Carter NP, Scherer SW, Lee C. Copy number variation: New insights in genome diversity. Genome Res, 2006, 16(8): 949-961.
[13] 曾勇庆, 王根林, 魏述东, 王林云, 杨海玲, 曹洪防, 徐云华. 含不同比例莱芜猪血缘杂交猪胴体品质及肉质特性的研究. 遗传, 2005, 27(1): 65-69.
[14] Park JK, Oh YH, Chung KC. Two key genes closely implicated with the neuropathological characteristics in Down syndrome: DYRK1A and RCAN1. BMB Rep, 2009, 42(1): 6-15.
[15] Ermak G, Sojitra S, Yin F, Cadenas E, Cuervo AM, Davies KJ. Chronic expression of RCAN1-1L protein induces mitochondrial autophagy and metabolic shift from oxidative phosphorylation to glycolysis in neuronal cells. J Biol Chem, 2012, 287(17): 14088-14098.
(责任编委: 李绍武)
Investigation of genes within copy number variation regions in pig chromosome 13 and analysis of the genetic law
Jing Liu, Yanan Wang, Yaqi Sun, Hongyang Wang, Chao Wang, Zhongzhen Peng, Bang Liu
Key Laboratory of Agricultural Animal Genetics, Breeding and Reproduction of Ministry of Education, College of Animal Science and Technology, Huazhong Agricultural University, Wuhan 430070, China
Copy number variation (CNV), referring to a genome structure variation, has attracted researchers’ great interests. Thirty-two CNV regions (CNV region, CNVR) have been detected on chromosome 13 in our previous work. In order to detect the genes located in these CNVRs, we first obtained the annotated information from Ensembl database, and searched gene functional enrichments using DAVID online tools. In the 32 CNVRs, a total of 236 genes were identified, in which 169 genes were annotated. Gene Ontology (GO) analysis revealed that these genes mainly participate in proteolysis, cell adhesion, and macromolecular catabolic process. To study the genetic law of these CNVs, we chose the RCAN1 (regulators of calcineurin 1) gene as the candidate. We quantified the copy number of RCAN1 gene in 38 Laiwu pigs by usingQPCR method, and analyzed the genetic laws in three Laiwu families including 15 pigs. QPCR results showed that both duplication and deletion occurred in RCAN1 gene among Laiwu pigs and the heredity mode corresponds with Mendelian genetic law.
copy number variation; chromosome 13; RCAN1 gene; genetic law; Laiwu pigs
2013-10-14;
2013-12-30
国家自然科学基金项目(编号:31072012)和全国大学生创新创业训练计划创新训练项目(编号:201210504001)资助
刘静, 硕士研究生, 专业方向:动物功能基因组学。E-mail: liujing7791621@163.com
刘榜, 博士, 教授, 研究方向:动物分子生物学与育种。E-mail: liubang@mail.hzau.edu.cn
10.3724/SP.J.1005.2014.0354
时间: 2014-2-13 14:24:20
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140213.1424.001.html

