胶红酵母JB401降解脱色三苯甲烷类染料
李国辉,高剑平,丁重阳,刘元法,张 梁,顾正华,石贵阳,章克昌
(1.江南大学 生物工程学院 工业生物技术教育部重点实验室,无锡 214122;2.江南大学 粮食发酵工艺与技术国家工程实验室,无锡 214122;3.漳州职业技术学院,漳州 363000;4.江南大学 食品学院,无锡 214122)
三苯甲烷类染料广泛地应用于纺织印染、造纸和制革等工业中[1-3],近期研究发现结晶紫(图1)等三苯甲烷类染料对动物细胞具有毒害作用,因此有必要研究该类染料的脱色降解处理。一些物理和化学方法虽然也可以用于染料的处理,但由于其代价高、污泥产生量多和二次污染等原因,限制了其在工业中的应用[4-6]。利用微生物处理染料具有无污染、耗能低、代价小和可持续性好等优点[7],因此筛选出对三苯甲烷类染料具有降解脱色能力的微生物,研究微生物降解染料的机制,对三苯甲烷类染料污染治理具有重要意义。
诸多微生物都具有降解脱色三苯甲烷类染料的能力,如细菌 Agrobacterium radiobacter MTCC 8161[8]和 Bacillus cereus DC11[9]完全脱色 10 mg/L的结晶紫分别需要8和24 h,丝状真菌 Trametes sp.SQ01 6 d内可脱色20 mg/L结晶紫30%以上[10],酵母 Saccharomyces cerevisiae MTCC 463 在7 h内可脱色100 mg/L孔雀石绿85%以上[7]。在细菌、酵母和丝状真菌中,酵母脱色三苯甲烷染料的能力较强,但是到目前为止,利用酵母脱色三苯甲烷类染料的研究较少,因此考虑筛选酵母来脱色三苯甲烷类染料。
笔者在烟梗样品中分离筛选获得1株高效脱色结晶紫的菌株,经ITS-5.8S rDNA鉴定后发现该菌株为胶红酵母。在初步确定脱色机理后,考察不同培养条件对胶红酵母降解脱色结晶紫的影响,以期为其在生物降解化工废水方面的应用提供基础数据。

图1 3种三苯甲烷染料的结构式Fig.1 Chemical structures of three triphenylmethane dyes
1 材料与方法
1.1 材料
1.1.1 菌种
胶红酵母(Rhodotorula mucilaginosa)JB401,筛选自四川地区产烟梗中。
1.1.2 培养基
种子培养基(g/L):NaCl 5,酵母膏5,蛋白胨10;pH 7。
脱色培养基(g/L):NaCl 5,酵母膏5,蛋白胨10,结晶紫0.05;pH 7。
以上培养基均在115℃下灭菌20 min,斜面在30℃下培养48 h后放入4℃冰箱保存,使用时用接种环取1环菌放入种子培养基中,30℃培养24 h后待用。
1.2 方法
1.2.1 脱色微生物的分离筛选与鉴定
称取2 g烟梗样品,加入98 mL无菌生理盐水,放入250 mL无菌三角瓶中,在30℃、150 r/min的摇床上培养1 h后,将培养液分别稀释104、105或106倍后涂布在水溶苯胺蓝平板上,并通过连续的分离纯化筛选出能够产生稳定、明显、较大透明圈的菌落(透明圈直径与菌落直径比值越大越好),斜面保藏。
将斜面保藏的菌种转接到种子培养基中,30℃、150 r/min培养,24 h后分别加入不同浓度的结晶紫,通过测定不同菌株对结晶紫的脱色率,从中筛选出脱色能力优良的菌株,保藏后进行后续试验。
按照Guillamm等[11]的方法对筛选得到的菌株进行ITS-5.8S rDNA序列扩增,所用引物ITS1:5'-TCCGTAGGTGAACCTGCGG-3',ITS4:5'-TCCTCCGCTTATTGATATGC-3'。将 PCR产物连接到载体pMD18T,转化感受态JM109,提取质粒,扩增得到的ITS-5.8S rRNA基因利用ABI 3130 DNA自动序列分析仪在上海生工生物工程公司进行测序,并在NCBI数据库中搜索进行同源性分析。最后利用ClustalX和Mega 5软件制作进化树。
1.2.2 全波长扫描
统计并记录所有患者诊断结果,比较CT扫描,增强MRI的诊断效能,计算CT扫描和增强MRI以及两者联合应用诊断的、敏感度、特异度和准确性。
收集胶红酵母脱色完全后的结晶紫培养液,8 000 r/min离心20 min去除沉淀,上清液中加入等体积乙酸乙酯萃取,用无水Na2SO4干燥后,在旋转蒸发仪上蒸干,获得的结晶用少量色谱级甲醇溶解,使其终质量浓度为0.1 mg/mL,对照为不加入胶红酵母的结晶紫培养基[12],对脱色前后的染料在200~900 nm波长范围进行扫描,获取的数据用UV-solutions1.2(日立集团)处理。
1.2.3 脱色实验
将生长24 h的种子按2%比例接种于脱色培养基中,30℃、150 r/min培养24 h后,加入50 mg/L结晶紫,每隔1 h取出一定量的样品,与无水乙醇按体积比1∶2的比例(V(样品)∶V(无水乙醇))混匀后,12 000 r/min离心5 min,利用分光光度法在染料的最大吸收峰处测定上清液中剩余染料的含量,并以不接种的染料培养基作为对照来测定染料的脱色率[13-14]。每组3个平行实验,脱色率按式(1)来计算。
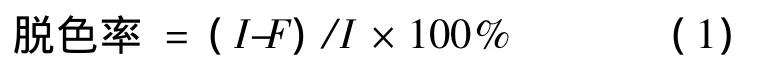
式中:I为对照在最大吸收峰处的吸光值;F为某一时刻样品在最大吸收峰处的吸光值。
在此条件下,依次分别对pH 4~11、温度(20~45)℃、接种量0.1% ~8%、培养方式(摇床和静置)、初始染料质量浓度(25~200)mg/L进行单因素实验,确定胶红酵母对结晶紫脱色的最优条件。
2 结果与讨论
2.1 脱色微生物的分离筛选与鉴定
从烟梗样品中一共筛选到20株能够在苯胺蓝平板上产生透明圈的微生物,复筛后发现其中4株可以明显降解结晶紫,4株中菌株JB401脱色效果最好,因此以JB401为出发菌株进行后续试验。
经过ITS-5.8S rDNA序列测定及GenBank数据库比对,结果见图2。由图2可知:菌株 JB401与Rhodotorula mucilaginosa strain ATCC 66034(EU853846.1)相似性达到100%,由红酵母属ITS-5.8S rDNA序列构建的N-J型进进化树中(图2),菌株JB401与R.mucilaginosa strain ATCC 66034(EU853846.1)和 R.mucilaginosaCBS9078(AF444655.1)亲缘关系最近,因此命名为Rhodotorula mucilaginosa JB401。

图2 JB401系统进化树Fig.2 The phylogenetic tree of JB401
2.2 全波长扫描分析
利用胶红酵母对结晶紫脱色后,将培养液进行全波长扫描,结果见图3。由图3可知:在589 nm处的最大吸收峰 λmax消失。而 Lee等[15]利用Pseudomonas otitidis WL-13降解结晶紫时发现,离心后上清液为无色,而菌体沉淀为紫色,认为该菌株对结晶紫的脱色主要是菌体的吸附作用。在本实验中,脱色后离心得到的菌体沉淀不显紫色,同时结晶紫脱色后在紫外区形成诸多小峰,表明有新的小分子产物生成,因此说明胶红酵母对结晶紫的脱色并不是由于胶红酵母的菌体吸附作用,而是由菌体的降解作用引起的。类似结果在Staphylococcus epidermidis降解结晶紫、孔雀石绿等三苯甲烷类染料过 程 中 也 有 报 道[16-17]。 此 外,Agrobacterium radiobacter MTCC 8161可利用漆酶的脱甲基和去苯环能力作用于结晶紫[8],使之分解为多种小分子物质,并在全波长扫描中产生新峰[18],这一现象也与R.mucilaginosa JB401降解结晶紫相似。

图3 全波长扫描结晶紫降解前后变化Fig.3 UV-VIS spectral analysis before and after crystal violet degradation
2.3 初始pH对胶红酵母脱色的影响
图4为初始pH对胶红酵母脱色的影响结果。由图4可知:胶红酵母对结晶紫的脱色效果在酸性范围较好,在pH 4~7之间脱色率可以保持在86%以上,其中pH 5时脱色率最高,分别为94.08%。当pH>7时,随pH的增加脱色率急剧降低,当pH为10时脱色率仅为21.97%。
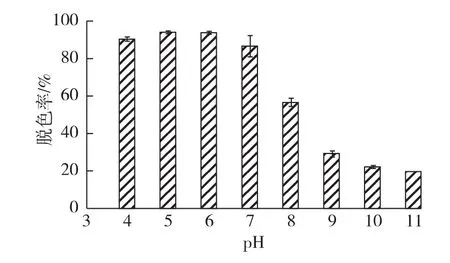
图4 pH对结晶紫脱色的影响Fig.4 Effects of pH on decolorization of crystal violet
这与前人的研究结果不同,可能是因为不同的微生物对不同的染料脱色的最适pH各有不同。如,黄孢原毛平革菌对甲基紫的最适脱色pH为4.5,pH增加或降低均对黄孢原毛平革菌的脱色产生明显影响,当pH从3增加到7时,黄孢原毛平革菌对甲基紫的脱色率降低了 75%[19],Trametes sp.SQ01产生漆酶脱色Remazol Brilliant Blue R时,也得到了相似的结果[20]。由此可见,胶红酵母在偏酸性pH范围内有较高的脱色能力,这一特点符合生物处理废水的要求。
2.4 温度对胶红酵母脱色的影响
考察不同的温度对结晶紫脱色影响,结果见图5。由图5可知:在20和45℃时,胶红酵母对结晶紫脱色率仍高于80%,分别为81.17%和85.50%;其中,在37℃时,脱色效率最高,达到93.10%。当温度低于37℃时,脱色率随温度升高逐渐升高,超过45℃后,随温度升高脱色率则下降。
Phanerochaete chrysosporium对结晶紫的最适脱色温度是(35±1)℃,当温度升高或降低时,会影响到染料的脱色率,这可能是由于生成的过氧化物酶减少或者酶失活造成脱色能力的降低[19]。对于纯化后的真菌漆酶也是如此,在一定范围内,当温度升高时,由于酶与底物间反应速率加快导致单位时间内脱色率升高,但是当温度超过一定值后,由于酶的失活引起脱色率降低[20]。
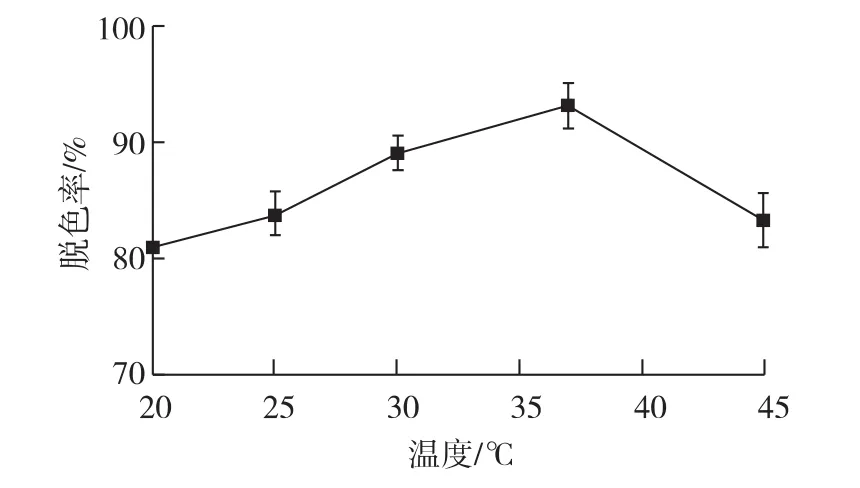
图5 温度对结晶紫脱色的影响Fig.5 Effects of temperature on decolorization of crystal violet
2.5 接种量对胶红酵母脱色的影响
图6为接种量对胶红酵母脱色的影响结果。由图6可知:当接种量小于2%时,脱色效率均随接种量的升高而升高;当接种量为0.1%时,脱色率仅为23.44%;当接种量为2%时,脱色率为90.13%;当接种量大于等于2%时,结晶紫的脱色率保持稳定,并保持在90%以上。
而在Candida krusei脱色过程中,最适接种量为5%;超过5%时并不会影响达到最大脱色率的时间;但小于5%时,达到最大脱色率的时间会有所延迟,如接种量为3%时,则延长了24 h[21]。这可能是由于当接种量较少时,菌体量较少不足以产生足够的酶来处理染料,且染料对菌体具有一定的毒性而抑制菌体生长,所以造成了脱色时间的延长。这一结果在利用Saccharomyces cerevisiae降解孔雀石绿的研究中也得到了证实[7]。
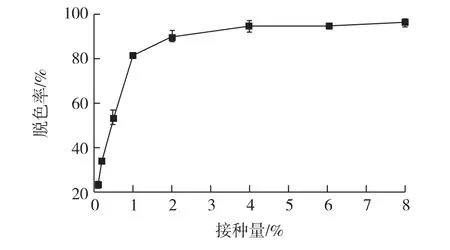
图6 接种量对结晶紫脱色的影响Fig.6 Effects of inoculum ratio on decolorization of crystal violet
2.6 培养方式对胶红酵母脱色的影响
考察不同培养方式对胶红酵母脱色的影响,结果见图7。由图7可知:摇床培养时脱色效率较高。在静置条件下,胶红酵母培养4 h时脱色率达到91.85%;而摇床培养2 h后,脱色率即达90.00%。在脱色的前5 h,摇床脱色一直明显优于静置脱色,但是5 h后二者几乎无差异(图7)。
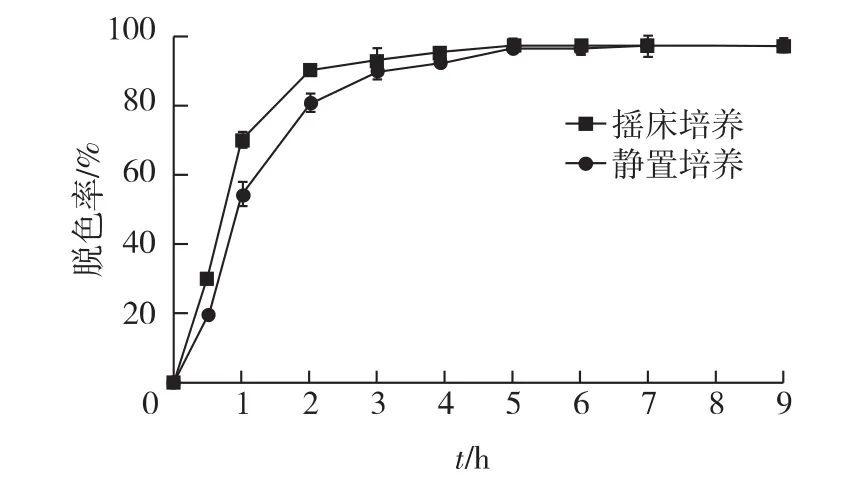
图7 培养方式对结晶紫脱色的影响Fig.7 Effects of culture condition on decolorization of crystal violet
Conway等[22]研究发现,几乎未见利用酵母和丝状真菌在厌氧条件下对染料进行脱色,这可能是因为在厌氧条件下,菌体的生长和代谢都受到较大影响,进而影响到染料的脱色和降解。然而,胶红酵母在对结晶紫染料脱色时,对氧的要求不大,静置条件下达到最大脱色率的时间仅比摇床条件下的延长了2 h,几乎未受到影响,这一特性使其可适用于工业染料废水处理中的各种复杂体系,并且不会影响染料废水的脱色处理。
2.7 初始染料浓度对胶红酵母脱色的影响
考察不同初始染料浓度对胶红酵母脱色的影响,结果见图8。由图8可知:在14 h内胶红酵母对结晶紫的脱色率均达到90%以上。其中,对于25 mg/L的结晶紫,在2 h内的脱色率就达94.46%,这和Candida krusei相似;对于200 mg/L的染料,二者都是在20 h左右达到最大脱色率[21]。由于染料对菌体具有一定的毒性,因此微生物对200 mg/L结晶紫的去除率通常低于40%[10,23]。胶红酵母对不同浓度结晶紫的适应性较强,在不同浓度条件下,结晶紫的脱色率都超过90%,而且耗时较短。工业染料废水中染料浓度较高,因此胶红酵母所具有的可降解不同浓度结晶紫的能力,使其适用于工业化处理染料废水。
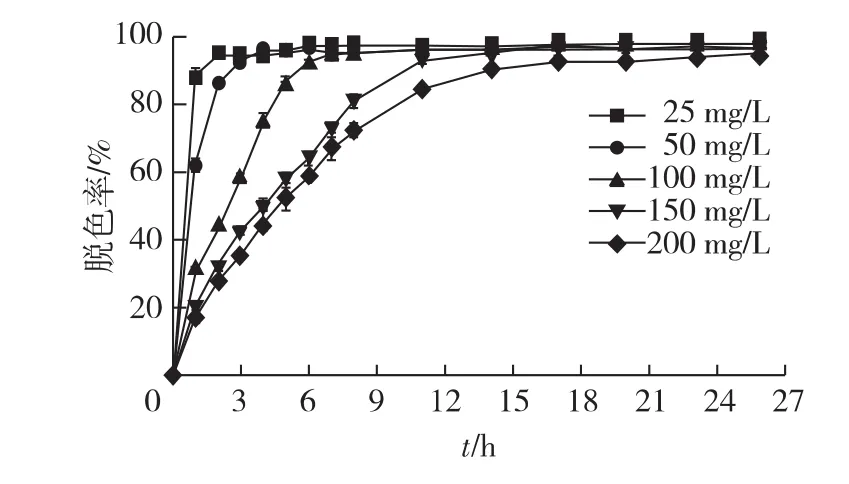
图8 初始染料浓度对结晶紫脱色的影响Fig.8 Effects of initial dye concentration on decolorization of crystal violet
3 结论
通过分离筛选获得1株具有降解结晶紫能力的胶红酵母,通过全波长扫描证实其降解能力,后续试验发现胶红酵母对温度和pH均有较好的适应性,对不同浓度结晶紫都可达到较高的脱色率。
在本文的研究基础之上,下一步将围绕胶红酵母降解结晶紫的详细过程进行研究:①降解过程中涉及的脱色酶及其作用方式;②染料降解过程中产生的小分子物质,以及可能的降解代谢途径;③染料降解前后的毒性变化,以期在生物降解化工废水方面提供更好的支持。
[1] Govindwar S,Parshetti G,Kalme S,et al.Biodegradation of malachite green by Kocuria rosea MTCC 1532[J].Acta Chim Slov,2006,53(4):492-498.
[2] Nohynek G J,Antignac E,Re T,et al.Safety assessment of personal care products/cosmeticsand theiringredients[J].Toxicol Appl Pharm,2010,243(2):239-259.
[3] Kumar C G,Mongolla P,Basha A,et al.Decolorization and biotransformation of triphenylmethane dye,methyl violet,by Aspergillus sp isolated from Ladakh,India[J].J Microbiol Biotechn,2011,21(3):267-273.
[4] Khataee A R,Pons M N,Zahraa O.Photocatalytic degradation of three azo dyes using immobilized TiO2nanoparticles on glass plates activated by UV light irradiation:influence of dye molecular structure[J].J Hazard Mater,2009,168(1):451-457.
[5] Mittal A,Kurup L,Gupta V K.Use of waste materials:bottom ash and de-oiled soya,as potential adsorbents for the removal of amaranth from aqueous solutions[J].J Hazard Mater,2005,117(2/3):171-178.
[6] Robinson T,Mcmullan G,Marchant R,et al.Remediation of dyes in textile effluent:a criticalreview on currenttreatment technologies with a proposed alternative[J].Bioresour Technol,2001,77(3):247-255.
[7] Govindwar S P,Jadhav J P.Biotransformation of malachite green by Saccharomyces cerevisiae MTCC 463[J].Yeast,2006,23(4):315-323.
[8] Parshetti G K,Parshetti S G,Telke A A,et al.Biodegradation of crystal violet by Agrobacterium radiobacter[J].J Environ Sci(China),2011,23(8):1384-1393.
[9] Sun G P,Deng D Y,Guo J,etal.Decolorization of anthraquinone,triphenylmethane and azo dyes by a new isolated Bacillus cereus strain DC11[J].Int Biodeter Biodegr,2008,62(3):263-269.
[10] Yang X Q,Wang J R,Zhao X X,et al.Increasing manganese peroxidase production and biodecolorization of triphenylmethane dyes by novel fungal consortium[J].Bioresour Technol,2011,102(22):10535-10541.
[11] Guillamon J M,Sabate J,Barrio E,et al.Rapid identification of wine yeast species based on RFLP analysis of the ribosomal internal transcribed spacer(ITS)region[J].Arch Microbiol,1998,169(5):387-392.
[12] Govindwar S P,Kalyani D C,Patil P S,et al.Biodegradation of reactive textile dye red BLI by an isolated bacterium Pseudomonas sp SUK1[J].Bioresour Technol,2008,99(11):4635-4641.
[13] Jadhav U U,Dawkar V V,Ghodake G S,et al.Biodegradation of direct red 5B,a textile dye by newly isolated Comamonas sp UVS[J].J Hazard Mater,2008,158(2/3):507-516.
[14] Yang Y Y,Hu H,Wang G,et al.Removal of malachite green from aqueous solution by immobilized Pseudomonas sp DY1 with Aspergillus oryzae[J].IntBiodeterBiodegr,2011,65(3):429-434.
[15] Lee Y C,Wu J,Jung B G,et al.Isolation and characterization of Pseudomonas otitidis WL-13 and its capacity todecolorize triphenylmethane dyes[J].J Environ Sci(China),2009,21(7):960-964.
[16] Asad S,Amoozegar M A,Pourbabaee A A,et al.Decolorization of textile azo dyes by newly isolated halophilic and halotolerant bacteria[J].Bioresour Technol,2007,98(11):2082-2088.
[17] Ayed L,Chaieb K,CherefA,etal.Biodegradation and decolorization of triphenylmethane dyes by Staphylococcus epidermidis[J].Desalination,2010,260(1/2/3):137-146.
[18] Chung Y C,Chen C C,Liao H J,et al.Biodegradation of crystal violet by Pseudomonas putida[J].Biotechnol Lett,2007,29(3):391-396.
[19] Radha K V,Regupathi I,Arunagiri A,et al.Decolorization studies of synthetic dyes using Phanerochaete chrysosporium and their kinetics[J].Process Biochem,2005,40(10):3337-3345.
[20] Yang X Q,Zhao X X,Liu C Y,et al.Decolorization of azo,triphenylmethane and anthraquinone dyes by a newly isolated Trametes sp SQ01 and its laccase[J].Process Biochem,2009,44(10):1185-1189.
[21] 余志晟,文湘华.酵母Candida krusei对合成染料的脱色[J].环境科学,2005,26(5):137-142.
[22] Conway E J,Kernan R P.The effect of redox dyes on the active transport of hydrogen,potassium and sodium ions across the yeast cell membrane[J].Biochem J,1955,61(1):32-36.
[23] Kang K H,Kim J D,Yoon J H,et al.Identification of Stenotrophomonas maltophilia LK-24 and its degradability of crystal violet[J].J Microbiol Biotechn,2002,12(3):437-443.

