花生栽野杂交后代抗病基因类似物的克隆与分析
李 忠,江立庚,唐荣华,韩柱强,钟瑞春,贺粱琼,蒋 菁,熊发前,唐秀梅
(1 广西大学 农学院,广西 南宁530007;2 广西农业科学院经济作物研究所,广西 南宁530004;3 广西作物遗传改良生物技术重点开放实验室,广西 南宁530007)
花生栽培种是异源四倍体作物,遗传基础狭窄,在形态、抗性、品质等性状上有一定变异,但变异程度较其他作物低,而且许多危害花生的病虫害缺乏抗源[1-2]。花生野生种具有丰富的遗传变异,也存在很多栽培花生所不具备的优良性状。前人的研究表明,花生野生种对一些病虫害的抗性显著高于栽培种,如对花生早斑病、晚斑病、锈病等病害高抗或者免疫[1,3-7]。通过种间杂交或其他生物技术将花生野生种的抗病基因转移到花生栽培种中,是扩大花生栽培种遗传基础、种质创新和新品种选育的重要途径。Arachiscorrentina是与花生栽培种杂交亲和的少数野生花生品种之一,在田间表现出耐贫瘠、抗病性好、耐连作等优良特性。
植物抗病基因的分离是近年来植物分子生物学领域的一个研究热点,所采用的技术手段主要有转座子标签法、图位克隆法和同源序列克隆法等。抗病基因类似物(Resistance gene analog, RGA)分子育种技术是一种利用已知抗病基因所编码的蛋白质保守结构域设计简并引物,并分离和克隆抗病基因及其同源片段的技术,它为作物品种的性状改良及抗病机制的研究提供了新的途径,且已在包括花生在内的许多作物研究方面取得了进展[8-16]。这些研究结果表明,应用RGA克隆技术分离植物抗病基因,并据此进行分子标记辅助抗病育种是一种可行且有效的方法。利用RGA克隆技术分离花生的 RGA,不仅有助于筛选花生各种抗病基因的分子标记,建立花生抗病育种分子标记辅助体系,而且通过对花生 RGA的结构、表达和调控的研究,将有助于深入探明花生的抗病机制。本研究根据已知抗病基因的保守结构域设计引物,在花生野生种A.correntina、栽培种“桂花21”和花生栽野杂交后代中克隆抗病相关基因片段,以期为花生抗病基因的进一步分离、鉴定及利用奠定基础。
1 材料与方法
1.1 材 料
供试花生材料共16个,其中0901~0914为2009年由花生野生种A.correntina(父本)与栽培种“桂花21”(母本)杂交所得的14个子7代材料;花生属花生区组野生种A.correntina,来自国家种质野生花生南宁分圃;抗锈病栽培品种“桂花21”由广西农业科学院经济作物研究所育成和提供。
1.2 方 法
1.2.1 引物的设计与合成 参照文献[12],依据植物R(Resistance)基因NBS区的保守序列设计合成1对引物,引物序列见表1。 引物由上海生物工程有限公司合成。

表1 依据植物R基因NBS区的保守序列设计的引物
1.2.2 gDNA的提取 取花生幼嫩叶片,用液氮于研钵中磨成粉状,用CTAB法[17]提取gDNA。
1.2.3 花生抗病基因类似物基因的克隆和测序 1) PCR 反应条件。以花生gDNA为模板进行PCR扩增。反应体系为:2.5 μL 10×PCR buffer,2.0 μL 25 mmol/L MgCl2,2.5 μL 1.0 mmol/L dNTPs,20 μmol/L 上下游引物各1 μL,1 UTaqDNA聚合酶,约100 ng模板,补足ddH2O至25 μL。PCR反应程序为:94 ℃预变性2 min;94 ℃变性30 s,50 ℃ 1 min,72 ℃ 1 min,40个循环;72 ℃延伸10 min,4 ℃ 终止反应。
2) PCR产物的克隆及测序。PCR产物用10 g/L琼脂糖凝胶进行电泳(80 V,200 A)40 min,用 OMEGA公司GEL EXTRACTION KIT柱式 DNA凝胶回收试剂盒回收差异 DNA片段。按 TIANGEN公司pGM-T载体试剂盒使用说明,将回收的PCR 产物与 pGM-T 载体连接。将连接的质粒载体转化大肠杆菌DH5α感受态细胞,涂布于含IPTG(200 mg/L)、氨苄青霉素(100 mg/L)和 X-Gal (20 mg/L)的 LB 固体培养基上,37 ℃倒置培养12~16 h。挑取5~7个白色单菌落,进行菌落PCR检测,检测插入片段的有无和大小。将阳性克隆菌用牙签挑出,置于用1.5 mL离心管制作的LB固体培养基小平板中,送样至上海Invitrogen公司测序。
1.2.4 序列比对与分析 使用DNAstar、DNAclub软件进行序列初步分析,包括DNA至氨基酸转换、ORF分析。应用PIR网站(http://pir.georgetown.edu/)进行蛋白质保守区结构分析。使用NCBI网站上的BLAST搜索同源序列,并对各组RGA氨基酸序列与已知R氨基酸序列的同源性进行综合性分析,用DNAMAN构建系统发育树。
2 结果与分析
2.1 花生RGA的扩增与鉴定
利用依据R基因NBS区保守序列设计合成的引物PLTR,以花生基因组gDNA为模板进行PCR扩增。对PCR扩增谱带的分析结果表明,引物PLTR的PCR产物出现1条500 bp左右的条带,条带较清晰,有1个花生材料0909没有条带(图1)。目的片段经回收、克隆,获得15个花生抗病基因类似物基因(13个来自栽野杂交后代,2个分别来自父母本),相应序列见图2。图2表明,15条序列均具有完整开放读码框(ORF),这些序列的大小在498~500 bp。将本试验所得基因序列提交NCBI网站,得到4个GenBank收录号,分别为:KF885730.1、KF885729.1、KF885731.1和KF885732.1。对推导出的15个花生RGA完整氨基酸序列蛋白质结构的分析表明,15条序列含有典型的NBS保守区的N端糖基化位点(NNSG)、蛋白激酶C磷酸化位点(TTR、SWK、SCK)、酪蛋白激酶Ⅱ磷酸化位点(TRND、SYDD)、N-豆蔻酰化位点(GLPLAI、GIFPAD)特征序列(图2)。
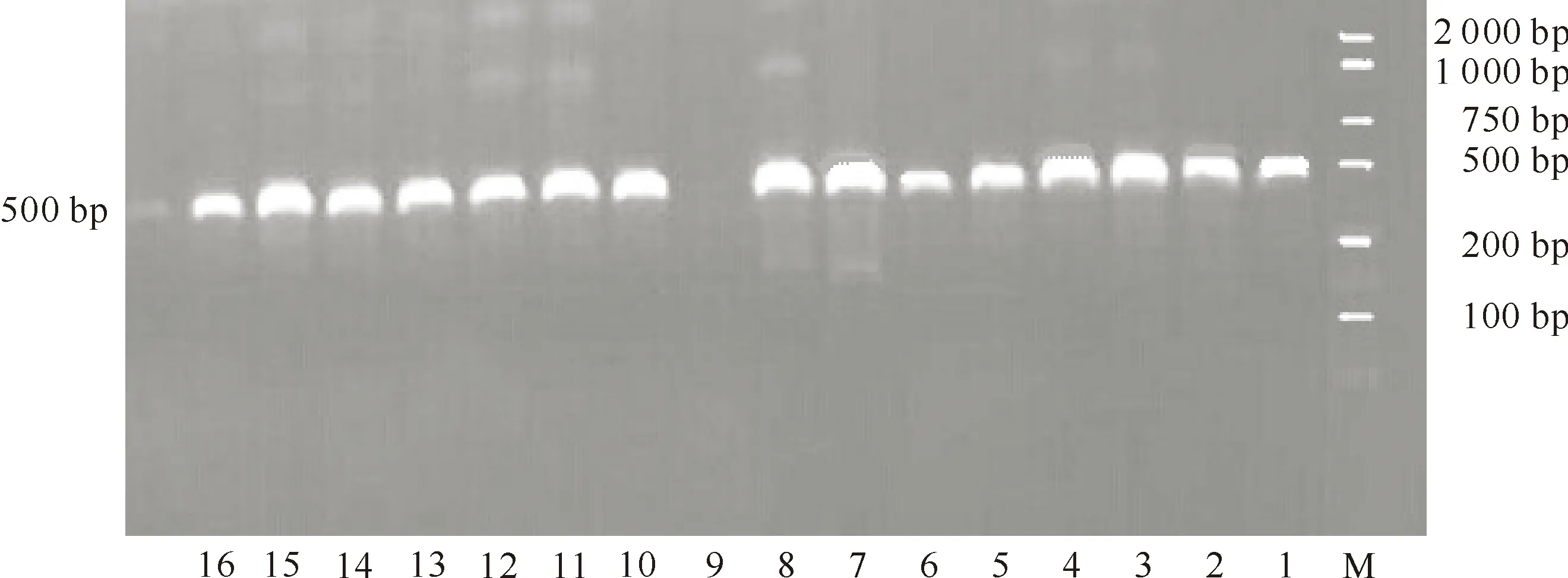
图1 花生RGA的PCR扩增结果
2.2 花生RGA序列的同源性比较
2.2.1 花生RGA氨基酸序列的相似性 利用 DNAMAN 软件,对15条RGA氨基酸序列与GenBank中收录的有代表性的8条来自花生的抗病R氨基酸序列进行分析,结果见图3。由图3可知,15个RGA氨基酸有很高的相似性,序列间的同源性在73.2%~100.0%;2个亲本序列的同源性为73.8%,其中母本“桂花21”与栽野杂交后代材料序列的相似性为73.9%~100.0%,比父本A.correntina(73.2%~98.2%)略高。15条RGA氨基酸序列与已发表的花生抗病基因氨基酸序列也具有较高的相似性,相似性在72.1%~100.0%,依据聚类结果可将这些序列分为2组:在第Ⅰ组中,只有基因0903、A.correntina和0907 聚在一起,序列间的相似性较高,为95.4%~98.2%;其余RGA材料均聚在第Ⅱ组中,序列间的相似性在81.6%~100.0%。第Ⅰ组与第Ⅱ组的相似性为74.0%。
2.2.2 花生与其他作物RGA R序列的相似性 将15个片段的氨基酸序列输入NCBI网站的GenBank数据库,利用BLAST进行同源性搜索,然后与其他作物的R序列氨基酸(表2)进行比对,结果见图4。由图4可以看出,与15条氨基酸序列同源性相对较高的作物主要有:番茄(Solanumlycopersicum,序号:1~3)、葡萄(Vitisvinifera,序号:4~9)、大豆(Glycinemax,序号:10~14)、毛果杨(Populustrichocarpa,序号:15~16)、鹰嘴豆(Cicerarietinum,序号:17~18)、菜豆(Phaseolusvulgaris,序号:19~22)、马铃薯(Solanumtuberosum,序号:23~27)、烟草NBS抗性基因类似蛋白(Nicotianatabacum,序号:28~31)、野生马铃薯(Solanumalbicans,序号:32~34)(表2)。通过氨基酸序列的相应区域BLAST 比对发现,15条氨基酸序列与其他植物之间的同源性均较小,在34.1%~54.6%,其中与大豆序列12的氨基酸序列同源性最高,达51.9%~54.6%;与烟草序列30的氨基酸序列同源性最低,为34.1%~39.1%。聚类结果可分为4大类,本试验所得的15个序列聚在一起,其他作物序号4~22的氨基酸序列聚为一类,这2大类的同源性为45%;序列1~3,23~26及28~34归为一类,与前述2大类的同源性为43%;序列27单独归为一类,与前述3大类的同源性为41%。这些具有一定程度同源的、来自于其他物种的抗病基因可能与花生具有近亲关系。

图2 花生RGA氨基酸序列的比对分析
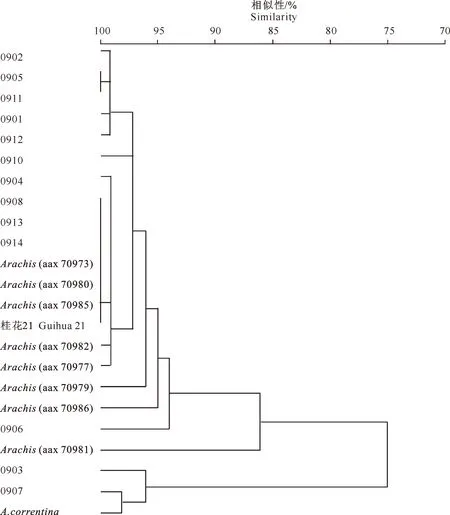
图3 15个花生RGA氨基酸序列与已知的8个花生R氨基酸序列的相似性分析
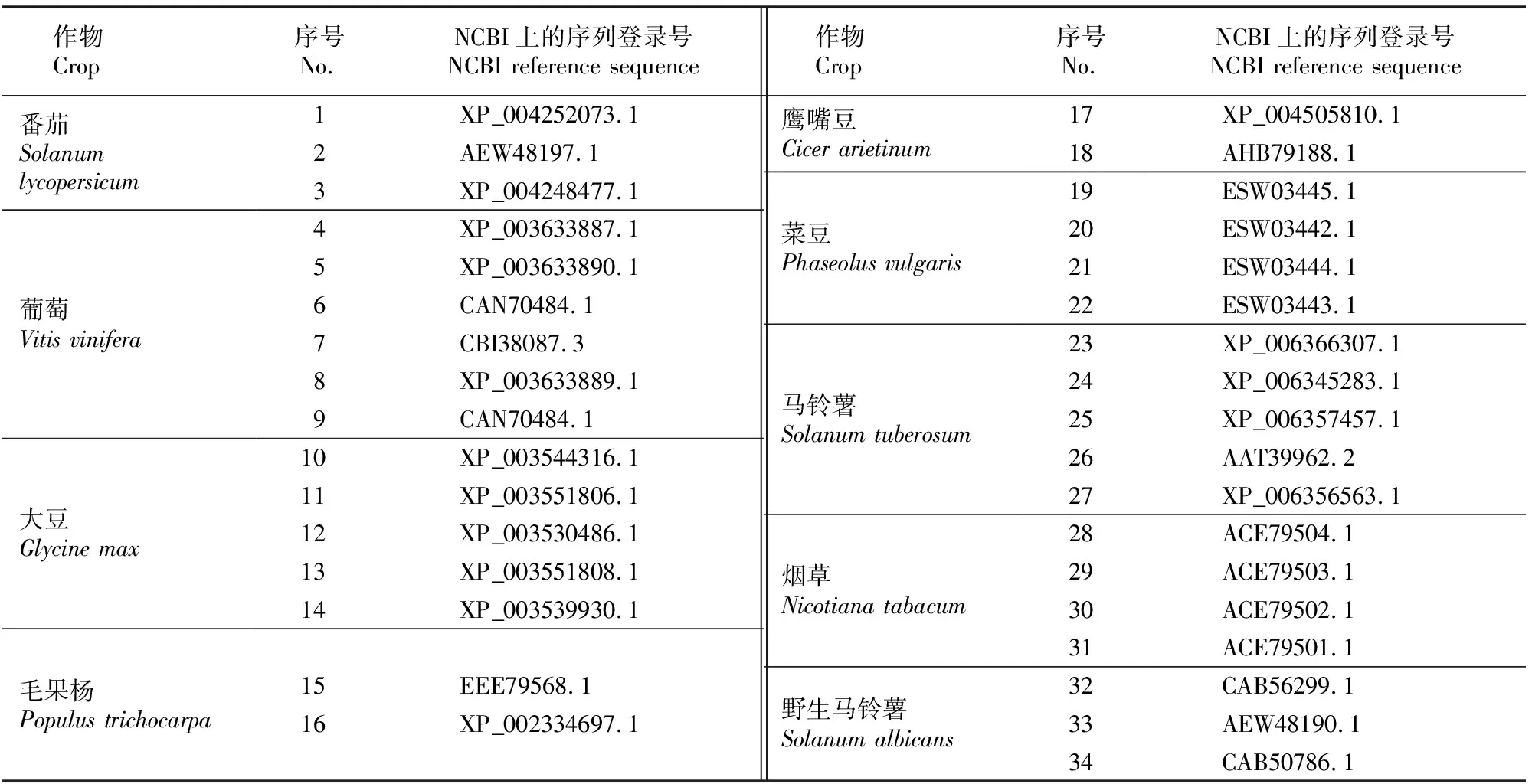
表2 用于与花生RGA序列相似性分析的其他作物的R序列
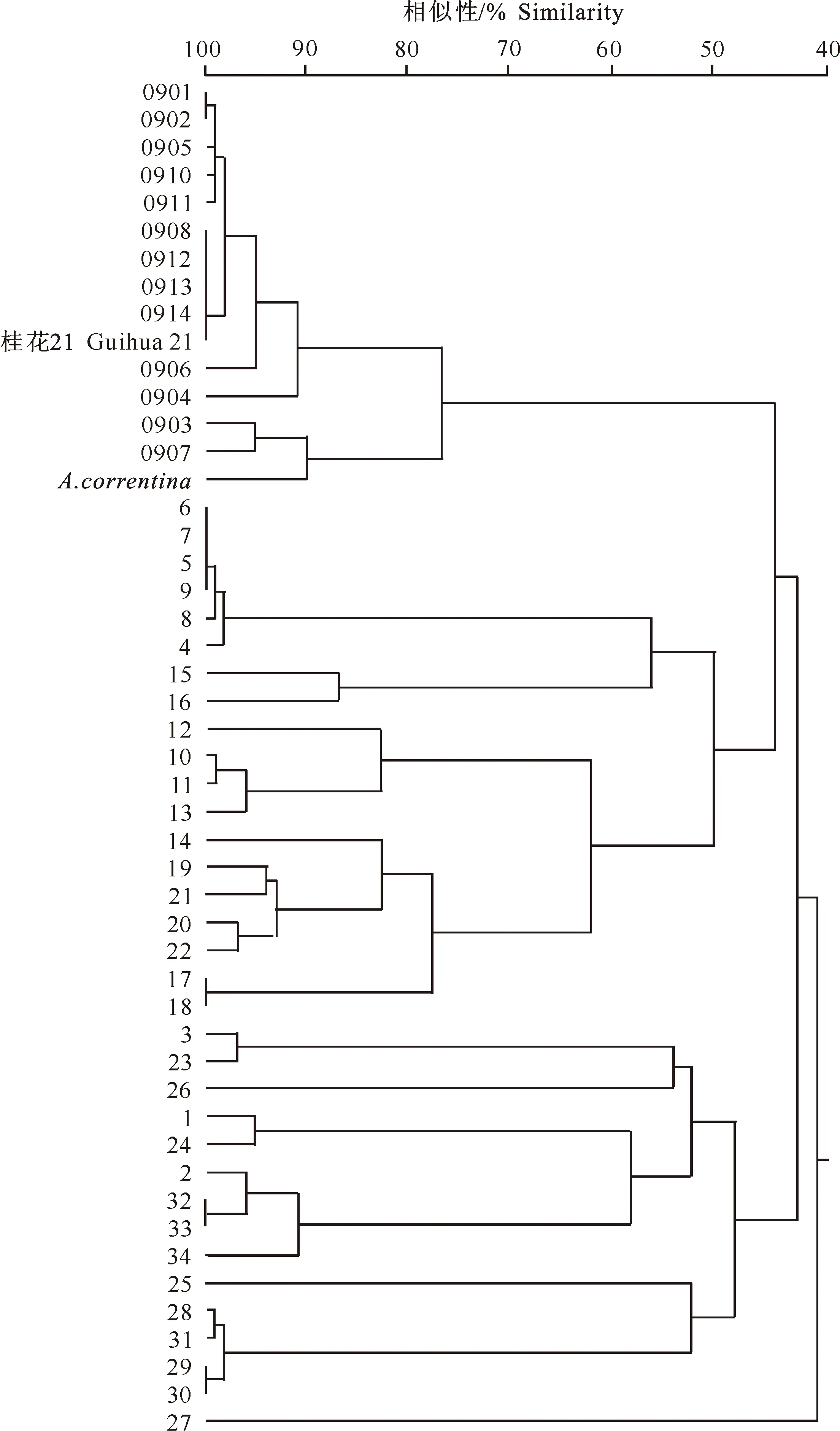
图4 花生RGA序列与其他作物的R氨基酸序列的相似性分析
3 讨 论
花生野生种的抗病基因可通过有性杂交转移至栽培品种中[18],获得高抗病性的花生新品系,这是花生抗病育种的创新途径之一。由于大多数野生种为二倍体,栽培种是四倍体,三倍体栽野种间杂种的育性需要染色体加倍才能恢复,杂种后代存在遗传不稳定性,野生种基因整合到栽培种基因组中的几率较低,因此如何准确地选择出含有野生种优良基因的目标单株显得非常重要。对花生属种间抗病基因同源序列进行克隆和分析,对深入明确抗病基因结构与功能的关系有重要意义。
本研究中15条氨基酸序列间以及与已发表的花生抗病基因类似物的同源性,均在72.1%以上,表明花生种间遗传基础窄、多态性水平低,这与其他研究结果[19-20]相似。15条氨基酸序列与其他作物的R氨基酸序列同源性在34.1%~54.6%,说明花生的RGA与其他植物之间的亲缘关系要远一些,这与经典分类学的结果基本一致。
本研究分离到的15个花生抗病基因同源片段,含有抗病基因保守结构域的N端糖基化位点、蛋白激酶C磷酸化位点、酪蛋白激酶Ⅱ磷酸化位点、N-豆蔻酰化位点,推测它们可能为抗病基因的部分序列。而且这些 RGA 的氨基酸序列存在差异,与已知花生的抗病基因相似性较高,与其他作物的RGA序列也存在一定的相似性,推断这些RGA 基因可能同属于一个基因家族。14个栽野杂交子代材料中有1个未扩增出抗病基因类似序列,各杂交子代间氨基酸序列存在单核苷酸差异位点,说明在子代繁育过程中可能发生了基因变异。
花生RGA序列的获得,对于花生抗病育种实践具有重要的现实意义。首先,可根据获得的RGA克隆设计特异引物继续扩增相关的抗病基因,获得基因全长序列及附近序列;另外,根据测序结果设计栽野种间抗病基因分子标记,有助于在种间杂种后代群体中选择含有野生种抗病基因的优良单株。
[参考文献]
[1] 万书波.中国花生栽培学 [M].上海:上海科学技术出版社,2003:245-269.
Wan S B.Chinese peanut cultivation [M].Shanghai:Shanghai Science and Technology Publishing House,2003:245-269.(in Chinese)
[2] 姜慧芳,段乃雄,任小平.花生种质资源的综合评价 [J].中国油料作物学报,1998,20(3):31-35.
Jiang H F,Duan N X,Ren X P.Evaluation of groundnut germplasm [J].Chinese Journal of Oil Crop Sciences,1998,20(3):31-35.(in Chinese)
[3] Mehan V K,Liao B S,Tan Y J,et al.Bacterial wilt of groundnut information bulletin No.35 [M].Patancheru,India:International Crops Research Institute for the Semi Arid Tropics,1994:1-20.
[4] 唐荣华,周汉群,蔡骥业.花生栽野种间杂交研究 [J].广西农业科学,1994(1):5-8.
Tang R H,Zhou H Q,Cai J Y.Study on peanut cultivated variety and wild varieties hybridization [J].Guangxi Agricultural Science,1994(1):5-8.(in Chinese)
[5] Sheikh M B,Sunil K P.Variations in methionine-rich protein composition of the genusArachis[J].Peanut Science,1984(1):1-3.
[6] 周 蓉.花生(野生种)叶部病害抗病性鉴定 [J].花生科技,1990(2):33-34.
Zhou R.Peanut (wild species) foliar disease resistance identification [J].Journal of Peanut Science,1990(2):33-34.(in Chinese)
[7] Bertioli D J,Leal-Bertioli S C M,Lion M B,et al.A large scale analysis of resistance gene homologues inArachis[J].Mol Gen Genomics,2003,270:34-45.
[8] Speulman E,Bouchez D,Holub E B,et al.Disease resistance gene homologs correlate with disease resistance loci ofArabidopsisthaliana[J].The Plant J,1998,14(4):467-474.
[9] Xiao W K, Xu M L,Zhao J R,et al.Genome-wide isolation of resistance gene analogs in maize (ZeamaysL.) [J].Theoretical and Applied Genetics,2006,113:63-72.
[10] Mago R,Nair S,Mohann M.Resistance gene analogues from rice:Cloning,sequencing and mapping [J].Theoretical and Applied Genetics,1999,99:50-57.
[11] Flieger S P,Lefebvre V,Caranta C,et al.Disease resistance gene analogs as candidates for QTLs involved in pepper-pathogen interactions [J].Genome,1999,42(6):1100-1110.
[12] Yuksel B,Estill J C,Schulze S R,et al.Organization and evolution of resistance gene analogs in peanut [J].Molecular Genetics Genomics,2005,274(3):248-263.
[13] 简桂良,赵 磊,张文蔚,等.陆地棉抗病基因同源序列的克隆与分析 [J].生物技术通报,2011(10):101-108.
Jian G L,Zhao L,Zhang W W,et al.Cloning and analysis of NBS type resistance gene analogs in upland cotton (Gossypiumhirsutum) [J].Biotechnology Bulletin,2011(10):101-108.(in Chinese)
[14] 魏 芳,马鸿翔.小麦抗赤霉病品种NBS同源序列克隆与分析 [J].生物技术通报,2012(2):128-135.
Wei F,Ma H X.Isolation and analysis of NBS analogs from wheat fusarium head blight resistant cultivars [J].Biotechnology Bulletin,2012(2):128-135.(in Chinese)
[15] 张向明,韦靖鸾,张 丹,等.野生稻NBS类抗病基因同源序列的分离与序列分析 [J].植物遗传资源学报,2010,11(6):741-748.
Zhang X M,Wei J L,Zhang D,et al.Isolation and sequence analysis of NBS resistance gene analogues in wild rice [J].Journal of Plant Genetic Resources,2010,11(6):741-748.(in Chinese)
[16] 王彦华,侯喜林,申书兴,等.不结球白菜抗病基因同源序列的克隆及分析 [J].中国农业科学,2006,39(12):2621-2626.
Wang Y H,Hou X L,Shen S X,et al.Isolation and characterization of resistant gene analogs fromBrassicacampestrisssp.chinensis[J].Scientia Agricultura Sinica,2006,39(12):2621-2626.(in Chinese)
[17] 颜子颖,王海林.精编分子生物学实验指南 [M].北京:科学出版社,1999:29-38.
Yan Z Y,Wang H L.Choreography molecular biology lab manual [M].Beijing:Science Press,1999:29-38.(in Chinese)
[18] 唐荣华,周汉群.花生属栽野杂种后代抗青枯病研究 [J].中国油料作物学报,2000,22(3):61-65.
Tang R H,Zhou H Q.Peanut plant wild bacterial wilt resistance research of hybrid progeny [J].Chinese Journal of Oil Crop Sciences,2000,22(3):61-65.(in Chinese)
[19] Raina S N,Rani V,Kojima T,et al.RAPD and ISSR fingerprints as useful genetic markers for analysis of genetic diversity,varietal identification,and phylogenetic relationships in peanut (Arachishypogaea) cultivars and wild species [J].Genome,2001,44(5):763-772.
[20] Milla S R,Isleib T G,Stalker H T.Taxonomic relationships amongArachissect.Arachisspecies as revealed by AFLP markers [J].Genome,2005,48(1):1-11.

