有机氯农药的微生物降解研究进展
田 云,姚财梁,肖慰祖,谭习羽,卢向阳
(1.湖南农业大学生物科学技术学院,湖南 长沙 410128;2.湖南省农业生物工程研究所,湖南 长沙 410128)
有机氯农药(organochlorine pesticides,OCPS)是20世纪80年代以前全球广泛使用的杀虫剂,但由于具有生物积蓄性、持久性、高毒性等特征,目前许多国家已经禁止使用[1-2]。但是近年来在水体、土壤、动植物体内能检测出有机氯农药残留,甚至有研究表明在北极也存在有机氯农药,更严重的是在人体血清中也检测到有机氯农药残留[3-7]。
随着有机氯农药对人类健康和环境危害的显现,如何安全、快捷地去除有机氯农药污染成为了国内外的研究热点。微生物因具有快速、有效、无污染地降解有机氯农药的特性,受到国内外研究者的高度重视。目前,有关有机氯农药的降解菌株的培育技术和相关降解理论的研究相对成熟,作者综述了近年来国内外在该领域的研究进展。
1 有机氯农药的简单介绍
1.1 有机氯农药的主要种类
有机氯农药的种类繁多,其中使用最早、运用最广、最具代表性的是DDT、六六六(hexachlorocyclohexane,HCH);此外,还有作为杀虫剂的氯丹、硫丹、艾氏剂、毒杀芬等;作为杀菌剂的五氯硝基苯、百菌清、道丰宁等;以及作为杀螨剂的三氯杀螨砜、三氯杀螨醇等。
1.2 有机氯农药在多介质中的迁移
有机氯农药不易降解、具有较强的亲脂性,容易在脂肪组织中聚集,并沿着食物链浓度逐级增大,经过多介质迁移最终被人体吸收,在肝、肾、心脏等器官中积蓄,对人体健康造成危害[8]。其主要迁移过程如图1所示。
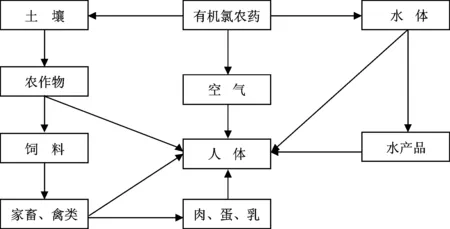
图1 有机氯农药的主要迁移过程
2 有机氯农药的微生物降解
2.1 有机氯农药降解菌
鉴于HCH和DDT具有高残留、高毒性和持久性等特点,被作为典型物质进行研究和分析。Nagata等[9]研究了将HCHs作为唯一碳源的SphingobiumjaponicumUT26菌株,发现了UT26的完整基因组序列,其中参与降解γ-HCH的基因主要分散在2个染色体和3个质粒上。陈婷[10]筛选获得HCHs降解菌SphingobiumindicumB90A,研究表明该菌对α-HCH和β-HCH的利用较好,其次是γ-HCH和δ-HCH。de Paolis等[11]研究发现,Arthrobactersp.能够降解HCHs,72 h后对α-HCH、β-HCH和γ-HCH的降解效率分别达到88%、60%和56%。高晶晶[12]以巢湖周边受DDT污染严重的土壤为材料,筛选获得DTT降解菌Alcaligenessp.DX8,研究发现该菌培养3 d后,对DDT的降解效率达到54.83%。茅燕勇等[13]筛选获得一株高效DDT降解菌Bacilluscereus2D-1,研究表明该菌在33 ℃、pH值6.5的最适条件下反应8 d后,对DDT的降解效率达到95.64%。
此外,硫丹也由于剧毒性而被广泛研究。Kong等[14]从硫丹污染严重的污泥中筛选出AlcaligenesfaecalisJBW4,研究发现该菌株培养5 d后,对α-硫丹和β-硫丹的降解效率分别达到83.9%和87.5%。Kalyani等[15]利用硫丹为唯一碳源,筛选获得降解菌PseudomonasaeruginosaSKL-1,研究表明该菌培养20 d内对α-硫丹和β-丹硫丹降解率分别达50.25%和69.77%。Singh等[16]将硫丹作为唯一硫源,分离获得降解菌株AchromobacterxylosoxidansC8B,研究发现该菌培养8 d后对α-对硫丹和β-丹硫丹的降解效率分别达到94.12%和84.52%。
研究人员还研究了其它有机氯农药高效降解菌。Sakakibara等[17]利用富集培养法分离获得狄氏剂降解菌Pseudonocardiasp. KSF27,研究发现该菌培养10 d后对狄氏剂的降解效率达到71.5%,并且该菌还能降解α-解硫丹、β-丹硫丹、硫丹硫酸盐、七氯、七氯环氧化物和十氯酮。肖鹏飞等[18]筛选获得七氯降解菌PhlebiaacanthocystisTMIC34875,研究表明该菌在35 ℃、pH值5.0的最适条件下反应1 h后对七氯的降解效率达到65%。杜芳芳[19]以七氯污染严重的土壤为材料,筛选获得4株高效降解菌株,其中H-3的降解效率最佳,在30 ℃、pH值8.0的最适条件下反应14 d后对七氯的降解效率达到80.01%。李成龙[20]以百菌清为唯一碳源筛选获得降解菌Pseudomonassp.B-7,研究表明该菌在30 ℃、pH值7.0的最适条件下反应14 d后对百菌清的降解效率达到85%。王光利等[21]从百菌清污染严重的土壤中筛选获得降解菌Pseudomonassp.CTN-3,研究表明该菌反应10 d后对百菌清的降解效率达到54%。部分降解有机氯农药的细菌见表1,部分降解有机氯农药的真菌见表2。

表1 部分降解有机氯农药的细菌

表2 部分降解有机氯农药的真菌[26]
2.2 微生物降解有机氯农药的主要机理
通过对SphingomonaspaucimobilisUT26和SphingomonaspaucimobilisB90等菌株降解有机氯农药过程的研究表明,有机氯农药的降解需要多种由相应基因编码的降解酶的共同参与,降解酶通过共同代谢、中间协同代谢完成对有机氯农药的降解。微生物中有机氯农药的降解酶主要包括如下几种:
2.2.1脱氯化氢酶
脱氯化氢酶是一种能从底物分子上脱去氯化氢形成碳碳双键的酶[27]。目前,报道最多的是从降解菌P.paucimobilisUT26中获得无需辅助因子的HCH脱氯化氢酶,该酶由linA基因编码[28]。Kumari等[29]从SphingomonaspaucimobilisB90中发现了linA的两个不同副本,分别是linA1和linA2。研究表明linA负责限速步骤,linA催化γ-HCH经过五氯苯,最终脱去3分子氯化氢,生成1,2,4-三氯苯[30]。还有在降解过程中需要辅助因子的脱氯化氢酶,如:从P.putida中鉴定到的三氯-D-丙氨酸-脱氯化氢酶[31]。
2.2.2水解酶
LinB是一种拥有广泛底物特异性的α/β-水解酶家族中的氯代烷烃脱氯酶,其与分离于XanthobacterautotrophicusGJ10的卤代烷烃脱卤酶DhlA、Moraxellasp.B的卤代乙酸脱卤酶DehH1和Pseudomonassp.CF600的2-羟基黏糠酸半醛水解酶DmpD具有高度的相似性[32-33]。研究认为LinB与底物结合,然后催化C-Cl键断裂,同时生成烷基-酶中间产物被水解。目前,Wu等[34]以Sphingomonassp.BHC-A为材料,克隆一个基因负责的β-HCH降解,转座子突变引入菌株BHC-A,序列研究表明该突变在一个卤代烷烃脱卤酶基因LinB2上。BLAST比对结果显示其与LinB具有97%的同源性,LinB与LinB2都能将γ-HCH转化为2,3,4,5,6-五氯环己醇,不同的是,LinB2还可将2,3,4,5,6-五氯环己醇转化为γ-氯环己醇,再进一步转化为四氯-1,4-环己二醇。
2.2.3脱氢酶
Nagata等[35]研究推断HCH降解过程中产生的酶LinC的氨基酸序列和LinX的短链乙醇脱氢酶家族有很高的相似性,因此认为LinC的酪氨酸和赖氨酸也高度保守,是一种NAD+依赖的脱氢酶,其将2,5-二氯环己二烯二醇转化为2,5-二氯氢醌[36]。Manickam等[37]编码LinB和LinC,通过研究DNA片段对应于两个初始基因在γ段的对应降解途径,证实了它们在itrc1基因组DNA进行Southern杂交的存在。
3 展望
随着人们环保意识的日益增强,成本低、无污染、效率高的微生物有机氯农药降解菌的发展前景广阔。尽管国内外众多研究者已筛选获得种类繁多的有机氯农药降解菌,但大多数降解菌仍存在受外部环境条件限制等问题。因此,有机氯农药降解菌的研究今后主要集中在以下几个方面:
(1)由于大部分受污染的土壤不是单纯的有机氯农药污染,而是包含其它多种污染在内的复合型污染。因此,在获得有机氯农药降解菌的基础上,发展复合型降解菌株或复合系,从而彻底、有效地改善土壤环境。
(2)进一步阐明微生物中有机氯农药的降解途径及机理,通过微生物基因组工程、代谢工程等技术构建适合于规模化降解有机氯农药应用的工程菌。
参考文献:
[1]KATSOYIANNIS A,SAMARA C.Persistent organic pollutants (POPs) in the conventional activated sludge treatment process:Fate and mass balance[J].Environ Res,2005,97(3):245-257.
[2]卢向明,陈萍萍.有机氯农药微生物降解技术研究进展[J].环境科学与技术,2012,35(6):89-93.
[3]王珺,祁士华,穆青,等.小海湾水体有机氯农药的浓度水平和特征[J].环境科学与技术,2007,30(8):33-34.
[4]汪光,吕永龙,史雅娟,等.北京东南化工区土壤有机氯农药污染特征和分布规律[J].环境科学与技术,2010,33(9):91-96.
[5]da CUNA R H,REY VAZQUEZ G,PIOL M N,et al.Assessment of the acute toxicity of the organochlorine pesticide endosufan inCichlasomadimerus(Teleostei,Perciformes)[J].Ecotoxicol Environ Saf,2011,74(4):1065-1073.
[6]VERREAULT J,MUIR D C,NORSTROM R J,et al.Chlorinated hydrocarbon contaminants and metabolites in polarbears (Ursusmaritimus) from Alaska,Canada,East Greenland,and Svalbard:1996-2002[J].Sci Total Environ,2005,351-352:369-390.
[7]MA W L,GAO C,BELL E M,et al.Analysis of polychlorinated biphenyls and organochlorine pesticides in archived dried blood spots and its application to track temporal trends of environmental chemicals in newborns[J].Environ Res,2014,133:204-210.
[8]BEMPAH C K,DONKOR A,YEBOAH P O,et al.A preliminary assessment of consumer′s exposure to organochlorine pesticides in fruits and vegetables and the potential health risk in Accra Metropolis[J].Food Chem,2011,128(4):1058-1065.
[9]NAGATA Y,OHTSUBO Y,ENDO R,et al.Complete genome sequence of the representativeγ-hexachlorocyclohexane-degrading bacteriumSphingobiumjaponicumUT26[J].J Bacteriol,2010,192(21):5852-5853.
[10]陈婷.SphingobiumindicumB90A降解六六六的特性及其对污染土壤的生物修复[D].南京:南京农业大学,2012.
[11]de PAOLIS M R,LIPPI D,GUERRIERO E,et al.Biodegradation ofα-,β-,andγ-hexachlorocyclohexane byArthrobacterfluorescensandArthrobactergiacomelloi[J].Appl Biochem Biotechnol,2013,170(3):514-524.
[12]高晶晶.有机氯农药的残留分析和DDT降解菌的初步研究[D].合肥:安徽农业大学,2012.
[13]茅燕勇,时号,马涛涛.DDT降解菌2D-1的分离鉴定与降解特性研究[J].安徽农业科学,2009,37(35):17810-17811.
[14]KONG L,ZHU S,ZHU L,et al.Biodegradation of organochlorine pesticide endosulfan by bacterial strainAlcaligenesfaecalisJBW4[J].J Environ Sci,2013,25(11):2257-2264.
[15]KALYANI S S,SHARMA J,SINGH S,et al.Enrichment and isolation of endosulfan-degrading microorganism from tropical acid soil[J].J Environ Sci Health B,2009,44(7):663-672.
[16]SINGH N S,SINGH D K.Biodegradation of endosulfan and endosulfan sulfate byAchromobacterxylosoxidansstrain C8B in broth medium[J].Biodegradation,2011,22(5):845-857.
[17]SAKAKIBARA F,TAKAGI K,KATAOKA R,et al.Isolation and identification of dieldrin-degradingPseudonocardiasp.strain KSF27 using a soil-charcoal perfusion method with aldrin trans-diol as a structural analog of dieldrin[J].Biochem Biophys Res Commun,2011,411(1):76-81.
[18]肖鹏飞,近藤隆一郎.一株木材腐朽菌及其粗菌酶液对七氯的降解[J].环境工程学报,2013,7(9):3683-3688.
[19]杜芳芳.七氯降解菌的分离及降解酶特性研究[D].西安:长安大学,2013.
[20]李成龙.百菌清降解菌的筛选及降解酶特性[D].长春:吉林农业大学,2012.
[21]王光利,陈宏宏,毕萌,等.假单胞菌菌株CTN-3对百菌清污染土壤的生物修复[J].应用生态学报,2012,23(3):807-811.
[22]SINGH A K,GARG N,LATA P,et al.Pontibacterindicussp.nov.,isolated from hexachlorocyclohexane-contaminated soil[J].Int J Syst Evol Microbiol,2014,64(1):254-259.
[23]SANGWAN N,LATA P,DWIVEDI V,et al.Comparative metagenomic analysis of soil microbial communities across three hexachlorocyclohexane contamination levels[J].PLoS One,2012,7(9):e46219.
[24]SKRYABIN G K,GOLOVLEVA L A,GOLOVLEV E L,et al.Decomposition of DDT and its analogs by soil microflora[J].Biol Bull Acad Sci USSR,1978,5(3):270-281.
[25]FOCHT D D,ALEXANDER M.Aerobic cometabolism of DDT analogues byHydrogenomonassp.[J].J Agric Food Chem,1971,19(1):20-22.
[26]郑永良,陈舒丽,刘德立.土壤中降解农药微生物的类别及降解特性[J].黄冈师范学院学报,2005,25(3):34-39.
[27]FETZNER S,LINGENS F.Bacterial dehalogenases:Biochemistry,genetics and biotechnological applications[J].Microbiol Rev,1994,58(4):641-685.
[28]NAGATA Y,HATTA T,IMAI R,et al.Purification and characterization ofγ-hexachlorocyclohexane (γ-HCH) dehydrochlorinase (LinA) fromPseudomonaspaucimoilis[J].Biosic Biotechnol Biochem,1993,57:1582-1583.
[29]KUMARI R,SUBUDHI S,SUAR M,et al.Cloning and characterization ofLingenes responsible for the degradation of hexachlorocyclohexane isomers bySphingomonaspaucimobilisstrain B90[J].Appl Environ Microbiol,2002,68(12):6021-6028.
[30]TRANTIREK L,HYNKOVA K,NAGATA Y,et al.Reaction mechanism and stereochemistry ofγ-hexachlorocy clohexane dehydrochlorinase LinA[J].J Biol Chem,2001,276(11):7734-7740.
[31]NAGASAWA T,OHKISHI H,KAWASAKI B,et al.3-Chloro-D-alanine chloridelyase (deaminating) ofPseudomanasputidaCR1-1[J].J Biol Chem,1982,257(22):13749-13756.
[32]OLLIS D,CHEAH E,CYGLER M,et al.Theα/β-hydrolase fold[J].Protein Eng,1992,5(3):197-211.
[33]NAGATA Y,PROKOP Z,SATO Y,et al.Degradation ofγ-hexachlorocyclohexane by haloagenase LinB fromSphingomonaspaucimobilisUT26[J].Appl Environ Microb,2005,71(94):2183-2185.
[34]WU J,HONG Q,HAN P,et al.A genelinB2 responsible for the conversion ofγ-HCH and 2,3,4,5,6-pentachlorocyclohexanol inSphingomonassp.BHC-A[J].Appl Environ Microb,2007,73(5):1097-1105.
[35]NAGATA Y,OHTOMO R,MIYAUCHI K,et al.Cloning and sequencing of a 2,5-dichloro-2,5-cyclohexadiene-1,4-diol dehydrogenase gene involved in the degradation ofγ-hexachlorocyclohexane inPseudomonaspaucimobilis[J].J Bacteriol,1994,176(11):3117-3125.
[36]MIYAUCHI K,ADACHI Y,NAGATA Y,et al.Cloning and sequencing of a novel meta-cleavage dioxygenase gene whose product is involved in degradation ofγ-hexachlorocyclohexane inSphingomonaspaucimobilis[J].J Bacteriol,1999,181(21):6712-6719.
[37]MANICKAM N,MAU M,SCHLOMANN M.Characterization of the novel HCH-degrading strain,Microbacteriumsp.ITRC1[J].Appl Microbiol Biotechnol,2006,69(5):580-588.

