拟南芥花分生组织决定基因AGL24对花发育的影响
江为 谷慧英 王志敏 宋明 汤青林
(西南大学园艺园林学院 南方山地园艺学教育部重点实验室 重庆市蔬菜学重点实验室,重庆 400715)
拟南芥花分生组织决定基因AGL24对花发育的影响
江为 谷慧英 王志敏 宋明 汤青林
(西南大学园艺园林学院 南方山地园艺学教育部重点实验室 重庆市蔬菜学重点实验室,重庆 400715)
AGAMOUS-LIKE 24(AGL24)基因编码MADS蛋白,在植物花发育的不同时期发挥着重要的作用。综述了AGL24如何通过和其他花分生组织决定基因的相互作用来影响拟南芥花的发育,调节开花时间,这将有助于人们对开花基因调控网络有更进一步的认识,能够在生产上有效的调控开花时间,从而为植物育种提供借鉴。
拟南芥 AGL24 花发育
高等植物通过自主途径、光周期途径、低温春化途径以及赤霉素途径等响应发育及环境信号,调节植物从营养生长向生殖生长转变。在拟南芥中,自主途径会响应植物不同发育时期的内源性信号;光周期途径和春化途径受光照时长和温度变化等环境信号调控[1,2];赤霉素是非诱导性条件下开花所必须的,施加外源赤霉素能够促进开花。目前对拟南芥突变体库的研究发现,至少有20个基因参与对植株开花发育的调控。对突变体的表型与上位性关系的研究发现,这些开花调控基因分布在不同的开花途径中[3-5],调节植物开花时间。而植物成花过程是由一个复杂的基因网络调控的,在这个调控网络中,MADS-box转录因子中的SHORT VEGETATIVE PAHSE(SVP)和AGAMOUS-LIKE24(AGL24)在生殖发育的不同时期发挥着相当重要的作用[6-9]。这两个基因序列同源性较高,但是在开花转换时期它们的功能却截然相反。SVP是抑制开花的因子,而AGL24却能够促进开花转换。目前,有关SVP的功能和作用机制已有较多报道,但是花分生组织决定基因AGL24的分子机制鲜有报道。本文是在前人研究基础上综述了拟南芥AGAMOUS-LIKE 24(AGL24)的功能及其与互作基因调控拟南芥花发育和影响开花时间的机制(图1),这为人们深入了解AGL24在开花调控网络中的重要作用提供借鉴。
1 AGL24基因的表达特性
在拟南芥中,AGL24编码典型的MADS域蛋白,
包含N端保守的MADS-box结构域和第88-156残基间相对保守的K-box结构域。序列比对发现,AGL24与开花负调因子SHORT VEGETATIVE PHASE(SVP)高度相似[6]。原位杂交表明:AGL24能在营养生长期的茎端分生组织、新叶原基的整个区域以及生殖生长期的叶片维管束中表达。

图1 调控拟南芥花发育的基因模式图
拟南芥的花发育可细分为20个时期,每个发育时期的的特点各异(图2,表1)[10]。在花发育的第1个时期,AGL24在茎尖和新生花分生组织中表达。在花发育的第2、3时期以及整个花絮分生组织中,AGL24也都有表达。在花发育的第6个时期,AGL24基因的表达局限于雌蕊和雄蕊的原基中。到了花发育的第8个时期,AGL24主要在心皮和胚珠内中表达。
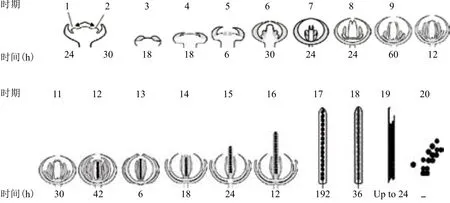
图2 拟南芥花发育的20个时期

表1 花发育不同时期的标志性事件
研究发现,在AGL24持续表达的转基因拟南芥植株中,萼片的腋处会有花芽的产生。然而在SVP持续表达的转基因拟南芥植株中也有类似的表型,植株产生了异位花。这说明在花发育的早期阶段(花发育时期1和2)AGL24和SVP在功能上具有一定的冗余性[11]。在ap1突变体中也有观察到异位花,通过原位杂交试验发现在ap1突变体中AGL24和SVP均异位表达了,这说明异位花的形成是由于这两个基因的异位表达造成的。这一点在ap1 agl24或ap1 svp突变体中也得到了证实[12,13],在这些双突变体中,异位花的数量明显降低。由此可以说明在ap1突变体中AGL24和SVP的异位表达的确是异位花芽形成的原因;同时也说明了在花发育第2个时期的后期,AGL24和SVP在花的中心必须被抑制以阻止异位花的形成。Liu等[12]认为这种抑制作用是由于AP1蛋白通过直接结合到AGL24和SVP启动子上的多个MADS-box蛋白结合位点(CArG boxes),从而抑制AGL24和SVP异位表达。在拟南芥中,AGL24的过量表达可以促进开花并能够使花分生组织转化为花序分生组织。由花分生组织基因APETALA1(AP1)直接抑制AGL24和另外两个调控花期基因SOC1和SVP,从而抑制了茎发育进程,促进花分生组织特征的形成[12,13]。在高温条件下,
花分生组织中的AGL24和SVP表达量对B类和C类花同源基因的调控至关重要[14]。因此,AGL24在调控花的发育和开花时间上发挥着非常重要的作用。
最近有研究发现,在大王花(又名“霸王花”,Rafflesia,一种肉质寄生草本植物,这种寄生性植物有着植物世界最大的花朵,它一生中只开一朵花且没有叶片)中含有一个和拟南芥中AGL24高度同源的基因RcMADS1,其同源度高于SVP。RcMADS1和AGL24相似,能够弥补agl24-1和FRIGIDA的晚花表型,但是对于svp-41的早花表型无影响[15]。
2 AGL24与SVP功能差异
已有报道表明5个MADS-box基因对拟南芥的开花起着调控作用:FLC、FLM/MAF1和SVP作为开花抑制因子,而FUL和SOC1作为开花促进因子,AGL24与花的抑制基因SVP有着非常高的相似性,但在调控开花时间上的功能存在差异。过量表达SVP的植株表现为异常开花,且花器官发育出现畸形。这表明SVP在调控开花时间和花的发育过程中有多种功能。与此类似的是开花促进基因FT和抑制基因TFL也是序列高度相似,但对于开花却起着截然相反的作用[16,17]。在营养发育期,SVP蛋白作为开花核心调节子直接抑制茎尖和叶中的开花整合子SUPPRESSOR OF OVEREXPRESSION OF CO 1(SOC1)的表达[18]。另外,SVP也能调控开花途径整合子FLOWERING LOCUS T(FT)的表达[18,19]。SVP蛋白与另一个开花抑制因子FLOWERING LOCUS C(FLC)相互作用,抑制开花[18]。与SVP蛋白的抑制功能相反,AGL24和FT蛋白在开花转变期能够促进SOC1的表达[8,20-22]。AGL24和SVP蛋白与AP1、LUG和SEU相互作用形成多元复合物,抑制AG的表达[9,14]。
3 AGL24与SOC1相互调控
虽然AGL24基因序列与SVP高度相似,但是agl24突变体的晚花表现型与soc1非常相似。agl24和soc1均表现出正常的光周期响应,但在长日照及短日照条件下是晚花。除此之外,两个突变体的晚花表型不能被春化作用有效抑制。结合遗传分析与表达特性研究发现:AGL24是SOC1的一个正调控因子,在含有FRI株系或者自主途径突变体中,SOC1的表达水平通常很低,这是由于FLC的高度表达造成的,但是AGL24的超表达会引起这些背景系和早花表型植株中SOC1的上调。有趣的是,SOC1的超表达也可以引起AGL24表达水平的上调,因此,这两个含有MADS域的转录因子可以彼此正调控。AGL24与SOC1分别超表达对soc1和agl24突变体的开花时间均有影响[8]。
AGL24和SOC1之间除能相互正调控,也可独立促进花的发育。二者功能上具有相似性,都能通过低温春化作用上调。但它们之间的作用机制并不相同,SOC1被FLC负调控。在哥伦比亚野生型拟南芥(Col)中,FLC的水平相对较低,SOC1的表达水平很高,在之前所提到的含有FRI株系或是自主途径突变体中,FLC抑制了SOC1的表达。春化作用能抑制FLC的表达,从而促进SOC1的高度表达。所以FRI株系以及自主途径突变体在经历春化作用后,SCO1上调而FLC下调。AGL24却相反,不受FLC调控。在FRI具有活性或活性丧失的植株经历春化作用后,AGL24的表达均上调。因此,AGL24的表达被春化作用上调是独立于FLC调控途径的,不受其调控。有试验表明缺失FLC的突变体呈现出春化响应,这进一步说明AGL24的调控是独立于FLC的春化途径中的调控因子[8]。
AGL24和SOC1能够调控彼此的mRNA的水平,它们的表达模式具有一定的冗余性。在蔬菜作物中克隆得到的AGL24和SOC1同源基因在花序分生组织中大量表达[8]。然而原位杂交显示,AGL24在花原基时期到花发育第2个时期均能大量表达。SOC1却相反,在新生花原基中不表达,但是在花发育第2个时期的花中重新出现,并且持续到花发育的整个过程,在花蕊和心皮中也可检测到SOC1的表达[23]。AGL24和SOC1在表达模式上的不同表明这两个基因在调控拟南芥花的发育上有一些不同的作用。例如,AGL24的过量表达会引起花的异常发育,但是目前还没有关于SOC1的超表达会引起花异常的报道。其次,当AGL24在soc1突变体中过量表达时,花异常现象的发生频率会大大降低,说明AGL24与SOC1基因在花发育中的作用具有一定的冗余性。
在调控开花时间上AGL24与SOC1能影响彼此间的表达[7,8],AGL24和SOC1在开花调控中的差
异:首先,虽然AGL24的表达被春化所调控,但它不受开花抑制因子FLC的影响。相反,FLC在分生组织中抑制SOC1的表达并通过抑制FT延迟SOC1的表达。FT编码的蛋白作为一种开花信号,可从叶维管组织中长距离运输到茎尖分生组织[21,24,25];其次,在光周期途径中,AGL24被CONSTANS(CO)影响却不受FT调控[7],但SOC1主要受FT直接调控。同时,CO通过一个目前未知的DNA结合因子间接的调控SOC1[23,25,26]。最后,AGL24活性的改变决定开花时间,部分异于SOC1;反之亦然,说明AGL24与SOC1可在各自独立的途径中促进开花[7,8]。
4 AGL24受AP1调控
拟南芥的花由4种类型的花器官组成,即花萼、花瓣、雄蕊和心皮。花器官排列成向心的圆环形,称作轮性(whorl)。拟南芥的花由4个花萼组成最外的第1轮、依次向内为4个花瓣组成第2轮,6个雄蕊组成第3轮,两个融合的心皮组成第4轮。
在花发育过程中,AP1蛋白参与调控AGL24和SVP的表达。AP1结合到这些基因的上游调控区域,并且在ap1突变体的花发育过程中AGL24和SVP的异位表达导致了在第1轮器官的叶腋内有花芽的形成。在ap1-10突变体中,第1轮中含有苞片状的器官,在萼片中含有二次花,没有花瓣并且在第3轮中有少量的雄蕊。将ap1-10突变体与agl24-2 svp-41突变体杂交,后代表现为花分生组织缺失,但能持续产生花序分生组织,这与ap1-1 cal-1双突变体的表型非常相似[27,28],表明AP1、SVP和AGL24对花分生组织形成具有一定的冗余性。
另外,自花发育的第2个时期以后,除了AP1,其他花器官决定基因如SEPALLATA1(SEP1)、SEP2、SEP3和AG也会调控AGL24和SVP,因为在ap1、ag以及sep1 sep2 sep13突变体中,AGL24和SVP的异位表达形成异位花。AGL24和SVP对AP1有部分的冗余作用,但其功能并不完全相同,AP1蛋白对其起关键的调控作用。AP1和CAL或AP1和SVP/AGL24的缺失将会完全抑制花的形成,但CAL和SVP/AGL24的缺失却因AP1的活性仍可诱导花的产生。agl24-2 svp-41 cal-01突变体与agl24 svp-41突变体的表型差异不大,两个突变体在分子水平上呈现出相似的特点。其突变体中的LFY、AP1的表达水平均减少,其变化规律相似,FMI基因表达量会减少但并不会消失。在这两突变体中,AP1可能不与SOC1蛋白互作,而是与其他的蛋白发生相互作用。在agl24-2 svp-41 cal-01 soc1-2突变体中FMI没有消失,表明在没有AGL24和SVP蛋白作用的情况下,SOC1蛋白的存在与否对AP1蛋白无关键性影响。
5 AGL24调控TFL1的表达
拟南芥的花序为总状花序,在新生花分生组织中,AGL24、SVP、SOC1及SEP4通过抑制TFL1的表达共同调控其花序的分支,TFL1也可影响LFY和AP1的表达,从图3可以看到在野生型花序分生组织(IM)的横向的分生组织中,SEP4、SOC1、AGL24及SVP对于AP1抑制TFL1的表达不可或缺,若这4个基因缺失会使TFL1抑制AP1的表达,从而使花分生组织(FM)转化为花序分生组织(IM)[29]。在新生的花分生组织中,AP1和CAL需要TFL1被抑制才能表达[27,28,30,31]。此外,Folter等[32]通过酵母试验发现,AP1、CAL基因能与AGL24、SOC1、SVP和SEP4蛋白发生相互作用。双分子荧光互补(BiFC)检测显示,在植物细胞中AP1或CAL蛋白与AGL24、SOC1、SVP或SEP4蛋白发生了相互作用。在ap1-1 gAP1-2HA转基因家系中,融合有2HA的AP1能在很大程度上弥补ap1-1的开花突变表型。对ap1-1 g AP1-2HA进行免疫共沉淀分析显示:AP1∶2HA蛋白被抗体特异性沉淀下来,这些抗体分别抵抗AGL24、SOC1、SVP及SEP4蛋白。由此表明,AGL24、SOC1、SVP和SEP4蛋白也许是通过与AP1或CAL蛋白在体内相互作用来抑制TFL1基因的表达。研究发现,35S∷AP1对soc1-2 agl24-3 svp-41 sep4-1突变体的花序分支没有有效的抑制,这表明AGL24、SOC1、SVP和SEP4对AP1赋予花分生组织特性必不可少[29]。
为了进一步确定AGL24、SOC1、SVP和SEP4是否间接的抑制TFL1,Wellmer等[33]用地塞米松诱导的AP1-GR系统对ap1-1 cal-1突变体背景的家系进行染色体免疫共沉淀分析(CHIP)。利用这个系统他们发现在花序顶端对AP1蛋白进行1 h的地塞米松诱导处理后,TFL1的表达量下降
50%。CHIP分析表明在地塞米松处理的花序顶端,AGL24、SOC1和SVP附着到TFL1基因部位上的区域与AP1附着区域重叠[34],但其在空白处理的组织中未发现有结合,证实这3个蛋白结合到TFL1部位上依靠的是AP1蛋白。利用CHIP进一步发现在野生型的花序顶端SVP蛋白主要结合于TFL1上,而在svp-41中,AGL24和SOC1蛋白主要于TFL1上。这3个蛋白的结合区域与在地塞米松处理的ap1-1 cal-1 35S:AP1-GR材料中所发现的结合区域一样。但在soc1-2 agl24-3 svp-41突变体中,SEP4蛋白的结合区域和之前AGL24、SOC1和SVP蛋白结合到TFL1的部位相同。这与在soc1-2 agl24-3 svp-41突变体中SEP4的异位表达说明SEP4蛋白只有在AGL24、SOC1和SVP都缺失的情况下才会直接抑制TFL1的表达[29]。可见,AGL24、SOC1、SVP及SEP4间接的抑制TFL1的表达来共同调控拟南芥花序的分枝。
最近Chang等[29]研究发现,在水稻中这些MADS-box基因(SVP,AGL24,SOC1,SPE4)的同源基因通过调控类似于TFL1基因的表达来决定穗的分枝。他们的发现揭示了在开花植物中有一个决定花序结构的保守遗传途径,这也为作物生产提供了一个很重要的理论依据。
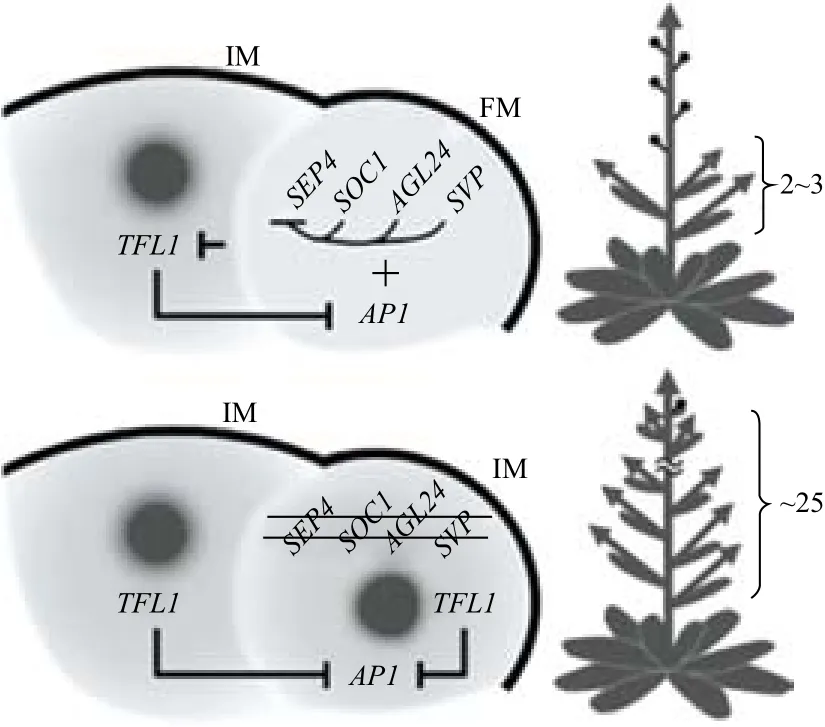
图3 花分生组织中MADS-box基因协同抑制TFL1的表达来调控花序的结构[29]
6 AGL24抑制B、C类花器官基因
拟南芥花器官的同源异型突变有3大类型,第一类为第1轮和第2轮器官受影响,产生心皮状的花萼和雄蕊状的花瓣;第二类为第2轮和第3轮器官受影响,产生花萼状的花瓣和心皮状的雄蕊;第三类为第3轮和第4轮器官受影响,产生花瓣状的雄蕊和花萼状的心皮,而且最内两轮器官的数量和轮数也发生了改变。这表明至少有一些基因在决定拟南芥花器官性质上具有同源的功能,而具有同源功能的基因在结构上可能也会有保守性,因此将这些基因称为同源异型基因。
基于拟南芥的3类同源异型突变以及它们之间的遗传关系,Coen和Meyerowitz提出了花器官发育的ABC模型。随着研究的深入和克隆出的花同源异型基因数量的增加,人们在拟南芥中又相继发现了D类和E类基因,即目前广泛所接受的拟南芥花发育的ABCDE模型。在拟南芥中,A类以AP2(apetala2)基因为代表,B类以AP3(APETALA3)基因和PI(PISTILLATA)基因为代表,C类以AG(AGAMOUS)基因为代表。D类基因包括SEEDSTICK(STK)、SHATTERPROOF1(SHP1)和SHATTERPROOF2(SHP2),这些基因共同参与控制了胚珠的发育。E类基因包括SEPALLATA1(SEP1),SEPALLATA2(SEP2),SEPALLATA3(SEP3)和SEPALLATA4(SEP4)。ABC类基因与SEP基因联合表达可以使叶片转化成为完整的花器官。每一类同源异型基因对邻近的两个轮起作用,这样每一轮都对应着不同的同源异型基因的组合,即A类同源异型基因独自作用于第1轮;A和B类同源异型基因共同作用于第2轮;B和C类同源异型基因共同作用于第3轮;C类同源异型基因独自作用于第4轮。
当agl24-2 svp-41突变体和ap1-12弱突变体杂交后会严重影响花的发育[14]。在这些花中,控制花瓣、雄蕊以及心皮特性的B类和C类基因提早表达且没有局限于某一特定轮生器官,这就导致了花器官数量的减少并使得各轮生器官性质具有相似性。由于该表型只在agl24-2 svp-41突变体与 ap1-12弱突变体的杂交后代中出现,并考虑到SVP仅仅在花发育的第1、2个时期表达,因此,这极有可能是花器官决定基因AGL24、AP1的在花发育早期异位表达导致花器官基因的异常。把agl24-2 svp-41突变体
与ap1-10强突变体杂交,这种花发育表型并未得到改善。而其表型类却似于ap1-1 cal-1突变体,花的发育在很大程度上受到抑制,然而花序分生组织大量增加,说明花分生组织特征在agl24-2 svp-41突变体与ap1-10强突变体杂交后代中消失[9]。
关于花分生组织的形成和生长(花发育的第1和2个时期),Gregis等[35]均认为AP1和SVP或AGL24首先相互作用形成对花分生组织特性和花器官的形成具有重要作用的二聚体,这些二聚体抑制控制花器官形成的基因。随后,当SVP和AGL24在花发育第2个时期末被抑制,AP1蛋白和SEP蛋白(从第2时期末开始表达)则发生相互作用,花器官的发育也就开始了。花分生组织形成在拟南芥花发育期是至关重要的一个时期,Gregis等提出了有关AGL24,SVP和AP1蛋白在花发育早期对AG,AP3,PI,SEP3的调控机制。他们发现AGL24,AP1和SVP能直接附着于B类和C类花器官基因的调控区域,从而说明了这3个蛋白在花分生组织形成时期的基本作用。Gregis等同时也发现了AP1-SVP或AP1-AGL24二聚体能与SEU-LUG复合体发生聚合,抑制靶基因SCO1。并且在agl24-2 svp-41突变体中,他们通过对SOC1的原位表达分析发现在花发育的第1、2个时期,SOC1异位表达了。这说明在花发育的早期,SOC1与AP1蛋白相互作用直接抑制B类和C类基因,以弥补AGL24、SVP的缺失[35]。
AP1与SVP或AGL24蛋白互作对花器官形态建成相当重要。AP1,AGL24和SVP在花发育的第1、2个时期,花分生组织开始变大但是花器官原基还未分化,这对花的形成至关重要,它们的缺失会影响花的后期发育。从花发育第3个时期开始,AP1结合到SEP蛋白上,诱导花器官发育[35]。
AGL24蛋白和SVP蛋白能直接作用于E类基因SEP3,而且B类基因和C类基因的异位表达因SEP3的异位引起[36]。但SEP3表达活性也许并非真正的影响因子,因为在花发育早期时期B类、C类花器官基因的活性应该受到抑制。Liu等[36]利用原位杂交分析表明:B类基因(AP3,PI)和C类基因(AG)的表达在agl24 svp soc1 sep3突变体中没有完全被抑制。另外,先前的研究已证实AG的调控机制是通过激活和抑制建立的[37-41]。由LUG蛋白通过作用于AG的第2个内含子达到抑制作用[37,39],这和Hong等[42]所观察到的AP1-SVP和AP1-AGL24二聚体附着于AG的第2个内含子的CArG-box上并和LUG和SEU相互作用相一致。其次,在这些CAr-Gbox上的突变会引起花发育的早期时期AG的异位表达。AP1-SVP和AP1-AGL24二聚体结合到AG第二内含子上的CArG-box并与LUG、SEU相互作用,从而抑制LUG、SEU的活性[37,39]。另外,在这些CArG-box上的突变会引起花发育早期时期AG的异位表达[42]。在花分生组织中,SVP似乎是在抑制花器官决定基因上起主要作用,这是由于AGL24没有结合到任何B类、C类和E类基因所致。
7 展望
AGL24、SVP、FLC和SOC1等开花时间控制基因均属于典型的TypeⅡMADS-box基因MIKCC型,它们在植物体内行使的特殊功能与其为MIKC型蛋白有着紧密的联系。TypeⅡ型MADS域蛋白中包含的功能区域:MADS、Intervening(I)、Keratin-like(K)和Carboxyl-terminal(C)4个结构域,即成为MIKC型蛋白。AGL24编码的是一个典型的MADS-box蛋白,与SVP的序列高度同源,两者只在C端有些差异[6]。而C域位于K域下游为富含疏水残基的非保守区域,与MADS域、I域和K域不同的是C域不参与蛋白相互作用域和靶DNA的结合区[43]。但将C域突变会造成植物功能性缺陷。在功能域划分中发现,C域对AP1、AP3、PI和AG相互作用的特异性没有影响,其可能参与多聚体对靶DNA的转录激活。AP1蛋白和其同源蛋白的C域具有对靶DNA的转录激活功能。但目前对于C域在MADS-box蛋白中的功能了解甚少,还需进一步研究[44]。
AGL24和SOC1调控拟南芥花发育的表达模式不同,说明其在调控发育有不同的作用。如AGL24的过量表达会引起花的异常发育,但是目前尚无有关SOC1超表达引起花异常的报道。其次,当AGL24在soc1突变体背景中过量表达时,花异常现象的发生频率会大大降低,这表明AGL24与SOC1基因在花发育中的作用具有一定的冗余性。关于这两个基因在调控花发育上有何不同的作用目前还有待研究。
在拟南芥中,作为TFL1上游的调控因子,
AGL24、SOC1、SVP以及SEP4在由营养生长到生殖生长的开花转变时期发挥着不同的作用。AGL24和SOC1是整合不同遗传途径中开花信号的开花启动子,然而SVP作为一个开花抑制因子和另外一个开花抑制因子FLC相互作用来共同抑制两个开花途径整合因子FT和SOC1的表达。SEP4和SEP3高度同源,它能够介导MADS-box蛋白形成各种高阶复合物[45,46]。另外,SEP4在许多组织中均有表达,但是在SEP4的功能缺陷的突变体,植株并没有表现出可见的缺陷[47]。因此,在发育的许多过程中,SEP4与一些相关MADS-box蛋白在功能上具有冗余性。但是SEP4对调控发育各种程序中的高阶MADS-box复合物的形成是否产生影响还需要进一步研究。
[1] Koornneef M, Alonso-Blanco C, Peeters AJM, et al. Genetic control of flowering time in Arabidopsis[J]. Ann Rev Plant Physiol, Plant Mol Biol, 1998, 49:345-370.
[2] Simpson GG, Dean C. Arabidopsis, the Rosetta stone of flowering time?[J]. Science, 2002, 296:285-289.
[3] Mouradov A, Cremer F, Coupland G. Control of flowering time:interacting pathways as a basis for diversity[J]. Plant Cell, 2002, 14:S111-S130.
[4] Koornneef M, Alonso-Blanco C, Blankestijn-de Vries H, et al. Genetic interactions among late-flowering mutants of Arabidopsis[J]. Genetics, 1998, 148:885-892.
[5] Levy YY, Dean C. The transition to flowering[J]. Plant Cell, 1998, 10:1973-1989.
[6] Hartmann U, Ho-hmann S, Nettesheim K, et al. Molecular cloning of SVP:a negative regulator of the floral transition in Arabidopsis[J]. Plant J, 2000, 21:351-360.
[7] Yu H, Xu Y, Tan EL, et al. AGAMOUS-LIKE 24, a dosagedependent mediator of the flowering signals[J]. Proc Natl Acad Sci USA, 2002, 99:16336-16341.
[8] Michaels SD, Ditta G, Gustafson-Brown C, et al. AGL24 acts as a promoter of flowering in Arabidopsis and is positively regulated by vernalization[J]. Plant J, 2003, 33:867-874.
[9] Gregis V, Sessa A, Colombo L, et al. AGAMOUS-LIKE24 and SHORT VEGETATIVE PHASE determine floral meristem identity in Arabidopsis[J]. Plant J, 2008, 56:891-902.
[10] Elena RA, Mariana B, Adriana C, et al. Flower development[M]. The Arabidopsis Book, 2010.
[11] Masiero S, Li MA, Will I, et al. INCOMPOSITA:a MADS-box gene controlling prophyll development and floral meristem identity in Antirrhinum[J]. Development, 2004, 131:5981-5990.
[12] Liu C, Zhou J, Bracha-Drori K, et al. Specification of Arabidopsis floral meristem identity by repression of floweringtime genes[J]. Development, 2007, 134:1901-1910.
[13] Yu H, Ito T, Wellmer F, et al. Repression of AGAMOUS-LIKE 24 is a crucial step in promoting flower development[J]. Nat Genet, 2004 36:157-161.
[14] Gregis V, Sessa A, Colombo L, et al. AGL24, SHORT VEGETATIVE PHASE, and APETALA1 redundantly control AGAMOUS during early stages of flower development in Arabidopsis[J]. Plant Cell, 2006, 18:1373-1382.
[15] Ramamoorthy R, Phua EE, Lim S, et al. Identification and characterization of RcMADS1, an AGL24 Ortholog from the Holoparasitic Plant Rafflesia cantleyi Solms-Laubach(Rafflesiaceae)[J]. PLOS ONE, DOI:10.1371/journal.done.0067243.
[16] Kardailsky I, Shukla VK, Ahn JH, et al. Activation tagging of the floral inducer FT[J]. Science, 1999, 286(5446):1962-1965.
[17] Kobayashi Y, Kaya H, Goto K, et al. A pair of related genes with antagonistic roles in mediating flowering signals[J]. Science, 1999, 286(5446):1960-1962.
[18] Li D, Liu C, Shen L, et al. A repressor complex governs the integration of flowering signals in Arabidopsis[J]. Dev Cell, 2008, 15:110-120.
[19] Lee JH, Yoo SJ, Park SH, et al. Role of SVP in the control of flowering time by ambient temperature in Arabidopsis[J].Genes Dev, 2007, 21(4):397-402.
[20] Yoo SK, Chung KS, Kim J, et al. Constans activates suppressor of overexpression of constans 1 through Flowering Locus T to promote flowering in Arabidopsis[J]. Plant Physiol, 2005, 139:770-778.
[21] Searle I, He Y, Turck F, et al. The transcription factor FLC confers a flowering response to vernalization by repressing meristem competence and systemic signaling in Arabidopsis[J]. Genes Dev, 2006, 20:898-912.
[22] Liu C, Chen H, Er HL, et al. Direct interaction of AGL24 and SOC1 integrates flowering signals in Arabidopsis[J]. Development, 2008, 135:1481-1491.
[23] Samach A, Onouchi H, Gold SE, et al. Distinct roles of CONSTANS target genes in reproductive development of Arabidopsis[J]. Science, 2000, 288(5471):1613-1616.
[24] Corbesier L, Vincent C, Jang S, et al. FT protein movement contributes to long-distance signaling in floral induction of Arabidopsis[J]. Science, 2007, 316:1030-1033.
[25] Hepworth SR, Valverde F, Ravenscroft D, et al. Antagonistic regulation of flowering-time gene SOC1 by CONSTANS and FLC via separate promoter motifs[J]. EMBO J, 2002, 21:4327-4337.
[26] Lee H, Suh SS, Park E, et al. The AGAMOUS-LIKE 20 MADS domain protein integrates floral inductive pathways in Arabidopsis[J]. Genes Dev, 2000, 14:2366-2376.
[27] Bowman JL, Alvarez J, Weigel D, et al. Control of flower development in Arabidopsis thaliana by APETALA1 and interacting genes[J]. Development, 1993, 119:721-743.
[28] Kempin SA, Savidge B, Yanofsky MF. Molecular basis of the cauliflower phenotype in Arabidopsis[J]. Science, 1995, 267:522-525.
[29] Chang L, Zhi WNT, Yang B, et al. A conserved genetic pathway determines inflorescence architecture in Arabidopsis and rice[J]. Dev Cell, 2013, 24:612-622.
[30] Mandel MA, Gustafson-Brown C, Savidge B, et al. Molecular characterization of the Arabidopsis floral homeotic gene APETALA1[J]. Nature, 1992, 360:273-277.
[31] Ratcliffe OJ, Bradley DJ, Coen ES. Separation of shoot and floral identity in Arabidopsis[J]. Development, 1999, 126:1109-1120.
[32] Folter S, Immink RG, Kieffer M, et al. Comprehensive interaction map of the Arabidopsis MADS Box transcription factors[J]. Plant Cell, 2005, 17:1424-1433.
[33] Wellmer F, Alves-Ferreira M, Dubois A, et al. Genome-wide analysis of gene expression during early Arabidopsis flower development[J]. PLoS Genet, 2006, 2:e117.
[34] Kaufmann K, Wellmer F, Muino JM, et al. Orchestration of floral initiation by APETALA1[J]. Science, 2010, 328:85-89.
[35] Gregis V, Sessa A, Dorca-Fornell C, et al. The Arabidopsis floral meristem identity genes AP1, AGL2 and SVP directly repress class B and C floral homeotic genes[J]. Plant J, 2009, 60:626-637.
[36] Liu C, Xi W, Shen L, et al. Regulation of floral patterning by flowering time genes[J]. Dev Cell, 2009, 5:711-722.
[37] Sieburth LE, Meyerowitz EM. Molecular dissection of the AGAMOUS control region shows that cis elements for spatial regulation are located intragenically[J]. Plant Cell, 1997, 9:355-365.
[38] Bomblies K, Dagenais N, Weigel D. Redundant enhancers mediate transcriptional repression of AGAMOUS by APETALA2[J]. Dev Biol, 1999, 216:260-264.
[39] Deyholos MK, Sieburth LE. Separable whorl-specific expression and negative regulation by enhancer elements within the AGAMOUS second intron[J]. Plant Cell, 2000, 12:1799-1810.
[40] Lohmann JU, Hong R, Hobe M, et al. A molecular link between stem cell regulation and floral patterning in Arabidopsis[J]. Cell, 2001, 105:793-803.
[41] Bao X, Franks RG, Levin JZ, et al. Repression of AGAMOUS by BELLRINGER in floral and inflorescence meristems[J]. Plant Cell, 2004, 16:1478-1489.
[42] Hong RL, Hamaguchi L, Busch MA, et al. Regulatory elements of the floral homeotic gene AGAMOUS identified by phylogenetic footprinting and shadowing[J]. Plant Cell, 2003, 15:1296-1309.
[43] Riechmann JL, Meyerowitz EM. Determination of floral organ identity by Arabidopsis MADS domain homeotic proteins AP1, AP3, PI, and AG is independent of their DNA-binding specificity[J]. Mol Biol Cell, 1997, 8:1243-1259.
[44] Cho RJ, Fromont-Racine M, Wodicka L, et al. Parallel analysis of genetic selections using whole genome oligonucleotide arrays[J]. Proc Natl Acad Sci USA, 1998, 95:3752-3757.
[45] Immink RG, Tonaco IA, Shchennikova A, et al. SEPALLATA3:the‘glue’ for MADS box transcription factor complex formation[J]. Genome Biol, 2009, 10:R24.
[46] Smaczniak C, Immink RG, Muino JM, et al. Characterization of MADS-domain transcription factor complexes in Arabidopsis flower development[J]. Proc Natl Acad Sci USA, 2012, 109:1560-1565.
[47] Ditta G, Pinyopich A, Robles P, et al. The SEP4 gene of Arabidopsis thaliana functions in floral organ and meristem identity[J]. Curr Biol, 2004, 14:1935-1940.
(责任编辑 马鑫)
The Flower Development of Arabidopsis thaliana Affected by Floral Meristem Identity Gene AGL24
Jiang Wei Gu Huiying Wang Zhimin Song Ming Tang Qinglin
(Key Laboratory of Horticulture Science for Southern Mountainous Regions,Ministry of Education,Key Laboratory of Olericulture,College of Horticulture and Landscape Architecture,Southwest University,Chongqing 400715)
AGAMOUS-LIKE24(AGL24)encodes a typical MADS-domain protein that plays a very important role at different stages of flower development. This paper reviews how AGL24 interacts with other floral meristem identity genes to affect flower development of Arabidopsis thaliana, then regulate flowering time, which will help us to have a better understanding of flowering gene regulatory network and to regulate effectively the flowering time in production. It can provide reference for plant breeding.
Arabidopsis thaliana AGL24 Flower development
2013-10-08
国家自然科学基金项目(31000908),重庆市自然科学基金重点项目(2011BA1002),中央高校基本科研业务费专项(XDJK2012B020)
江为,男,硕士研究生,研究方向:蔬菜遗传育种与生物技术;E-mail:jainveborry@163.com
汤青林,男,副研究员,硕士生导师,研究方向:蔬菜遗传育种与发育调控;E-mail:swutql@163.com宋明,男,教授,研究方向:蔬菜遗传育种与生物技术;E-mail:swausongm@163.com

