秦岭细鳞鲑人工繁育群体与野生群体遗传变异分析
张艳萍 王 太 杜岩岩 虎永彪 娄忠玉 焦文龙
(甘肃省水产研究所, 甘肃省冷水性鱼类种质资源与遗传育种重点实验室, 兰州 730030)
秦岭细鳞鲑人工繁育群体与野生群体遗传变异分析
张艳萍 王 太 杜岩岩 虎永彪 娄忠玉 焦文龙
(甘肃省水产研究所, 甘肃省冷水性鱼类种质资源与遗传育种重点实验室, 兰州 730030)
研究利用线粒体DNA(细胞色素b基因序列和D-loop区序列)序列对秦岭细鳞鲑(Brachymystax lenok tsinlingensis)野生群体和人工繁育群体的种群遗传结构进行了分析。结果表明, 在86个个体扩增出的线粒体D-loop区730 bp片段中, A+T含量(63.5%)明显高于G+C含量(36.5%)。Cyt b基因序列扩增1141 bp, A+T含量(52.8%)明显高于G+C含量(47.2%)。野生群体43个个体共检测到18个单倍型, 繁育群体43个个体中共检测到 24个单倍型, 两个群体共享 8个单倍型; 秦岭细鳞鲑野生群体的单倍型多样性和核苷酸多样性(h=0.907±0.026; π=0.00287±0.00074)低于繁育群体(h=0.917±0.035; π=0.00349±0.00083), AMOVA分析显示, 98.37%的分子差异位于群体内, 1.63%的分子差异位于群体间, 两群体之间的遗传分化水平较低(Fst=0.01631, P=0.1075; Nm=30.16)。采用邻接法构建的系统发育树和单倍型网络图分析表明, 各群体内的个体不形成单系群, 两者之间互有交叉。总之, 秦岭细鳞鲑野生群体与繁育群体之间基因交流充分, 未出现遗传分化。
秦岭细鳞鲑; 线粒体D-loop区; 线粒体细胞色素b基因; 遗传多样性
细鳞鲑(Brachymystax lenok)隶属于鲑形目鲑科鲑亚科。在我国主要分布在东北地区黑龙江流域、绥芬河、图们江、鸭绿江, 新疆的额尔齐斯河, 河北省北部白河及滦河上游以及秦岭北麓渭河流域[1,2]。李思忠[3]根据幽门盲囊、侧线鳞和最小性成熟年龄等形态学和生理学特征, 把分布在秦岭地区的细鳞鲑定名为细鳞鲑的一个新亚种-秦岭亚种(Brachymystax lenok tsinlingensis Li), 为我国特有种。由于其肉质鲜美、营养价值高, 早在1938年就被人工引入到 湑水河中进行人工养殖[4,5]。近年来环境污染加剧、不当的开发和过度捕捞等原因的影响,秦岭细鳞鲑资源量急剧减少, 呈点状分布在渭河上游部分支流中。1998年秦岭细鳞鲑被收录于《中国濒危动物红皮书-鱼类》中, 属濒危物种, 国家Ⅱ级保护水生野生动物[6]。目前, 针对秦岭细鳞鲑的资源调查[7]、生物学[8—10]、胚胎发育[11]和人工繁殖[12]等方面的研究已有大量的报道, 然而对其遗传背景的研究较少, 仅见原居林等采用RAPD技术对黑河种群和 湑水河种群的遗传多样性进行了分析[13]。为了逐步恢复和保护秦岭细鳞鲑的种质资源, 对其自然种群数量进行合理的补充, 了解人工繁育群体和野生群体的遗传背景是十分重要的。
线粒体DNA (mtDNA)由于结构简单、进化速度快和几乎不发生重组等特点, 使其成为研究物种起源、系统发生和种内遗传结构的有效遗传标记[14],其中一些基因被广泛应用在鱼类的种群遗传变异和系统发育分析。王培欣等[15]利用线粒体D-loop区分析得出8个流域叉尾斗鱼种群遗传多样性很低且存在地理差异; 高天翔等[16]利用线粒体Cytb基因分析得出松江鲈8个群体的遗传多样性较高; 张迪等[17]利用线粒体COⅠ基因序列分析太湖新银鱼的遗传多样性, 得出有人工移植历史种群遗传多样性较高。本研究利用线粒体D-loop区和Cytb基因序列对秦岭细鳞鲑人工繁育群体和野生群体的遗传多样性进行比较分析, 以期为秦岭细鳞鲑放流效果的跟踪监测、种质资源遗传保护提供重要依据。
1 材料与方法
1.1 样品采集和DNA提取
收集自渭河上游支流马鹿河野生秦岭细鳞鲑为野生群体, 将收集自马鹿河和渭河另一支流西河的秦岭细鳞鲑人工驯化后, 繁殖的子一代为繁育群体,各采集43个尾鳍样品, 用无水乙醇固定带回实验室进行分析, 采用酚/氯仿法提取基因组DNA[18]。
1.2 PCR扩增和序列测定
扩增 mtDNA控制区序列的引物: 正向引物为: LRBT-25: 5′-AGAGCGCCGGTGTTGTAATC-3′; 反向引物为: LRBY-1195:5′-GCTAGCGGGACTTTC TAGGGT-3′[19]。PCR反应体系为25 μL, 其中包括1 U TaqDNA聚合酶(TaKaRa), 1 μL dNTPs (2.5 mmol/L), 5 μL 10×Taq buffer (TaKaRa, 含 Mg2+), 两条引物(10 mmol/L)各1 μL, 3 μL DNA模板, 其余双蒸水补足。PCR反应程序为: 94℃预变性3min; 94℃变性45s, 55℃退火45s, 72℃延伸45s, 共30个循环; 反应结束后在 72℃再延伸8min。PCR产物经琼脂糖凝胶电泳检测后送上海美吉生物工程公司纯化并测序, 测序引物为扩增引物。
扩增线粒体 Cytb基因序列的引物: L14724 (5′-GACTTGAAAAACCACCGTTG-3′); H15915 (5′-CTCCGATCTCCGGATTACAAGAC-3′)[20]。PCR反应体系为 25 μL, 其中包括 1 U TaqDNA聚合酶(TaKaRa), 1 μL dNTPs (2.5 mmol/L), 5 μL 10×Taq buffer (TaKaRa, 含Mg2+), 两条引物(10 mmol/L)各1 μL, 3 μL DNA模板, 其余双蒸水补足。PCR反应程序为: 94℃预变性5min; 94℃变性45s, 50℃退火45s, 72℃延伸 45s, 共 35个循环;反应结束后在72℃再延伸 10min。PCR产物经琼脂糖凝胶电泳检测后送上海美吉生物工程公司纯化并测序, 测序引物为扩增引物。
1.3 数据分析
测序获得的序列通过Chromas 1.45软件获得原始序列数据; 利用CLUSTAL X2软件对所有序列进行比对, 参照测序图进行人工校正。利用PAUP*V4.0软件进行同质性检验(Parition homogeneity test)序列片段联合分析的可靠性; 用 DnaSP 5.0软件计算单倍型数、单倍型多样性和核苷酸多样性等; 运用Arlequin 3.1软件中的分子变异(AMOVA)方法估算遗传变异在群体内和群体间的分布及遗传分化指数Fst值及其P值(用排列测验法, 1000次重排后的显著性检验), 根据 Nm=(1–Fst)/2Fst得到种群间的基因流值; 用Mega 4.0软件统计碱基组成, 并基于Kimura 2-papamter模型计算单倍型及群体间的遗传距离, 用邻接法(Neighbor-joining, NJ)构建系统进化树, 系统树中节点的自举置信水平应用自引导估计, 循环次数为1000次。Network软件构建单倍型网络图, 用以检测单倍型之间的进化关系。
2 结果
2.1 序列分析
对野生群体和繁育群体的线粒体控制区序列进行比对排序后, 得到730 bp的同源序列。43尾繁育群体所得序列共包含20个核苷酸变异位点, 其中简约信息位点15个, 单突变位点5个。所有序列间没有出现缺失与插入, 4个转换位点, 1个颠换位点, 平均转换与颠换数比值为4.3。在野生群体中, 43个个体共检测到 26个变异位点, 其中简约信息位点 21个, 单突变位点 5个, 平均转换与颠换数比值为3.3。在秦岭细鳞鲑养殖群体与野生群体中, 86个个体共检测到 27个变异位点, 其中简约信息位点 26个, 单突变位点 1个, 平均转换与颠换数比值为3.4。碱基组成分析显示, A、T、C和G碱基平均含量分别为31.9%、31.6%、20.8%和15.7%, 其中A+T含量(63.5%)明显高于 G+C含量(36.5%), 表现出明显的AT偏好和反G偏倚, 与其他脊椎动物线粒体控制区核苷酸的组成特点相一致[21]。
获得 86尾个体线粒体 Cytb基因全序列共1141 bp。所得的序列共包含25个变异位点, 其中简约信息位点24个, 单突变位点1个。A、T、C和G碱基平均含量分别为24.6%、28.2%、31.6%和15.6%,其中A+T含量(52.8%)明显高于G+C含量(47.2%)。43尾野生群体所含序列共有 23个变异位点, 其中简约信息位点16个, 单突变位点7个。43尾繁育群体所含序列共有10个变异位点, 其中简约信息位点9个, 单突变位点1个。
2.2 遗传多样性
对86尾个体的控制区片段和Cytb基因片段进行同质性检验, 结果显示两者之间是一致的(P=0.13)。基于1871 bp线粒体基因联合片段分析秦岭细鳞鲑野生群体与繁育群体的遗传多样性信息见表1, 由表1中可以看出86尾个体共检测出34个单倍型, 显示出较高的单倍型多样性和较低的核苷酸多样性(h=0.919±0.022; π=0.00321±0.00077)。繁育群体与野生群体相比较, 繁育群体的单倍型多样性和核苷酸多样性(h=0.917±0.035; π=0.00349±0.00083)都 高 于 野 生 群 体 (h=0.907±0.026; π=0.00287± 0.00074)。在秦岭细鳞鲑繁育群体43尾个体中共检测出24个单倍型, 野生群体43尾个体中共检测出18个单倍型, 其中两群体共享 8个单倍型, 占总单倍型个数的23.5%。
2.3 群体遗传分化
通过计算两群体间的遗传距离得出, 繁育群体和野生群体内的遗传距离分别为0.004和0.003,繁育群体和野生群体之间的遗传距离为 0.003, 略低于繁育群体内的遗传距离, 与野生群体的遗传距离相近。分子变异(AMOVA)结果显示(表 2), 秦岭细鳞鲑群体内的存在较高的遗传变异, 占98.37%, 而群体间的变异仅占 1.63%, 表明分子之间遗传变异主要来自于群体内的个体之间。两群体之间的 Fst=0.01631(P=0.1075), Nm=30.16, 也显示出秦岭细鳞鲑繁育群体和养殖群体之间的遗传分化不显著。

表1 秦岭细鳞鲑野生与繁育群体遗传多样性参数Tab. 1 Parameters of genetic diversity in cultured and wild Brachymystax lenok tsinlingensis populations
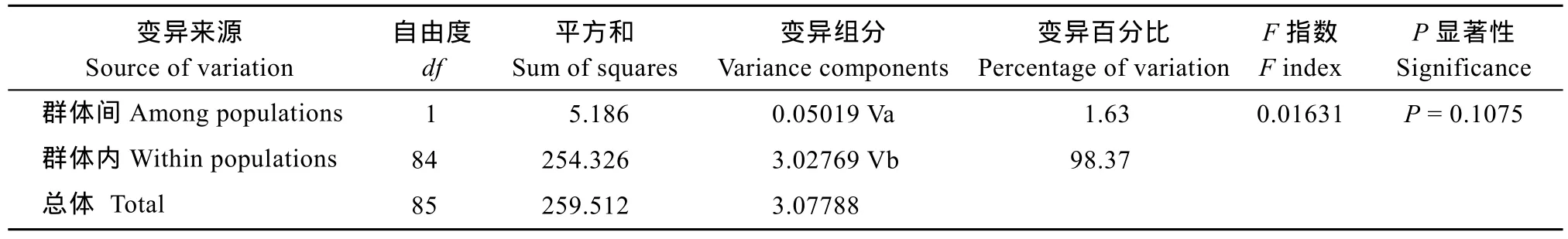
表2 秦岭细鳞鲑群体AMOVA分析结果Tab. 2 Results of AMOVA analysis of Brachymystax lenok tsinlingensis
2.4 系统发育树
由秦岭细鳞鲑繁育群体24个单倍型和野生群体18个单倍型构建的系统发育树(图1)可以看出, 各野生群体和繁育群体的个体均为形成单系群, 两者之间互有交叉, 表明两群体的遗传相似性较高, 亲缘关系较近。由 network软件形成秦岭细鳞鲑群体34个单倍型的简约网络图(图2)也显示出发散状态势,两群体未形成各自的单系群。单倍型H3位于网络图的中心, 且享有该单倍型的个体数量在种群中所占的比例明显高于其他单倍型所占比例, 表明H3可能是原始单倍型。
3 讨论
3.1 群体的遗传多样性
物种遗传多样性的高低是评判物种能否长期存在的依据, 核苷酸多样性是衡量一个种群 mtDNA的遗传多样性的重要指标, 表示各种线粒体 DNA单倍型在群体中所占的比例[22]。由实验结果可知,秦岭细鳞鲑 86尾个体的单倍型多样性(h=0.919± 0.022)较高, 但核苷酸多样性(π=0.00321±0.00077)较低, 在银鲳[23]、鲇鱼[24]和松江鲈[16]等鱼类中也存在这种现象, 这种单倍型多样性较高核苷酸多样性较低的现象表明秦岭细鳞鲑群体可能是由一个较小种群经历奠基者效应或瓶颈效应后迅速扩张, 随着个体数量的增多, 导致单倍型多样性提高, 但缺少足够的时间来积累核苷酸序列的变异[25], 这种推测与秦岭细鳞鲑的起源历程相一致[2]。但本文中得到的秦岭细鳞鲑单倍型多样性较高的结论与夏颖哲等[26]分析黄河地区细鳞鲑的单倍型多样性(0.622)的结果不符, 这可能由于夏颖哲等[26]采集样本数量(n=10)较少影响了遗传多样性的正确评估。
在本研究中繁育群体的单倍型多样性和核苷酸遗传多样性(h=0.917±0.035; π=0.00349±0.00083)都高于野生群体(h=0.907±0.026; π=0.00287±0.00074),与原居林等[13]湑对黑河种群和 水河种群的遗传多样性进行分析得出秦岭细鳞鲑繁殖群体后代遗传多样性降低的结论不相符, 这可能是由于本实验繁殖群体亲本来源分布较广, 且每年都会补充一定数量的亲本, 有效避免了近亲繁殖和瓶颈效应, 保持了秦岭细鳞鲑人工繁育群体的遗传多样性。而原居林等所采集样本为20世纪30 年代引入到 湑水河中进行驯养的群体, 在长期的人工繁殖过程中, 由于基础繁育群体数量较少、近亲繁殖和遗传漂变等因素,使得养殖群体丧失一些多态等位基因造成遗传多样性水平降低。
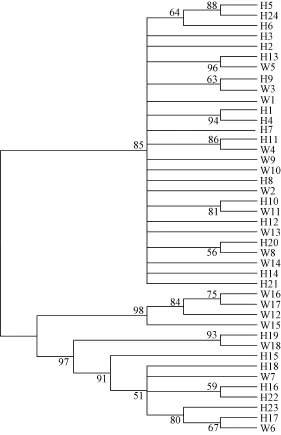
图1 基于线粒体基因联合片段构建秦岭细鳞鲑的NJ树(繁育群体H、野生群体W)Fig. 1 The neighbor-joining tree obtained by combining the control region and cytochrome b sequences (1871 bp) of Brachymystax lenok tsinlingensis population (H. Cultured population; W. Wild population)

图2 基于线粒体基因联合片段构建秦岭细鳞鲑34个单倍型的简约网络图Fig. 2 The statistical parsimony network based on the combined sequence data for the 34 haplotypes of Brachymystax lenok tsinlingensis population
3.2 群体的遗传结构分析
群体间的遗传距离和遗传分化指数是评价一个群体多态程度的重要指标。种群间遗传距离D值的范围是 0—0.05, 亚种间的遗传距离 D值范围是0.02—0.2[26]。本研究结果表明, 秦岭细鳞鲑 2个种群内的遗传距离为(0.003—0.004), 繁育群体与野生群体之间的遗传距离为 0.003, 表明秦岭细鳞鲑繁育群体与野生群体之间的遗传变异程度低, 分化程度不明显。秦岭细鳞鲑群体单倍型之间的系统发育树和网络进化图分析结果表明, 繁育群体和野生群体之间无明显的遗传分化, 两者的遗传相似程度较高。根据 Wright关于遗传分化程度的理论认为 Fst值在 0—0.05表示低度遗传分化[27], 秦岭细鳞鲑繁育群体和野生群体之间的遗传分化指数(Fst)为0.01631, 表明两群体之间存在低度遗传分化, 分子变异(AMOVA)的分析结果显示, 98.37%的遗传变异来源与群体内, 仅有 1.63%的变异来源与两群体之间。由基因流Nm值来看, 基因流是指基因从一个群体迁移至另一个群体时产生的基因流动, 一般认为Nm>1, 表明群体间的基因流水平较高, 遗传分化较小。秦岭细鳞鲑野生群体与繁育群体之间的基因流Nm值达到30.16, 远大于1, 也同样表明了两群体之间的遗传分化较小, 这可能是由于两者之间共享单倍型较多有关。秦岭细鳞鲑资源量的减少可以通过人工增殖手段来恢复, 但如果其种群遗传多样性的降低却难以弥补。目前秦岭细鳞鲑的繁育群体主要用于增殖放流, 保持其较高的种群遗传多样性具有重要意义。
[1] Qin S Z, Wang S A. Studies on the subspecies of Brachymystax lenok [J]. Salmon Fishery, 1989, 2(1): 52—61 [秦树臻, 王所安. 细鳞鱼亚种问题的研究. 鲑鳟渔业, 1989, 2(1): 52—61]
[2] Li S Z. Discussed the geographical distribution of China salmonidae fish [J]. Chinese Journal of Zoology, 1984, (1): 34—37 [李思忠. 中国鲑科鱼类地理分布的探讨. 动物学杂志, 1984, (1): 34—37]
[3] Li S Z. On a new subspecies of fresh-water trout, Brachymystax lenok tsinlingensis, from taipaishan, Shanxi, China [J]. Acta Zootaxonomica Sinica, 1966, 3(1): 92—94 [李思忠. 陕西太白山细鳞鲑的一新亚种. 动物分类学报, 1966, 3(1): 92—94]
[4] Shaanxi Institute of Zoology. Ichthyography of Qinling Mountains [M]. Beijing: Science Press. 1987, 14—15 [陕西省动物研究所. 秦岭鱼类志. 北京: 科学出版社. 1987, 14—15]
[5] Du H B, Wu J X, Wang L Z. Story behind Brachymystax lenok tsinlingensis in Xushui river valleys [J]. China Fisheries, 2006, 373(12): 79—80 [杜汉斌, 武金星, 王绿洲.湑水河流域秦岭细鳞鲑背后的故事. 中国水产, 2006, 373(12): 79—80]
[6] Wang S, Yue P Q, Chen Y Y. China Red Data Book of Endangered Animals: Pisces [M]. Beijing: Science Press. 1998, 35—37 [汪松, 乐佩琪, 陈宜瑜. 中国濒危动物红皮书(鱼类). 北京: 科学出版社. 1998, 35—37]
[7] Ren J, Liang G. Resource survey report of Brachymystax lenok tsinlingensis in Qianhe river valleys of Qinling Mountains [J]. Journal of Shaanxi Normal University (Natural Science Edition), 2004, 32(9): 165—168 [任健, 梁刚. 千河流域秦岭细鳞鲑资源调查报告. 陕西师范大学学报(自然科学版), 2004, 32(9): 165—168]
[8] Yang J Y, Zhong L, Wang K F, et al. Biological studies and protection proposes of Brachymstax lenok in Heihe River Shaanxi province [J]. Journal of Shaanxi Normal University (Nature Science Edition), 2006, 34(S1): 67—69 [杨军严, 钟林, 王开锋, 等. 陕西黑河细鳞鲑生物学研究及资源保护.陕西师范大学学报(自然科学版), 2006, 34(S1): 67—69]
[9] Wang G X, Zhou J S, Qian X M. Preliminary studies on oxygen consumption rate and suffocated point of Brachymystax lenok tsinlingensis [J]. Chinese Journal of Zoology, 2006, 41(2): 72—75 [王高学, 周继术, 强晓明.秦岭细鳞鲑耗氧率和窒息点的初步研究. 动物学杂志, 2006, 41(2): 72—75]
[10] Zheng X L, Duan M, Bu S H, et al. Analysis of the Nutritional Components of Muscle of Brachymystax lenok from Qinling Mountains [J]. Acta Nutrimenta Sinica, 2010, 32(5): 516—520 [郑雪莉, 段梅,卜书海, 等. 秦岭细鳞鲑肌肉营养成分的分析. 营养学报, 2010, 32(5): 516—520]
[11] Shi D L, Wei Q W, Sun Q L, et al. Early ontogenesis of Brachymystax lenok tsinlingensis [J]. Journal of Fishery Sciences of China, 2012, 19(4): 557—567 [施德亮, 危起伟,孙庆亮, 等.秦岭细鳞鲑早期发育观察. 中国水产科学, 2012, 19(4): 557—567]
[12] Xue C J. Technology of breeding and cultivation for Brachymystax lenok tsinlingensi [J]. China Fisheries, 2011, (10): 32—33 [薛成杰. 秦岭细鳞鲑繁育与养殖技术. 中国水产, 2011, (10): 32—33]
[13] Yuan J L, Zhu J J, Wang G X. Analysis of the genetic diversity in Heihe River and Xushuihe River populations of Qinling Brachymystax lenok [J]. South China Fisheries Science, 2009, 5(3): 63—66 [原居林, 朱俊杰, 王高学. 秦岭细鳞鲑黑河种群和 湑水河种群的遗传多样性分析. 南方水产, 2009, 5(3): 63—66]
[14] Avise J C, Arnold J, Ball R M, et al. Intraspecific phylogeography: the mitochondrial DNA Bridge between population genetics and systematics [J]. Annual Review of Ecology and Systematics, 1987, 18: 489—522
[15] Wang P X, Bai J J, Hu Y C, et al. Population genetic variations and phylogeography of Macropodus opercularis [J]. Acta Ecologica Sinica, 2011, 31(2): 441—448 [王培欣,白俊杰, 胡隐昌, 等. 叉尾斗鱼种群遗传变异与亲缘地理.生态学报, 2011, 31(2): 441—448]
[16] Gao T X, Bi X X, Zhao L L, et al. Population genetic structure of roughskin sculpin Trachidermus fasciatus based on the mitochondrial Cytb sequence [J]. Acta Hydrobiologica Sinica, 2013, 37(2): 199—207 [高天翔, 毕潇潇, 赵林林,等. 基于线粒体 Cytb 基因全序列的松江鲈群体遗传结构分析. 水生生物学报, 2013, 37(2): 199—207]
[17] Zhang D, Lei G C, Gong C, et al. Genetic diversity of Neosalanx taihuensis based on mitochondrial CO Ⅰsequences [J]. Journal of Lake Sciences, 2012, 24(2): 299—306 [张迪, 雷光春, 龚成, 等. 基于 COⅠ基因序列的太湖新银鱼遗传多样性. 湖泊科学, 2012, 24(2): 299—306]
[18] Sambrook J, Russell D W. Molecular Cloning: a Laboratory Manual [M]. Cold Spring Harbor Laboratories Press. 2001, 463—470
[19] Ma B, Jiang Z F, Huo T B. Study on the taxonomic status of species of brachymystax in Heilongjiang River and Tumen River systems based on mitochondrial control region sequence [J]. Acta Zootaxonomica Sinica, 2009, 34(3): 499—506 [马波, 姜作发, 霍堂斌. 基于线粒体DNA控制区序列变异探讨黑龙江和图们江细鳞鲑属鱼类的分类地位. 动物分类学报, 2009, 34(3): 499—506]
[20] Xiao W H, Zhang Y P, Liu H Z. Molecular systematics of Xenocyprinae (Teleostei: Cyprinidae): taxonomy, biogeography, and coevolution of a special group restricted in East Asia [J]. Molecular Phylogenetics and Evolution, 2001, 18(2): 163—173
[21] Brown G G, Gadaleta G, Pepe G, et al. Structural conservation and variation in the D-loop containing region of vertebrate mitochondrial DNA [J]. Journal of Molecular Biology, 1986, 192: 503—511
[22] Liu Y H, Zhang M H. Population genetic diversity of Roe Deer (Capreolus pygargus) in mountains of Heilongjiang province [J]. Zoological Research, 2009, 30(2): 113—120 [刘艳华,张明海. 黑龙江省不同山系狍种群遗传多样性分析. 动物学研究, 2009, 30(2): 113—120]
[23] Peng S M, Shi Z H, Hou J L. Comparative analysis on the genetic diversity of cultured and wild silver pomfret populations based on mt D-loop and CO Ⅰ gene [J].Journal of Fisheries of China, 2010, 34(1): 19—25 [彭士明, 施兆鸿,侯俊利. 基于线粒体D-Loop区与 COⅠ基因序列比较分析养殖与野生银鲳群体遗传多样性. 水产学报, 2010, 34(1): 19—25]
[24] Xiao M S, Cui F, Kang J, et al. Genetic structure and variation of wild Chinese catfish from Huaihe River based on Cytb gene sequence [J]. Chinese Journal of Zoology, 2013, 48(1): 75—86 [肖明松, 崔峰, 康健, 等. 淮河野生鲇鱼线粒体 Cytb基因的序列变异与遗传结构分析. 动物学杂志, 2013, 48(1):75—86]
[25] Bowen B W, Bass A L, Rocha L A, et al. Phylogeography of the trumpetfishes (Aulostomus): ring species complex on a global scale [J]. Evolution, 2001, 55(5): 1029—1039
[26] Nei, Masatoshi. Molecular Population Genetics and Evolution [M]. American Elsevier Pub. Co. 1975, 128—132
[27] Wright S. The gentical structure of population [J]. Annals of Eugenics, 1949, 15(1): 323—334
ANALYSIS OF THE GENETIC DIVERSITY OF CULTURED AND WILD BRACHYMYSTAX LENOK TSINLINGENSIS POPULATIONS BASED ON MTDNA D-LOOP AND CYT B
ZHANG Yan-Ping, WANG Tai, DU Yan-Yan, HU Yong-Biao, LOU Zhong-Yu and JIAO Wen-Long
(Gansu Key Laboratory of Cold Water Fishes Germplasm Resources and Genetics Breeding, Gansu Fishers Research Institute, Lanzhou 730030, China)
Brachymystax lenok tsinlingensis is one type of endemic fishes distributed in Qinling Mountains. Due to the environmental deterioration and overfishing, the size of its population has decreased sharply. To rescue this species it is necessary to study its genetic structure and phylogeography and hereby obtain better understanding of the population. In this study, we determined the genetic diversity of cultured and wild Brachymystax lenok tsinlingensis populations by analyzing their mtDNA Cyt b and D-loop genes. The results showed that the average contents of A, T, C and G in D-loop were 31.9%, 31.6%, 20.8% and 15.7%, respectively. The contents of A+T (63.5%) were higher than those of C+G (36.5%). In the Cyt b gene, the average contents of A, T, C and G were 24.6%, 28.2%, 31.6% and 15.6%, respectively. The contents of A+T (52.8%) were also higher than those of G+C (47.2%). We observed that 43 wild individuals had 18 haplotypes, and that 43 cultured individuals had 24 haplotypes. The two populations shared 8 haplotypes in common. The average haplotype diversity and nucleotide diversity of the wild population (h=0.907±0.026; π=0.00287±0.00074) were lower than those of the cultured population (h=0.917±0.035; π=0.00349±0.00083). The AMOVA analysis showed that the molecular variation between the two populations was 98.37%, and the variation within a population was 1.63%. The level of genetic differentiation was low (Fst=0.01631, P=0.1075; Nm=30.16). Phylogenetic tree based on NJ and minimum spanning network for Brachymystax lenok tsinlingensis showed that individuals within the two populations did not form a monophyletic clade. Our results suggested that there was an efficient gene flow between the cultured and the wild populations, and there was no significant genetic differentiation between the two populations.
Brachymystax lenok tsinlingensis; mtDNA D-loop; mtDNA Cyt b; Genetic diversity
Q346
A
1000-3207(2014)05-0828-06
10.7541/2014.124
2013-07-01;
2014-01-26
国家自然科学基金(31160529); 甘肃省自然科学基金(1208RJYA033)资助
张艳萍(1966—), 女, 甘肃武威人; 博士; 主要从事冷水鱼类养殖。E-mail: zypfish@sina.com
王太, E-mail: aqhongqi@qq.com

