高等植物硝酸盐转运蛋白的功能及其调控机制
贾宏昉 张洪映 刘维智 崔红 刘国顺
(河南农业大学烟草学院 国家烟草栽培生理生化基地,郑州 450002)
高等植物硝酸盐转运蛋白的功能及其调控机制
贾宏昉 张洪映 刘维智 崔红 刘国顺
(河南农业大学烟草学院 国家烟草栽培生理生化基地,郑州 450002)
硝酸盐是植物从土壤中吸收的重要无机氮素形态。植物为适应含有不同浓度NO3-的土壤环境,进化出了高亲和硝酸盐转运系统(HATS)和低亲和硝酸盐转运系统(LATS),两个基因家族NRT1和NRT2家族分别参与了LATS和HATS的NO-3的吸收和转运。近年来,随着分子生物学技术和植物基因组学的快速发展,研究人员克隆出了大量参与硝酸盐吸收和转运的基因,并对这些基因的功能进行了深入研究,逐渐形成了复杂的硝酸盐调控网络。综述了植物中硝酸盐转运蛋白基因的克隆、表达及调控,并对进一步的研究作了展望,这些结果对于理解植物硝酸盐吸收的调控机制具有重要作用。
高等植物 硝酸盐转运蛋白 调控机制 进展
氮素是植物体最重要的矿质营养元素,对植物的生长发育、产量及品质形成起着重要的作用[1]。铵态氮(NH4+)和硝态氮(NO3-)是植物从土壤中吸收的两种主要的矿质氮源。在通气良好的旱田土壤中,植物主要是以NO-为主要氮源;而在淹水的
3状态下,土壤中的氮素主要是以铵态氮(NH4+)形式存在[2]。NO3-不仅可以作为营养物质调控植物的生长,还可以作为重要的信号物质调控下游基因的转录、种子萌发、气孔活动以及植物形态发展[3,4]。到目前为止,在拟南芥、水稻、大麦等植物上已克隆多个硝酸盐转运蛋白[2,5]。人们对植物吸收NO-3的分子生物学基础已经有了相当了解,这方面的研究将有助于人们更好地理解植物是如何吸收氮素以及这种吸收过程是如何受到调控的,对今后进行作物氮高效品种改良具有重要意义。
1 植物硝酸盐转运蛋白的生理功能
植物根系从土壤中吸收NO-、植物体内NO-的
33运输和细胞内NO3-再分配都需要硝酸盐转运蛋白来执行,编码硝酸盐转运蛋白主要有NRT1和NRT2家族,分别负责低亲和(LATS)和高亲和(HATS)
硝酸盐转运系统。当外界NO3-浓度在1 μmol/L-1 mmol/L时,负责NO3-的吸收主要是高亲和转运系统(HATS);而当外界NO3-浓度高于1 mmol/L时,NO3-吸收的主要由低亲和转运系统(LATS)负责[6,7]。HATS又包括诱导型高亲和转运系统(iHATS)和组成型高亲和转运系统(cHATS);LATS又包括诱导型低亲和转运系统(iLATS)和组成型低亲和转运系统(cLATS)[5]。随着分子生物学研究技术和生物信息学的日趋成熟,很多参与硝酸盐转运的基因已经被相继分离、克隆,并对其功能做了较为深入的鉴
定(图1)[7]。
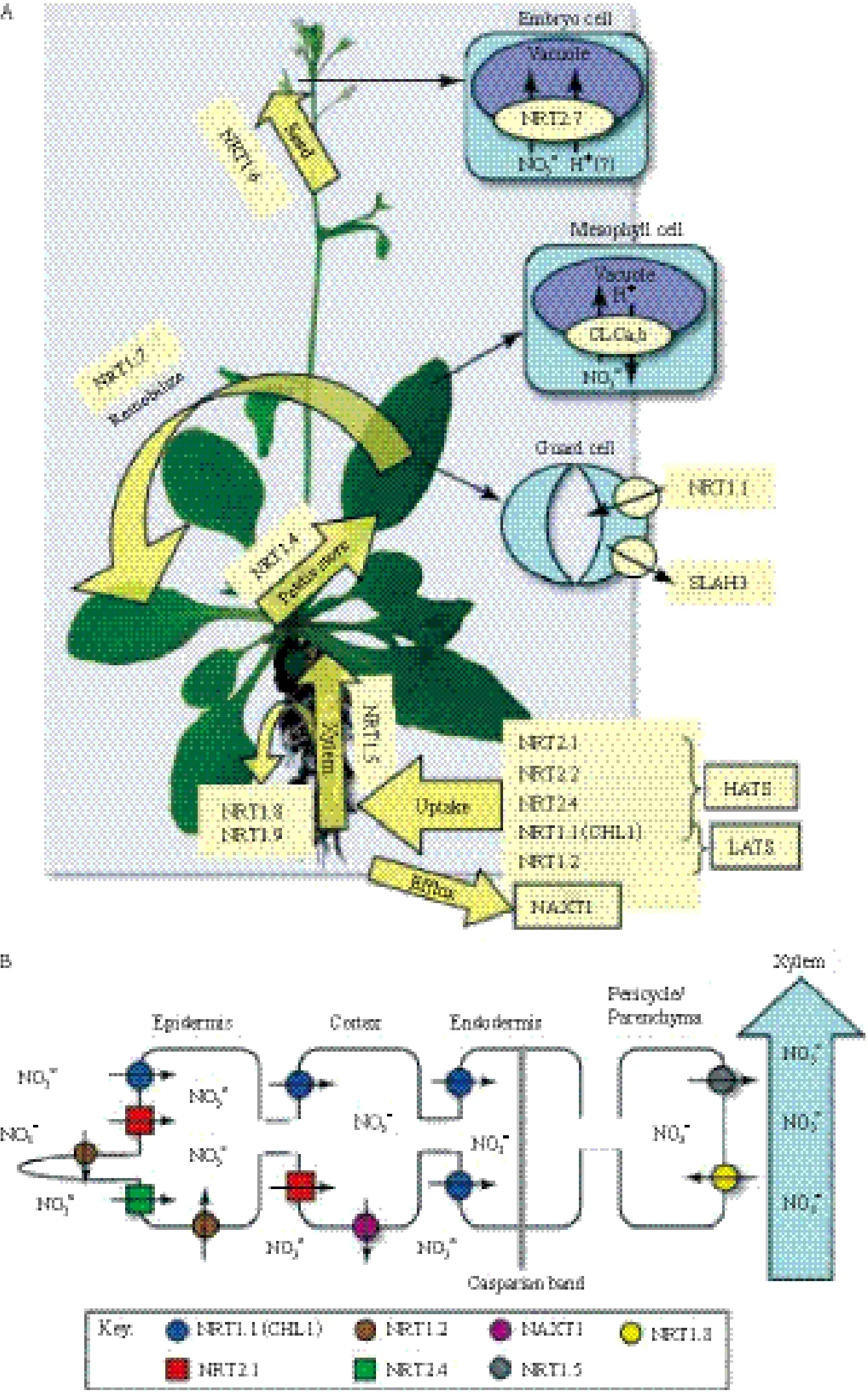
图1 拟南芥硝酸盐转运蛋白的分布[7](颜色标识见电子版)
1.1 NRT1家族转运蛋白
植物中NRT1属于小肽转运体(Peptide transporters,PTR)家族,一般含有450-600个氨基酸,有12个跨膜结构域,在NRT1的第6个和第7个跨膜域之间有一个大的亲水环状结构。双子叶植物拟南芥NRT1家族有53个成员基因,其中CHL1(AtNRT1.1)是最早被克隆的NRT1家族成员[8],随后大量拟南芥中NRT1成员基因被克隆出来。继AtNRT1.1基因以后,人们又在拟南芥中克隆到多个NRT1基因,其中AtNRT1.2是一个组成型表达基因,是拟南芥cLATs的成员[9]。该基因与AtNRT1.1有所不同,AtNRT1.2只具有低亲和力硝酸盐转运特性的转运蛋白,并不具有高亲和力硝酸盐转运特性。AtNRT1.4主要在叶柄及叶脉表达,可以调控硝酸盐在叶片的分配[10];AtNRT1.5主要在根部靠近木质部的薄壁细胞表达,主要负责将硝酸盐向木质部运输,从而调控硝酸盐由地下向地上的长距离运输[11];与AtNRT1.5不同的是AtNRT1.8在地上部及地下部靠近木质部的薄壁细胞都有表达,通过将木质部的硝酸盐向外运出调控硝酸盐在地上及地下的分配[12];AtNRT1.6主要通过调控硝酸盐从母体向胚的运输影响胚的早期发育[13];AtNRT1.7主要在叶片韧皮部的薄壁细胞表达,负责将硝酸盐向韧皮部运输从而调控衰老叶片中硝酸盐的再利用[14];而AtNRT1.9主要在根部韧皮部细胞表达,通过将硝酸盐向根系韧皮部运输调控硝酸盐的分配和再利用[15]。
到目前为止,人们已在番茄(Lycopersicon esculentum)、水稻(Oryza sativa)、烟草(Nicotiana plumbaginifolia)、油菜(Brassica napus)和苜蓿(Medicago truncatula)等植物中都克隆到了NRT1家族基因,其中水稻NRT1家族成员最多,至少有100多个[16]。大量的研究表明NRT1家族基因并不一定只是低亲和硝酸盐运输蛋白,已报道的拟南芥CHL1和苜蓿MtNRT1.3被验证是双亲和硝酸盐运输蛋白[17-19]。
1.2 NRT2家族转运蛋白
NRT2属于硝酸盐-亚硝酸盐转运体家族,该家族属于MFS(Major facilitator superfamily,MFS)超家族成员。第一个NRT2家族基因是在真核生物曲霉菌(Aspergill usnidulans)中克隆到的CrnA(NRTA)基因[20],将CrnA基因注射蟾卵异源表达系统后发现,硝酸盐的跨膜运输有2种机制:介质NO3-浓度较低时,是H+偶联的2H+/1NO3-共转运的主动转运机制,Km值为24 μmol/L[21];当介质NO3-浓度较高时采用被动吸收机制吸收NO3-,Km值为127 μmol/L[22]。但是植物的HATS似乎只是单一的2H+/1NO3-共转运系统。自从真菌中克隆到CrnA基因之后,人们随后在单细胞绿藻(Chlamydomonas reinhartii)和海洋蓝藻(Marine cyanobacterium)也分离到NRT2基因[23,24]。最早分离的植物NRT2基因是大麦基因HvNRT2.1和HvNRT2.2[25]。到目前为止,人们已在不同高等植物中分离到几十个NRT2基因。在同一种植物中,一般有多个NRT2基因,如在大麦中可能有7-10个NRT2基因,但是目前只克隆到其中的4个;在拟南芥中可能有7个NRT2基因。在水稻中已经克隆的高亲和硝酸盐运输蛋白基因有OsNRT2.1、OsNRT2.2、OsNRT2.3和OsNRT2.4及两个NAR2s(OsNAR2.1和OsNAR2.2),其中OsNRT2.3又通过选择性剪切转录成OsNRT2.3a和OsNRT2.3b两个转录本[26-28]。Tang等[28]的研究表明OsNRT2.3a在水稻硝酸盐从地下部向地上部运输过程中起关键作用,而OsNRT2.3b在水稻前期氮素积累和后期氮素转运中发挥关键作用,该基因过量表达显著提高了水稻的氮素利用效率和产量。最近本课题组利用RACEPCR方法从普通烟草(Nicotiana tabacum)中克隆出来5个高亲和硝态氮转运蛋白基因(NtNRT2.1-NtNRT2.5),其中NtNRT2.1为已报道的基因,其余4个基因均未报道,初步结果表明其余4个基因均受硝酸盐诱导增强表达。
目前已有的报道表明部分单独的高亲和磷转运蛋白并不具有NO3-转运功能,其功能的发挥依赖于NAR2蛋白的辅助。在绿藻中Zhou等[29]利用蟾卵异源表达系统证实只有同时注射CrNRT2.1和CrNAR2的mRNA的蟾卵才具有NO3-的运输活性。高等植物也具有这种双组份运输系统,Tong等[30]从大麦上克隆得到3个NAR2s类似的基因,将3个NAR2和HvNRT2.1mRNA分别注射到蟾卵时,只有HvNAR2.3和HvNRT2.1同时注射时才有硝态氮
运输活性。在植物上的研究表明NAR2和NRT2蛋白之间作用也不是特异的,Orsel等[31]通过酵母双杂交和蟾卵异源表达证明在拟南芥中AtNAR2.1与AtNRT2.1蛋白相互作用行使硝酸盐运输功能,同时他们在酵母双杂交时还发现AtNRT2s 其他成员AtNRT2.3和AtNAR2.1蛋白之间有微弱的互作。体外蛋白杂交的结果也表明HvNAR2.3和HvNRT2.1、AtNAR2.1与AtNRT2.1蛋白在根系细胞膜上存在互作[32,33]。Kotur等[34]进一步发现除了AtNRT2.7的其他AtNRT2s成员,即AtNRT2.1-AtNRT2.6蛋白都需要AtNAR2.1蛋白的协助才能定位于拟南芥原生质体膜上,在蟾卵和酵母双杂交系统AtNRT2.1-2.6蛋白和AtNAR2.1蛋白的互作也得到验证。已有的报道表明NAR2s蛋白都是小蛋白,仅有200多个氨基酸;AtNAR2.1和HvNAR2.1的蛋白结构中2个跨膜域形成一个大的环状结构域,该环状结构域是和NRT2作用的关键区域[30,32]。
1.3 植物硝酸盐运输蛋白的其他功能
研究表明硝酸盐运输蛋白基因存在着其他功能特性。在拟南芥中CHL1基因突变影响初生器官的发育,通过CHL1启动子融合GFP和GUS蛋白发现CHL1基因在叶片保卫细胞中强烈表达;进一步研究发现该基因通过改变气孔开放和蒸腾作用提高植物的抗干旱能力[35,36]。最近Li等[37]通过对atnrt1.8突变体的研究发现该基因具有调节植物对镉毒害抗性的作用。硝酸盐转运蛋白可以调控植物的根系形态建成。最先报道的是拟南芥AtNRT1.1基因,它能感知外界NO3-和Glu信号从而影响植物根系形态建成;进一步的研究表明AtNRT1.1通过诱导ANR1基因的表达来影响侧根的发生和发育[38]。除AtNRT1.1影响根系形态建成外,在低硝酸盐的外界条件下AtNRT2.1在拟南芥根系形态建成中也发挥重要作用,其作用机制一方面是通过硝酸盐吸收系统间接地影响根系形态;另一方面是在氮缺乏条件下直接影响侧根的发生[39,40]。
2 植物硝酸盐转运蛋白的表达调控机制
植物硝酸盐转运蛋白的表达水平(mRNA和蛋白)调控决定着对硝酸盐的吸收。硝酸盐转运蛋白表达受到很多因素的调控,如NO3-、NO2-、NH4+、 pH和光等,许多调控NO3-吸收的分子机制已经得到了证实(表1)。
2.1 硝酸盐转运蛋白的表达活性受NO3-调控
植物的高亲和硝酸盐转运系统能够迅速被外界的硝酸盐诱导,但是数小时后吸收速率显著下降,推测可能是由于NO-吸收后代谢产物或者内部NO-33的反馈抑制作用。在大麦、大豆、拟南芥以及水稻中,经氮素饥饿处理过的植株在恢复外界NO3-供应后,即使外界NO3-浓度只有10-50 μmol/L,植物体内NRT家族基因的mRNA丰度也会迅速升高[40]。在拟南芥中AtNRT2.1和AtNRT2.2两个基因受NO-3诱导后在转录和蛋白水平的表达都迅速提高[41],而其他一些氮素形态比如NH4+[36]、谷氨酰胺[42]都会对硝酸盐转运蛋白的表达产生一定的抑制效果。Yan等[26]也报道了在水稻中硝酸盐转运蛋白基因(OsNRT2.1、OsNRT2.2、OsNRT2.3a、OsNAR2.1)表达受NH4+和天冬氨酸的抑制。
AtNRT1.1是硝酸盐的重要感受器,能感知外界硝酸盐浓度并通过调节自身N端Thr101的磷酸化状态而对硝酸盐表现不同的亲和特征,当外界硝酸盐浓度较高时,AtNRT1.1的Thr101去磷酸化,表现低亲和特性,此时AtNRT1.1抑制AtNRT2.1和AtNAR2.1的表达;当外界硝酸盐浓度较低时,CIPK23催化AtNRT1.1的Thr101磷酸化,使其表现高亲和特性[43]。
2.2 硝酸盐转运蛋白的表达活性受pH调控
Zhou等[44]研究表明pH能够导致转运蛋白对与之结合底物的改变,在pH6.0下BnNRT1.2蛋白可以大量运输硝态氮,但在pH8.5条件下该蛋白又能大量运输组氨酸。Tong等[30]发现HvNRT2.1和HvNAR2.3的双组份高亲和硝态氮运输蛋白的活性随着细胞质酸化而减弱,推测可能是因为细胞质内的酸性pH影响了HvNRT2.1和HvNAR2.3的二聚体的正确折叠进而导致蛋白活性下降。
2.3 硝酸盐转运蛋白的表达活性受昼夜节律调节
植物NO3-吸收在白天最高夜间最低。研究发现AtNRT1.1和AtNRT2.1基因的mRNA表达结果出现一致的趋势,说明昼夜节律对NRT家族基因起调节作用。另外,在遮光下供应蔗糖也能让基因转录水
平的表达大为提高[45]。说明NRT基因受昼夜节律调节关键的影响是光合作用的产物量,而6-磷酸-葡萄糖作为信号物质调控AtNRT1.1和AtNRT2.1基因的表达。
2.4 硝酸盐转运蛋白的表达活性受NAR2蛋白的协同调控
对于某些高亲和硝酸盐转运蛋白需要其伴侣蛋白调控其活性,拟南芥(AtNRT2.1)、大麦(HvNRT2.3)以及水稻的(OsNRT2.1,OsNRT2.1,OsNRT2.3a)等转运蛋白都需要在NAR2 蛋白存在条件下才具有硝酸盐运输活性,AtNRT2.1-AtNRT2.6蛋白都需要在AtNAR2.1 蛋白辅助下共定位到细胞质膜上。因此,NAR2很可能是高亲和硝酸盐转运蛋白的调控因子。
2.5 硝酸盐转运蛋白的表达活性在蛋白水平上的调控
人们早期关于NRT2家族基因的研究表明NRT2基因的mRNA水平和硝酸盐转运蛋白活性相关性较大。最近,Laugier等[43]将35S-AtNRT2.1转入突变体atnrt2.1得到基因回补植株,研究发现黑暗条件下野生型拟南芥中AtNRT2.1 的mRNA水平和硝酸盐转运蛋白活性下降,但是回补植株的AtNRT2.1的mRNA表达水平虽然没有变化,其HATS活性也下降,蛋白检测表明是因为基因在蛋白水平表达降低了[46]。这表明硝酸盐转运蛋白的表达活性在蛋白水平上的调控更为重要。
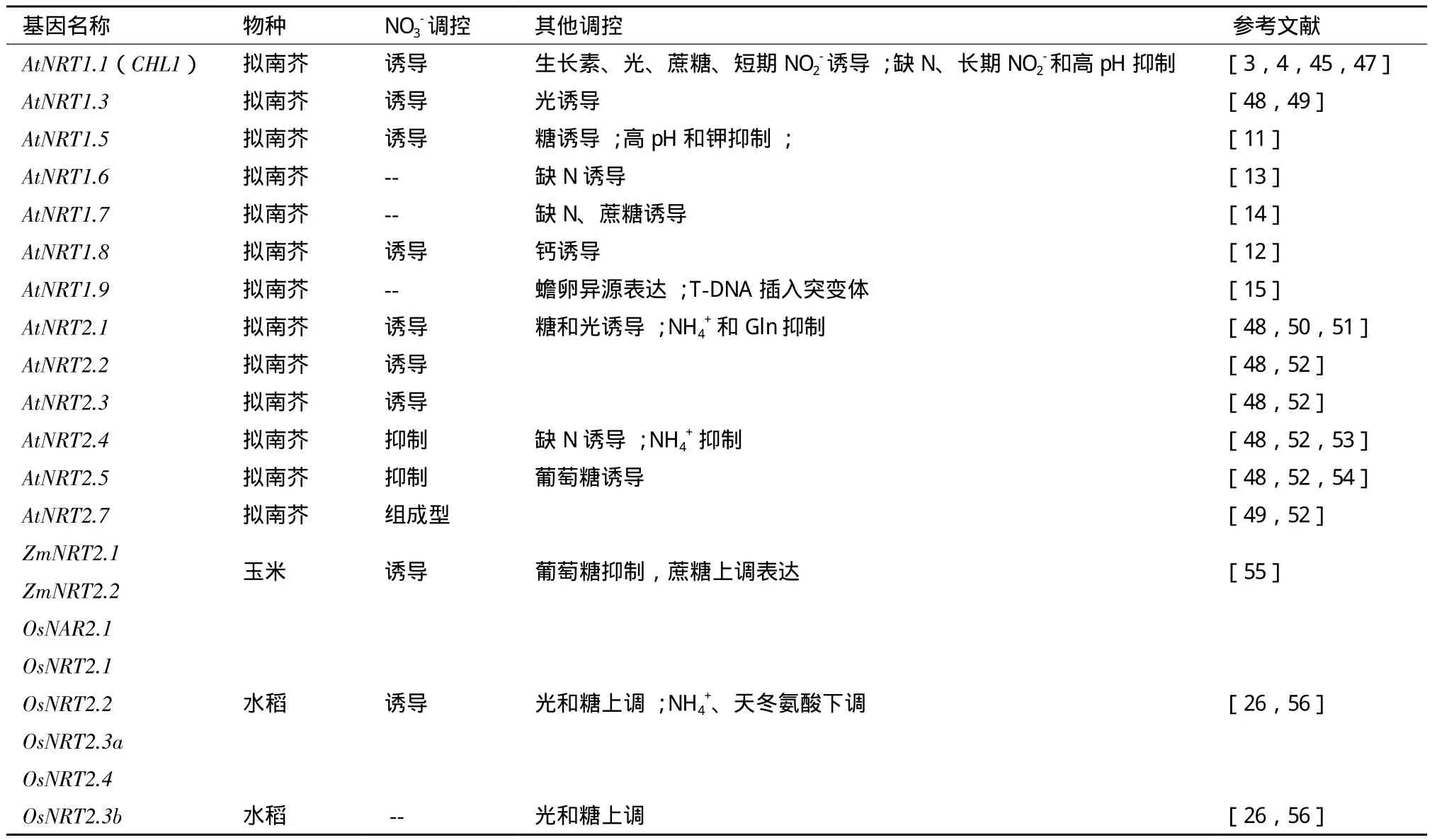
表1 植物硝酸盐转运蛋白基因受外界调控的研究进展
3 硝酸盐响应的顺式作用元件(NRE)和转录因子(TF)
植物中存在可能的响应硝酸盐的顺式作用元件NRE(Nitrate responsive cis-elements,NRE)。Rastogi等[57]在菠菜NiR基因启动子序列中找到位于-200 bp/-230 bp的GATAN24bpGATA序列是响应硝酸盐的关键元件,而且能和GATA类型的转录因子结合。另一个顺式元件‘5-(A/T)7A(C/G)TCA-3’是发现于AtNIA1、AtNIA2基因,被认为是可能的NRE,但是也没有在植物体内验证该NRE的活性。Girin等[50]发现AtNRT2.1启动子中一段150 bp的序列是NRE,并且在融合35S最小启动子启动的报告基因在转基因拟南芥中有硝酸盐响应的活性。
2010年,Konishi 等[58]报道AtNIR1基因启动子序列一段-43 bp的序列5'-GACCCTTNAAG-3'为NRE,这段序列在融合35S最小启动子和报告基因转入拟南芥后报告基因GUS具有响应硝的活性。到目前为止还很难找到在所有不同硝酸盐诱导的基因启动子序列中都保守的NRE,这可能是因为对硝酸盐响应的转录因子需要与多个NRE结合。目前在植物中还没有明确被验证的和这些NRE结合的硝响应的转录因子,仅在真菌和绿藻中发现属于GATA类型的转录因子NirA和NIT4,能够调节硝酸盐响应基因的表达[59]。
4 结语
如何提高作物氮素利用率是目前生产中急需解决的问题。作物的高产和氮素利用率密不可分,随着分子生物学技术的发展,人们越来越重视从分子水平上对作物的氮素利用效率进行遗传改良。硝酸盐转运蛋白在作物吸收和转运硝酸盐过程中起关键作用。通过调控硝酸盐转运蛋白的表达可以提高作物的氮素利用率,为通过基因工程手段提高作物氮素利用效率提供了重要的理论依据。到目前为止,虽然已从不同植物中克隆出了多个硝酸盐转运蛋白基因,并通过过量表达植物硝酸盐转运蛋白基因研究了一些硝酸盐转运蛋白的功能,但是由于这些基因通常属于一个多基因家族,单个硝酸盐转运蛋白基因所发挥的具体功能还不是特别清楚。例如,研究表明某些硝酸盐转运蛋白还具有负责其他元素的吸收及提高作物抗逆性的功能等。因此,仍然需要不断深入的研究硝酸盐转运蛋白的基因功能。
硝酸盐的吸收与利用是一个复杂的过程,除了硝酸盐转运蛋白外,还有许多硝酸盐相关的转录因子及调控基因参加这个过程。目前有关硝酸盐转录因子的研究还很少,与之相关的基因尚未被鉴定。随着对硝酸盐信号途径的研究,发现相关硝酸盐转运蛋白基因、激素、转录因子基因通过复杂的代谢网络调控植物的生长发育,但这复杂的基因调控网络目前尚未解析。因此,采用转录组测序以及功能基因组研究技术研究硝酸盐信号途径将是今后研究工作的一个重点。
[1] Singh B, Sekhon GS. Agriculture and enviroment[M]. New York:Marcel Dekker, 1990:357-398.
[2] Wang YY, Hsu PK, Tsay YF. Uptake, allocation and signaling of nitrate[J]. Trends in Plant Science, 2012, 17(8):458-467.
[3] Guo FQ, Yong J, Crawford NM. The nitrate transporter AtNRT1.1(CHL1)functions in stomatal opening and contributes to drought susceptibility inArabidopsis[J]. Plant Cell, 2003, 15(1):107-118.
[4] Ho CH, Lin SH, Hu H, et al. CHL1 functions as a nitrate sensor in plants[J]. Cell, 2009, 138(11):1184-1194.
[5] Forde BG. Nitrate transporters in plants:structure, function and regulation[J]. Biochimica Biophysica Acta, 2000, 1465(12):219-235.
[6] Williams LE, Miller AJ. Transporters responsible for the uptake and partitioning of nitrogenous solutes[J]. Annu Rev Plant Physiol Plant Mol Biol, 2001, 52(1):659-688.
[7] Wang YY, Hsu PK, Tsay YF. Uptake, allocation and signaling of nitrate[J]. Trends Plant Sci, 2012, 17(8):458-467.
[8] Liu KH, Huang CY, Tsay YF. CHL1 is a dual-affinity nitrate transporter ofArabidopsisinvolved in multiple phases of nitrate uptake[J]. Plant Cell, 1999, 11(5):865-874.
[9] Huang NC, Liu KH, Lo HJ, et al. Cloning and functional characterization of anArabidopsisnitrate transporter gene that encodes a constitutive component of low-affinity uptake[J]. Plant Cell, 1999, 11(8):1381-1392.
[10] Chiu CC, LIN CS, Hsia AP, et al. Mutation of a nitrate transporter, AtNRT1:4, results in a reduced petiole nitrate content and altered leaf development[J]. Plant Cell and Physiology, 2004, 45(9):1139-1148.
[11] Lin SH, Kuo HF, Canivenc G, et al. Mutation of theArabidopsisNRT1.5 nitrate transporter causes defective root-to-shoot nitrate transport[J]. Plant Cell, 2008, 20(9):2514-2528.
[12] Li JY, Fu YL, Pike SM, et al. TheArabidopsisnitrate transporter NRT1.8 functions in nitrate removal from the xylem sap and mediates cadmium tolerance[J]. Plant Cell, 2010, 22(3):1633-1646.
[13] Almagro A, Lin SH, Tsay YF. Characterization of theArabidopsisnitrate transporter NRT1.6 reveals a role of nitrate in early embryo development[J]. Plant Cell, 2008, 20(12):3289-3299.
[14] Fan SC, Lin CS, Hsu PK, et al. TheArabidopsisnitrate transporter NRT1.7, expressed in phloem, is responsible for source-to-sink remobilization of nitrate[J]. Plant Cell, 2009, 21(5):2750-2761.
[15] Wang YY, Tsay YF.Arabidopsisnitrate transporter NRT 1.9 is important in phloem nitrate transport[J]. Plant Cell, 2011, 23(5):1945-1957.
[16] Araki R, Hasegawa H. Expression of rice(Oryza sativaL.)genes involved in high-affinity nitrate transport during the period of nitrate induction[J]. Breeding Science, 2006, 56(1):295-302.
[17] Liu KH, Huang CY, Tsay YF. CHL1 is a dual-affinity nitrate transporter ofArabidopsisinvolved in multiple phases of nitrate uptake[J]. Plant Cell, 1999, 11(5):865-874.
[18] Liu KH, Tsay YF. Switching between the two action modes of the dual-affinity nitrate transporter CHL1 by phosphorylation[J]. EMBO J, 2003, 22(5):1005-1013.
[19] Morère-Le, Paven MC, Viau L, Hamon A, et al. Characterization of a dual-affinity nitrate transporter MtNRT1.3 in the model legumeMedicago truncatula[J]. J Exp Bot, 2011, 62(15):5595-5605.
[20] Unkles SE, Hawker KL, Grieve C, et al. CrnA encodes a nitrate transporter inAspergillus nidulans[J]. Proc Natl Acad Sci USA, 1991, 88(1):204-208.
[21] McClure PE, Kochian LV, Spanswick RM, et al. Evidence for cotransport of nitrate and protons in maize roots[J]. Plant Physiology, 1990, 93(1):281-289.
[22] Zhou JJ, Trueman LJ, Boorer KJ, et al. A high-affinity fungal nitrate carrier with two transport mechanisms[J]. J Biol Chem, 2000, 275(51):39894-39899.
[23] Quesada A, Galvan A, Fernandez E. Identification of nitrate transporter genes inChlamydomonas reinhardtii[J]. Plant Journal, 1994, 5(3):407-419.
[24] Sakamoto T, Inoue SK, Bryant DA. A novel nitrate/nitrite permease in themarine Cyanobacterium synechococcussp. strain PCC7002[J]. Journal Bacteriol, 1999, 181(23):7363-7372.
[25] Trueman LJ, Richardson A, Forde BG. Molecular cloning of higher plant homologues of the high-affinity nitrate transporters ofChlamydomonas reinhardtii[J]. Gene, 1996, 175(1-2):223-231.
[26] Yan M, Fan XR, Feng HM, et al. Rice OsNAR2.1 interacts with OsNRT2.1, OsNRT2.2 and OsNRT2.3a nitrate transporters to provide uptake over high and low concentration ranges[J]. Plant Cell and Environment, 2011, 34(8):1360-1372.
[27] Xu G, Fan X, Miller AJ. Plant nitrogen assimilation and use efficiency[J]. Annu Rev Plant Biol, 2012, 63(1):153-82.
[28] Tang Z, Fan XR, Li Q, et al. Knock down of a rice stellar nitrate transporter alters long distance translocation but not root influx[J]. Plant Physiology, 2012, 160(4):2052-2063.
[29] Zhou JJ, Fernandez E, Galvan A, et al. A high affinity nitrate transport system fromChlamydomonasrequires two gene products[J]. FEBS Letter, 2000, 466(2-3):225-227.
[30] Tong YP, Zhou JJ, Li ZS, et al. A two-component high-affinity nitrate uptake system in barley[J]. Plant J, 2005, 41(3):442-450.
[31] Orsel M, Chopin F, Leleu O, et al. Characterization of a twocomponent high-affinity nitrate uptake system inArabidopsis. Physiology and protein-protein interaction[J]. Plant Physiology, 2006, 142(3):1304-1317.
[32] Ishikawa S, Ito Y, Sato Y, et al. Two-component high-affinity nitrate transport system in barley:membrane localization, protein expression in roots and a direct protein-protein interaction[J]. Plant Biotech, 2009, 26(2):197-205.
[33] Yong Z, Kotur Z, Glass ADM. Two-component high-affinity nitrate transport system in barley:membrane localization, protein expression in roots and a direct protein-protein interaction[J]. Plant Journal, 2010, 63(2):739-748.
[34] Kotur Z, Mackenzie N, Ramesh S, et al. Nitrate transport capacity of theArabidopsis thalianaNRT2 family members and their interactions with AtNAR2.1[J]. New Phytologist, 2012, 194(3):724-31.
[35] Guo FQ, Wang RC, Chen MS, et al. TheArabidopsisdual-affinity nitrate transporter gene AtNRT1.1(CHL1)is activated and functions in nascent organ development during vegetative and reproductive growth[J]. Plant Cell, 2001, 13(8):1761-1778.[36] Muños S, Cazettes C, Fizames C, et al. Transcript profiling in thechl1-5mutant ofArabidopsisreveals a role of the nitrate transporter NRT1.1 in the regulation of another nitrate transporter, NRT2.1[J]. Plant Cell, 2004, 16(9):2433-2447.
[37] Li JY, Fu YL, Pike SM, et al. TheArabidopsisnitrate transporter NRT1.8 functions in nitrate removal from the xylem sap and mediates cadmium tolerance[J]. Plant Cell, 2010, 22(5):
1633-1646.
[38] Zhang HM, Forde BG. AnArabidopsisMADS box gene that controls nutrient-induced changes in root architecture[J]. Science, 1998, 279(5349):407-409.
[39] Little DY, Rao H, Oliva S, et al. The putative high-affinity nitate transporter NRT2.1 represses lateral root initiation in response to nutritional cues[J]. Proc Natl Acad Sci USA, 2005, 102(38):13693-13698.
[40] Remans T, Nacry P, Pervent M, et al. A central role for the nitrate transporter NRT2.1 in the integrated morphological and physiological responses of the root system to nitrogen limitation inArabidopsis[J]. Plant Physiology, 2006, 140(3):909-921.
[41] Filleur S, Daniel VF. Expression analysis of a high-affinity nitrate transporter isolated fromArabidopsis thalianaby differential display[J]. Planta, 1999, 207(3):461-469.
[42] Vidmar JJ, Zhuo D, Siddiqi MY, et al. Regulation of high-affinity nitrate transporter genes and high-affinity nitrate influx by nitrogen pools in roots of barley[J]. Plant Physiology, 2000, 123(1):307-318.
[43] Laugier E, Bouguyon E, Mauries A, et al. Regulation of high-affinity nitrate uptake in roots ofArabidopsisdepends predominantly on posttranscriptional control of the NRT2.1/NAR2.1 transport system[J]. Plant Physiology, 2012, 158(2):1067-1078.
[44] Zhou JJ, Theodoulou FL, Muldin I, et al. Cloning and functional characterization of aBrassica napustransporter which is able to transport nitrate and histidine[J]. Journal of Biological Chemistry, 1998, 273(20):12017-12023.
[45] Lejay L, Tillard P, Lepetit M, et al. Molecular and functional regulation of two NO3-uptake systems by N and C status ofArabidopsisplants[J]. Plant Journal, 1999, 18(5):509-519.
[46] Liu KH, Tsay YF. Switching between the two actions modes of the dual-affinity nitrate transporter CHL1 by phosphorylation[J]. EMBO Journal, 2003, 22(5):1005-1013.
[47] Huang NC, Chiang CS, Crawford, et al. CHL1 encodes a component of the low-affinity nitrate uptake system inArabidopsisand shows cell type-specific expression in roots[J]. Plant Cell, 1996, 8(12):2183-2191.
[48] Lejay L, Wirth J, Pervent M, et al. Oxidative pentose phosphate pathway-dependent sugar sensing as a mechanism for regulation of root ion transporters by photosynthesis[J]. Plant Physiology, 2008, 146(4):2036-2053.
[49] Okamoto M, Vidmar JJ, Glass ADM. Regulation of NRT1 and NRT2 gene families ofArabidopsis thaliana:Responses to nitrate provision[J]. Plant Cell Physiology, 2003, 44(3):304-317.
[50] Girin T, Lejay L, Wirth J, et al. Identification of a 150 bp cis-acting element of the AtNRT2.1 promoter involved in the regulation of gene expression by the N and C status of the plant[J]. Plant Cell and Environment, 2007, 30(11):1366-1380.
[51] Wirth J, Chopin F, Santoni V, et al. Regulation of root nitrate uptake at the NRT2.1 protein level inArabidopsis thaliana[J]. J Biol Chem, 2007, 282(1):23541-23552.
[52] Orsel M, Krapp A, Daniel-Vedele F. Analysis of the NRT2 nitrate transporter family inArabidopsis. Structure and gene expression[J]. Plant Physiology, 2002, 129(2):886-896.
[53] Kiba T, Feria-Bourrellier A, Lafouge F, et al. TheArabidopsisnitrate transporter NRT2.4 plays a double role in roots and shoots of nitrogen-starved plants[J]. Plant Cell, 2012, 24(1):245-258.
[54] Pourtau N, Jennings R, Pelzer E, et al. Effect of sugar-induced senescence on gene expression and implications for the regulation of senescence inArabidopsis[J]. Planta, 2006, 224(3):556-568.
[55] Trevisan S, Borsa P, Botton A, et al. Expression of two maize putative nitrate transporters in response to nitrate and sugar availability[J]. Plant Biology, 2008, 10(4):462-475.
[56] Feng HM, Yan M, Fan XR, et al. Spatial expression and regulation of rice high-affinity nitrate transporters by nitrogen and carbon status[J]. J Exp Bot, 2011, 62(7):2319-2332.
[57] Rastogi R, Bate NJ, Sivasankar S, et al. Foot printing of the spinach nitrite reductase gene promoter reveals the preservation of nitrate regulatory elements betweenfungiand higher plants[J]. Plant Molecular Biology, 1997, 34(3):465-476.
[58] Konishi M, Yanagisawa S. Identification of a nitrate-responsive ciselement in theArabidopsisNIR1 promoter defines the presence of multiple cis-regulatory elements for nitrogen response[J]. Plant Journal, 2010, 63(2):269-282.
[59] Marzluf GA. Genetic regulation of nitrogen metabolism in thefungi[J]. Microbiology and Molecular Biology Reviews, 1997, 61(1):17-32.
(责任编辑 狄艳红)
Function and Regulation Mechanisms of Nitrate Transporters in Higher Plants
Jia Hongfang Zhang Hongying Liu Weizhi Cui Hong Liu Guoshun
(College of Tobacco Science,Henan Agricultural University;National Tobacco Cultivation,Physiology and Biochemistry Research Center,Zhengzhou 450002)
Nitrate(NO3-)is main nitrogen sources which plants absorb from soil. There are two different uptake systems in plants to cope with low or high NO3-concentrations in soil, the high affinity systems(HATS)and low affinity NO3-uptake systems(LATS). It is suggested that NRT1 and NRT2 families contribute to LATS and HATS for both NO3-uptake and distribution within the plant. Specific transport systems are essential for the uptake of NO3-and for its internal redistribution within plant. Over the past years, a significant advance has been obtained on the gene cloning, expression regulation and functional characterization of NRT1/2 transporters in higher plants. In this article, the research progresses on molecular level of known Nitrate transporters in higher plant in last decade were reviewed.
Higher plants Nitrate transporters Regulation mechanism Recent advances
2013-11-03
国家自然科学基金项目(31301837),中国烟草总公司特色优质烟叶开发重大专项浓香型项目(110201101001TS-01)
贾宏昉,男,博士,讲师,研究方向:植物营养分子遗传;E-mail:jiahongfang@126.com
刘国顺,男,教授,博士生导师,研究方向:烟草生理生化;E-mail:liugsh1851@163.corn

