长江流域片国家区试大豆品种分子ID构建及遗传多样性分析
何琳,何艳琴,刘业丽,杨中路,栾怀海,韩雪,蒋洪蔚,刘春燕,胡国华
(1.黑龙江省农垦科研育种中心,哈尔滨 150090;2.全国农业技术推广服务中心;3.中国农业科学院油料作物研究所)
大豆在人们生活中占有重要的地位,是我国四大油料作物之一,也是蛋白质的重要来源[1]。我国长江流域地区大豆栽培历史悠久,由于自然地理、气候条件及轮作复种制度复杂多样,形成了丰富的大豆种质资源[2-3],但对参加长江流域地区国家区域试验大豆品种的纯度、分子ID及遗传多样性分析研究报道较少,不利于大豆种质资源的保存利用、大豆新品种的审定、保护和选育等研究工作的开展。
SSR分子标记随机、均匀、广泛地分布于大豆整个基因组中,且符合作物品种鉴别的4个基本准则:环境的稳定性、品种间变异的可识别性、最小的品种内变异和实验结果的可靠性[4]。高运来等[5]应用SSR标记构建黑龙江13个育种单位6个积温带的83份大豆品种的分子ID,很多科研工作者对黄淮夏大豆[6]、河北[4]、湖北[7]、吉林[8]、山东[9]和黑龙江[10]等多个地区的大豆品种进行了遗传分析,说明SSR标记已广泛应用于大豆品种鉴别、遗传多样性分析等方面。
研究用SSR分子标记技术对长江流域片国家区域试验大豆品种进行分子ID的建立及品种间的遗传多样性分析,能快速准确鉴定参试大豆品种,了解该地区参试大豆品种的遗传背景,指导大豆品种的选育工作,对长江流域地区大豆种子质量标准化、审定大豆品种和种质资源遗传评价等具有重要意义。
1 材料与方法
1.1 试验材料
研究所用材料为参加2012年长江流域片国家区域试验大豆品种45份(见表1)。由中国农业科学院油料作物研究所提供,每个大豆品种取50粒种子,于2012年在黑龙江省农垦科研育种中心实验室进行试验。
1.2 试验方法
1.2.1 DNA提取与SSR分析
参照中华人民共和国农业行业标准大豆品种纯度鉴定技术规程SSR分子标记法[11]。
1.2.2 SSR引物
根据大豆公共遗传图谱,对20个连锁群上均匀分布的100对引物进行筛选,选择了8个连锁群上扩增稳定、多态性高的13对SSR引物用于PCR扩增,根据SoyBase提供的SSR引物序列(见表2),上海生物工程有限公司合成。

表2 用于PCR扩增的13对引物详情Table 2 Primers details of PCR amplification
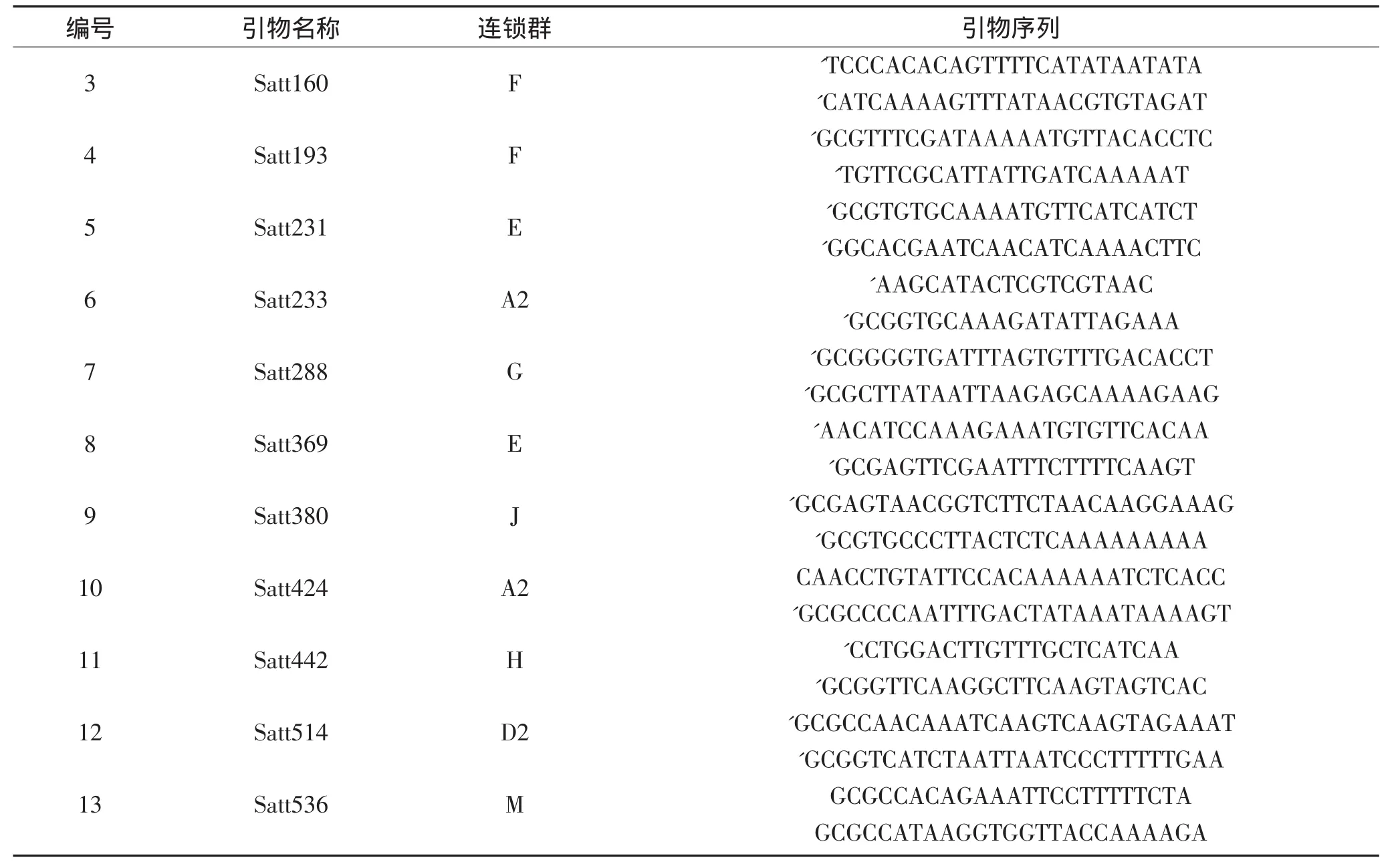
续表2 用于PCR扩增的13对引物详情Continued table 2 Primers details of PCR amplification
1.2.3 构建大豆参试品种分子ID的数据分析
根据扩增片段的大小,对应的分子量从大到小从阿拉伯数字的1开始记录,依次为2、3、4……N统计数据。0表示零等位基因(即该泳道由于基因片段丢失而无带),-1表示该品种数据由于实验操作造成缺失,-2表示该泳道出现杂合带型。采用黑龙江省农垦科研育种中心自行开发的资源特征分析软件(IDAnalysis 1.0,软件登记号:2007SR11870)构建大豆品种分子ID。将统计数据转换为文本文档后,可直接用该软件进行分析,其实现结果的过程为淘汰缺失位点过多和引物之间相似系数过高的引物后以最少的引物来区分品种[6]。
1.2.4 大豆参试品种遗传多样性的数据分析
按照Nei和Li(1979)的公式:GS=2Nxy/(Nx+Ny)计算品种间的遗传相似系数。式中,GS为品种间的遗传相似系数,Nxy为2个品种共有的等位基因,Nx和Ny分别为品种x和y的等位基因数。遗传距离(GD)= 1-GS[11]。采用黑龙江省农垦科研育种中心自行开发的应用遗传学统计软件(Genetics Statistics 3.0,软件登记号为2007SR11872)分析大豆参试品种间的相似系数。
2 结果与分析
2.1 大豆参试品种分子ID的建立
品种分子ID分为两类,一类是利用引物的特异等位基因区分品种;由于含有特异等位基因的品种较少,很多品种只能用另一类即用多个引物的等位基因组合来区分品种。图1为引物Satt536对参试大豆品种扩增的电泳结果,共有4个等位基因。

图1 引物Satt536等位基因特征Fig.1 Alleles feature of primer Satt536
利用13个SSR引物中的6个引物:Satt138、Satt514、Satt231、Satt380、Satt442和Satt369就可以将全部45个大豆品种区分,并获得唯一的分子ID。其中引物Satt138在材料31中显示特异条带1;引物Satt138在材料39中显示特异条带7;引物Satt138在材料2中显示特异条带12;引物Satt138在材料20中显示特异条带13;引物Satt231在材料18中显示特异条带1;引物Satt231在材料30中显示特异条带4;引物Satt380在材料10中显示特异条带1;引物 Satt442在材料 31中显示特异条带 6;引物Satt514在材料10中显示特异条带4。
依据引物顺序Satt138、Satt514、Satt231、Satt380、Satt442和Satt369,建立了45份大豆品种的分子ID(表3)。
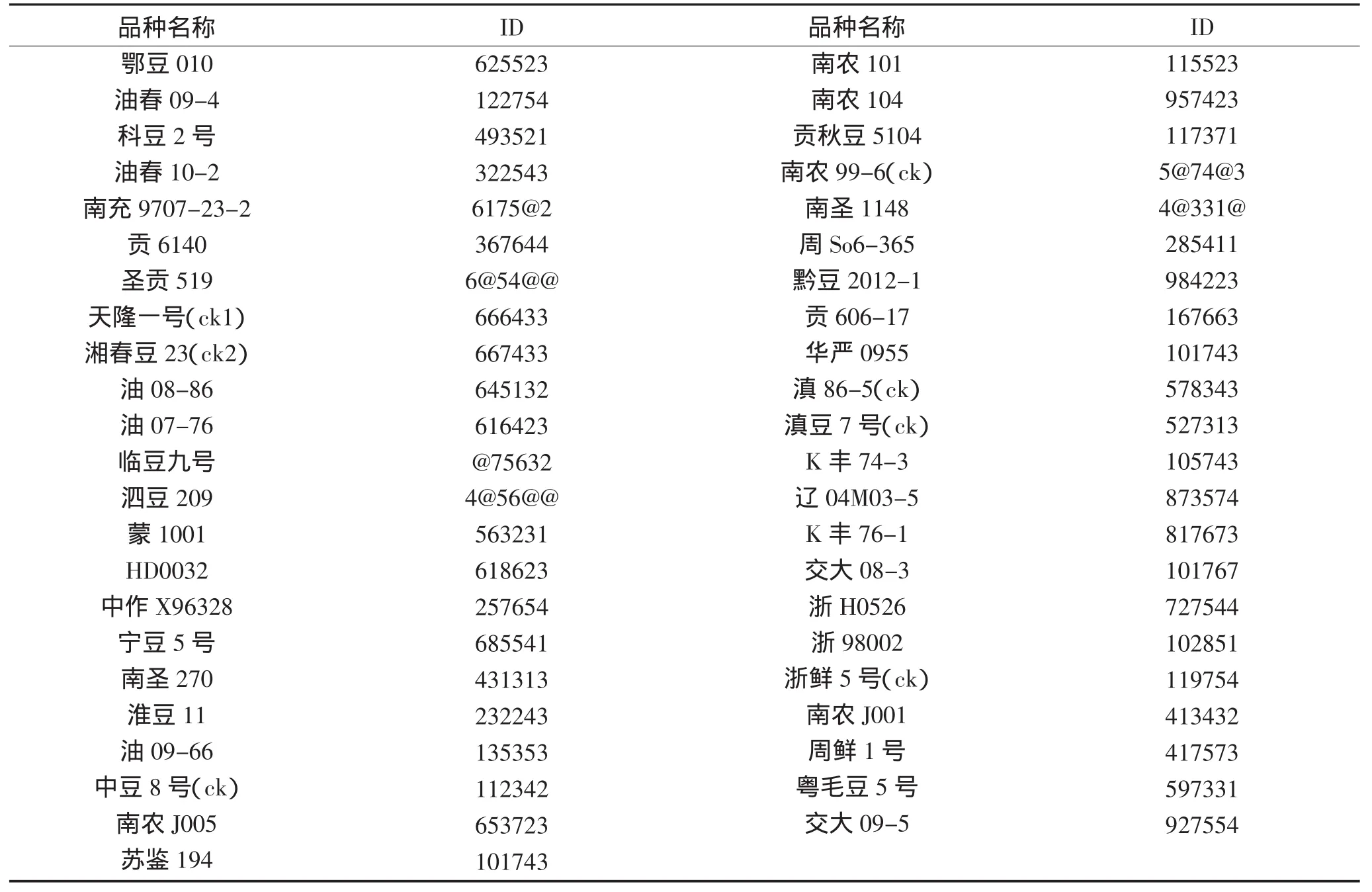
表3 45份大豆参试品种的分子IDTable 3 Molecule ID of 45 tested soybean varieties
2.2 大豆参试品种遗传关系分析
利用应用遗传学统计软件Genetics Statistics V3.0计算45个大豆参试品种间的遗传相似系数,发现这45个大豆品种间遗传相似系数介于0~1.00,平均相似系数为0.442 4。结果表明,长江流域片2012年国家区试大豆品种间遗传同源性较小,差异性较大。在45个大豆参试品种中有10组(12份)品种间的相似性高于80%(表4),其中第10组材料临豆九号和泗豆209品种间的遗传关系最近,遗传相似系数为1,需要继续增加引物加以区分。

表4 遗传相似性高于80%的大豆品种及遗传距离Table 4 Soybean varieties and genetic distance above 80%

续表4 遗传相似性高于80%的大豆品种及遗传距离Continued table 4 Soybean varieties and genetic distance above 80%
3 结论与讨论
分子ID(即分子身份证)是把品种的特征数字化,即每个品种都有属于自己的一套字符串形式的代码,从而可以简单明了的区分品种。高运来等[5]利用43对SSR引物对黑龙江省6个积温带的83个大豆品种构建了分子身份证;李伟忠等[12]利用64对SSR引物对257份玉米自交系建立了分子身份证;辜大川[13]和陆徐忠[14]分别用SSR标记构建了水稻品种的分子身份证,说明利用SSR标记构建作物品种的分子身份证有较强的可靠性,可作为区分品种的一个有效的方法。试验中仅用13对SSR引物中的6对SSR引物就将45个参试大豆品种区分开,说明利用SSR标记区分鉴定参试大豆品种是非常可行的,也为大豆品种审定和申请品种保护提供理论依据。
研究应用遗传学统计软件Genetics Statistics V3.0对长江流域片国家区试大豆品种的遗传多样性分析发现,长江流域地区国家区试大豆品种间遗传背景差异较高。在45份参试大豆品种中有12个大豆品种彼此的遗传相似性高于80%,可能是由于长江流域地区各个省份自然条件比较相近,相互间的异地引种比较频繁使得这12个参试大豆品种遗传同源性较大。SSR标记分析大豆品种的遗传多样性可直接反映品种间在基因型上的差异,了解品种间亲缘关系远近,从而可以快速准确合理地选择亲本材料,以指导配制杂交组合,进而有效的指导长江流域地区大豆的育种和生产工作。
[1] 孙冰.大豆发芽过程中营养及功能性物质检测[D].大庆:黑龙江八一农垦大学,2013.
[2] 周蓉,张小娟,王贤智,等.湖北省大豆种质资源的遗传多样性分析[J].大豆科学,2005,25(3):212-217.
[3] 费贵华,盖钧镒,马育华.华南四省区大豆地方品种群体遗传特点的研究[J].南京农业大学学报,1994,7(2):1-6.
[4] 贾晓艳.河北省大豆推广品种DNA指纹图谱构建及遗传多样性评价[D].保定:河北农业大学,2006.
[5] 高运来,朱荣胜,刘春燕,等.黑龙江部分大豆品种分子ID的构建[J].作物学报,2009,35(2):211-218.
[6] 崔艳华,邱丽娟,常汝镇,等.黄淮夏大豆遗传多样性分析[J].中国农业科学,2004,37(1):215-224.
[7] 周蓉,王贤智,张小娟,等.湖北夏大豆种质SSR标记的遗传多样性研究[J].武汉植物学研究,2007,25(6):544-549.
[8] 张逸鸣,李英慧,郑桂萍,等.吉林省大豆育成品种的遗传多样性特点分析[J].植物遗传资源学报,2007,8(4):456-463.
[9] 韩雪芹,林延慧,张礼凤,等.山东省不同年代栽培大豆SSR标记遗传多样性分析[J].中国农学通报,2008,24(1):74-77.
[10] 高运来,姚丙辰,刘春燕,等.黑龙江省主栽大豆品种遗传多样性的SSR分析[J].植物学报,2009,44(5):556-561.
[11] NY/T 17888-2009.大豆品种纯度鉴定技术规程SSR分子标记法[S].北京:中华人民共和国农业部,2009.
[12] 李伟忠,许崇香,安英辉,等.257份玉米自交系分子ID的构建[J].玉米科学,2013,21(2):24-30.
[13] 辜大川,邱东峰,殷明珠,等.水稻优异资源分子身份证的建立[J].湖北农业科学,2012,51(24):5579-5583.
[14] 陆徐忠,从夕汉,刘海珍,等.杂交水稻亲本分子身份证及SSR指纹数据库的建立[J].核农学报,2012,26(6):853-861.

