反向杂交检测肝癌相关乙型肝炎病毒前C区突变方法的建立和应用
赵芝梅 朱宇 吴燕 樊春笋 陈陶阳 甘愉 屠红
1.上海交通大学医学院附属仁济医院上海市肿瘤研究所癌基因及相关基因国家重点实验室,上海200032;2.启东肝癌防治研究所,启东肿瘤医院病因室,江苏 启东 226200
反向杂交检测肝癌相关乙型肝炎病毒前C区突变方法的建立和应用
赵芝梅1 朱宇1 吴燕1 樊春笋1 陈陶阳2 甘愉1 屠红1
1.上海交通大学医学院附属仁济医院上海市肿瘤研究所癌基因及相关基因国家重点实验室,上海200032;2.启东肝癌防治研究所,启东肿瘤医院病因室,江苏 启东 226200
背景与目的:日益增多的研究表明,乙型肝炎病毒(hepatitis B virus, HBV)DNA前C区G1896A和G1899A突变是肝癌发生的危险因素。本研究旨在建立简单、快速、灵敏和准确的检测HBV前C区突变的反向杂交方法,并应用于检测江苏省启东地区HBV DNA前C区突变与肝癌发生的关系。方法:设计并合成HBV DNA前C区1896和1899位点的特异性探针,通过优化条件建立特异、敏感的杂交体系,并与直接测序检测结果进行比较。将该方法应用于检测启东100例肝癌和100例慢性HBV携带者(对照组),分析HBV DNA前C区突变与肝癌的关系。结果:反向杂交对血清样本的最低检测下限为HBV DNA 103copy/mL,检测混合感染时比直接测序更占优势,混合株中10%以上的突变株均可被检测。启东地区HBV DNA前C区G1899A突变与肝癌高发具有相关性(P=0.000, OR=4.846, 95%CI:2.240~10.485),而G1896A突变未见其相关性。结论:反向杂交检测HBV DNA前C区突变方便、快速、准确,可有效监控肝癌的发生,适合临床大规模推广应用。
乙型肝炎病毒;前C区;反向杂交;突变;肝癌
乙型肝炎病毒(hepatitis B virus,HBV)慢性感染是导致我国肝癌发生的重要原因[1]。HBV DNA有4个开放读码区(open reading frame,ORF):前S/S区、前C/C区、X区和P区。目前已有多项研究证实,HBV DNA前C区G1896A和G1899A突变均能增加肝癌发病的风险[2-7]。荟萃分析显示,HBV DNA前C区G1896A和G1899A突变导致肝癌发生风险分别提高1.46和3.13倍[8]。因此建立—种简单、灵敏、准确的HBV前C区突变检测方法具有重大意义。目前已有各种HBV DNA前C区突变的检测方法,如聚合酶链反应(polymerase chain reaction,PCR)产物直接测序[2-7]、限制性片段长度多态性分析[9]、荧光定量PCR[10-11]和基因芯片[12]等,其中DNA测序是序列分析的金标准,但对混合感染的检出率低。反向杂交技术是一种建立于PCR基础上的方法,已应用于结核杆菌和地中海贫血致病基因的检测等[13-14]。本研究通过建立检测HBV DNA前C区G1896A和G1899A突变的反向杂交技术,为肝癌预警提供适合大规模临床推广的突变检测方法。
1 资料和方法
1.1 研究对象
性别和年龄相匹配的100例肝癌和100例慢性HBV携带者(慢性肝炎,对照组)均为2007年10月—2012年10月期间江苏省启东市人民医院收治的临床患者。所有患者血清均置于-70 ℃冰箱冻存。肝癌的诊断符合2001年中国抗癌协会肝癌专业委员会修订的《原发性肝癌的临床诊断与分期标准》[15]。患者均签署知情同意书,本研究获得启东人民医院及上海市肿瘤研究所伦理委员会批准。
1.2 方法
1.2.1 HBV标准质粒构建
根据测序结果为G1896/G1899、1896A、1899A和1896A/1899A的患者血清样本用Q5TM超保真DNA聚合酶(New England Biolabs公司)分别进行HBV DNA前C区PCR扩增,将PCR产物克隆在平端载体pEASY-Blunt Zero(北京TransGens公司)上,并通过质粒抽提(美国Omega公司),测序筛选,构建HBV野生型和3种突变型标准质粒。
1.2.2 引物和探针
通过对比GenBank数据库中所有人HBV全基因组序列,选择保守区设计引物和扩增质控探针,上游引物5’端生物素标记。根据检测位点特异性设计探针: 1896和1899位点的野生型探针(G1896/G1899)、1896位点的突变型探针(1896A)、1899位点的突变型探针(1899A)、1896和1899位点的突变型探针(1896A/1899A)、扩增质控探针(amplification control)和显色质控探针(conjugate control)。扩增质控探针为HBV基因组一段保守序列,能与任何PCR阳性产物结合,若该探针未显色,则说明HBV DNA阴性或者HBV DNA含量低于本方法的检测下限。显色质控探针3’端生物素标记,不与目的产物结合,只与显色试剂结合,质控显色过程。引物与探针序列见表1,由上海英骏生物技术有限公司合成。
1.2.3 HBV DNA提取及前C/C基因DNA扩增
取患者血清100 μL按QIAamp MinElute virus Spin试剂盒(德国Qiagen公司)说明书的方法进行血清HBV DNA抽提,产物溶解于30 μL无菌水中。第一轮引物为Pre-CF1和HBV2433R,第二轮引物为Pre-CF1和Pre-CR2,引物序列见表1。以PrimeSTAR Max(日本Takara公司)系统进行PCR扩增,反应条件为:94 ℃预变性4 min;94 ℃变性40 s,60 ℃退火30 s,72 ℃延伸1 min,共30个循环;72 ℃延链7 min。第1轮PCR结束后,取1 μL产物为模板,进行第二轮PCR。获得目的产物后,直接用于反向杂交或4 ℃储存。
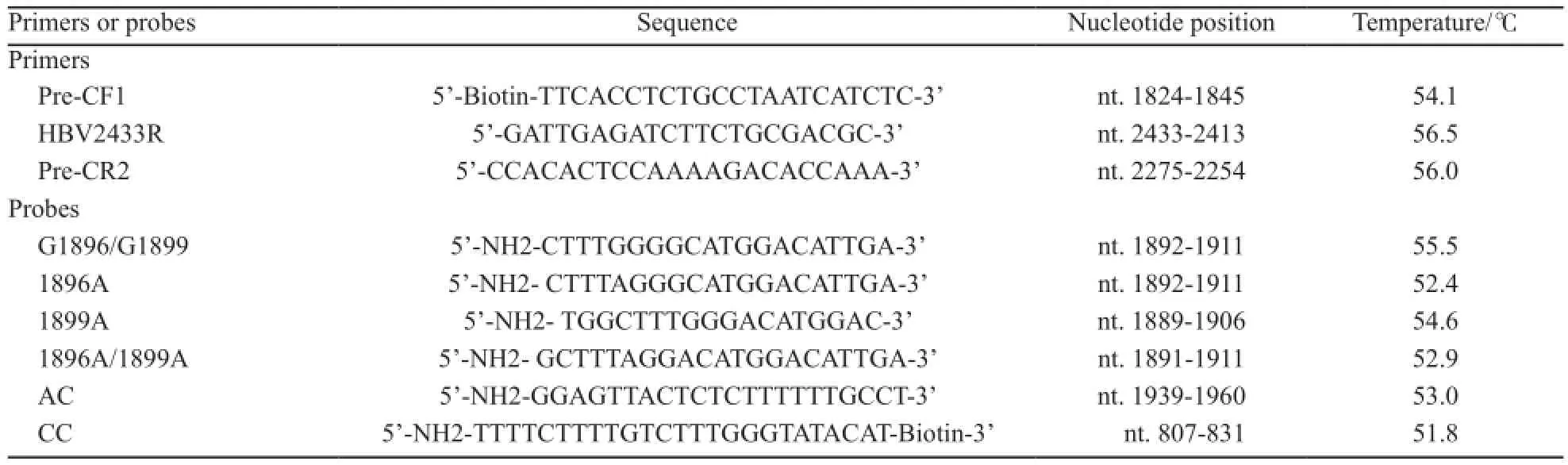
表1 引物与探针列表Tab. 1 The list of primers and probes used in the study
1.2.4 反向杂交检测HBV DNA前C区突变
①杂交膜的制备:将探针配制成10 pmol/ μL,每条探针各取1 μL按G1896/G1899、1896A、1899A、1896A/1899A、扩增质控和显色质控探针顺序固定于尼龙膜上。②反向杂交条件的优化:杂交与洗膜是杂交体系的关键步骤。用HBV野生型和突变型标准质粒依次对杂交液浓度、洗膜液浓度、洗膜时间和杂交温度进行优化。③显色:按照Biotin Chromogenic检验试剂盒(美国Thermo Scientific公司)的说明书进行操作。
1.2.5 直接测序及平端克隆
PCR产物经试剂盒(美国Axygen公司)纯化后送上海博尚生物技术有限公司进行直接测序。对于反向杂交检测结果与直接测序结果不一致的样本,进行平端载体克隆(方法同1.2.1),每份样本挑选10个克隆进行测序。
1.3 统计学处理
应用SPSS 12.0软件进行统计学分析,分类变量采用χ2检验或确切概率法检验,并比较显著性差异,计算OR值。所有检验均为双侧检验,P<0.05为差异有统计学意义。
2 结 果
2.1 反向杂交检测探针的设计
由于膜上所有的探针与PCR扩增片段均须在同一条件下(温度、离子强度和pH值)进行分子杂交和洗膜,故首先要求各种探针具有尽可能接近的退火温度。经过探针的优化,总结出探针的主要设计原则:①长度在17~26 bp;②退火温度在50~56 ℃;③待检测的突变位点尽量靠近序列的中间位置。具体序列见表1。
2.2 反向杂交检测HBV DNA前C区突变方法的建立及特异性检测
首先根据之前文献报道的条件进行预实验[16]:20 μL变性的野生型和3种突变型质粒PCR产物分别加入反应管中,用杂交液(6×SSC和0.1%SDS)于44 ℃杂交45 min,洗膜液(0.7×SSC和0.1%SDS)洗5 min,显色10~30 min。结果显示信号强度较好,但非特异性信号明显(图1A)。产生非特异性信号最常见的2个原因:杂交过强和洗脱不完全。因此我们首先优化杂交液浓度(6×SSC和0.1%SDS、4×SSC和0.1% SDS、2×SSC和0.1%SDS),其他条件不变。当杂交液浓度选用4×SSC和0.1%SDS,信号强度与特异性相对较好(图1A)。然后选用0.7×SSC和0.1%SDS、0.5×SSC和0.1%SDS、0.3×SSC和0.1%SDS共3种浓度依次降低的洗膜液洗膜,当选用0.5×SSC和0.1%SDS时,信号强度与特异性相对较好(图1B)。最后选用5、10和15 min这3种洗膜时间,可见在10 min的洗膜时间信号强度与特异性最好,无非特异性信号(图1C)。以此杂交条件为基础,缩小或增大杂交温度,杂交信号无明显改变,44 ℃仍然是理想的杂交温度(图1D)。
2.3 反向杂交检测的灵敏度
选用HBV DNA前C区1899位点进行灵敏度的检测,将G1896/G1899、1899A、扩增质控和显色质控探针置于膜上。半巢式PCR扩增102~106copy/mL的野生型和1899位点突变型标准质粒,各取20 μL变性的PCR产物分别进行反向杂交,可见该方法的最低检测下限为HBV DNA 103copy/mL(图2A和B)。
根据临床血清样本拷贝数检测,拷贝数大多数在104~105copy/mL,因此本研究选用104copy/mL野生型和突变型质粒,用反向杂交方法检测按100∶0、95∶5、90∶10、80∶20、60∶40、20∶80和0∶100比例进行混合的野生型和1899位点突变型质粒PCR产物。结果显示,突变株占混合株比例10%以上的均可被该方法检出(图2C)。此外,我们也对1896位点进行同样的灵敏度检测,发现结果与1899位点一致。
2.4 反向杂交与直接测序检测临床样本的结果比较
反向杂交检测50例肝癌血清样本HBV DNA前C区1896和1899位点的突变情况,并与直接测序结果比较(表2),2种方法符合率达98%,仅1例样品结果不一致,反向杂交检测为混合感染,而直接测序检测为野生型。将该例血清样品PCR扩增出HBV DNA前C区片段,并将PCR产物连接到平端载体克隆后,进行测序,测序结果显示该样品为混合感染株,与反向杂交检测结果一致。说明在检测混合感染方面,反向杂交更加准确。

图1 反向杂交方法的建立及特异性检测Fig. 1 Establishment and speci fi city detection of reverse hybridization to detect the HBV precore mutation
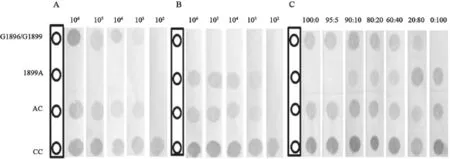
图2 反向杂交检测灵敏度Fig. 2 Determination of the sensitivity of the reverse hybridization
2.5 分析启东地区HBV DNA前C区突变与肝癌的关系
本研究继续用反向杂交方法检测启东地区50例肝癌和100例慢性肝炎患者的HBV前C区突变情况,并分析启东地区HBV DNA前C区G1896A和G1899A突变与肝癌的关系。结果显示:G1896A突变在肝癌组的发生率为48% (48/100),在慢性肝炎组的发生率为35%(35/100),两组间差异无统计学意义(P=0.062,OR=1.714,95%CI:0.971~3.026)。G1899A突变在肝癌组的发生率为35%(35/100),在慢性肝炎组的发生率为10%(10/100),两组相比差异有统计学意义(P=0.000,OR=4.846,95%CI:2.240~10.485,表3)。

表2 反向杂交与直接测序检测临床样本HBV DNA前C区突变的结果比较Tab.2 Comparison between direct sequencing and reverse hybridization results for the detection of HBV DNA precore mutation(n)

表3 分析江苏省启东地区HBV DNA前C区突变与肝癌的关系Tab. 3 Analysis of precore mutation in HCC and non-HCC patients from Qidong, Jiangsu Province
3 讨 论
肝癌是一种高度恶性的肿瘤,HBV感染是导致肝癌的重要原因[17]。日益增多的研究表明,HBV前C区突变是肝癌的危险因素,但由于方法学的缺陷限制了其在大规模危险人群的筛检[2-7]。目前已有的HBV前C区突变检测方法中,直接测序是检测前C区突变最常用的方法[2-7],可观察到HBV的已知和未知突变,但<20%的突变株容易被忽略。克隆技术虽然能弥补上述不足,但需要分析大量的克隆株。限制性片段长度多态性分析是使用核酸内切酶对扩增后的靶序列进行特异性位点的切割,再经过电泳判断结果。由于HBV基因组分子进化速度较快、变异频繁,因此结果分析准确率较低。实时荧光PCR不能同时检测多位点突变,其应用也受到限制。近年来,基因芯片技术已成功应用于核酸分析,对检测HBV基因组多态性具有很大的优势,但因其检测费用昂贵而限制了该技术在临床实验室的推广和应用。相比上述这些方法,反向杂交法具有以下几项优点:①探针固定于膜上,干燥后储存于4 ℃备用,保存期在12个月内一般不影响杂交结果。此膜可大量预先制备,检测时,只需加入PCR产物进行杂交,然后加酶显色即可。这样可节省制备膜的时间,减少操作步骤。②操作简单快速,无需特殊设备,从血清DNA抽提到样本检测完毕只需花费6.5 h,便于现场大规模人群的筛查及临床推广应用。③灵敏性高,可检测混合感染中10%以上的突变株。④高通量,可实现同张膜多个突变位点的检测,便于在临床中推广应用[18]。因此本研究建立了一种简单、快速、灵敏、准确的方法,该方法为肝癌预警提供了有效的监测工具。
反向杂交技术最为关键的是探针设计。要求各种探针具有尽可能接近的Tm,并需进行多次预试验优化探针长度、杂交温度、杂交液和洗膜液浓度等。生物素标记的扩增产物的大小,一般<500 bp,否则会因位阻效应影响膜上的寡核苷酸探针与生物素标记的基因扩增片段结合的效率和稳定性;此外,杂交膜条在不同溶液间转移速度太慢和显色时间过长也会产生非特异性杂交信号,从而影响结果的正确判断。
通过对启东地区HBV前C区突变的检测,结果显示,前C区1896位点突变与肝癌发生具有相关性,这也与其他文献的报道相一致[2,8]。HBV 1896位点突变可能是在宿主免疫压力下产生的一种对前C区其他突变的补偿机制,其出现可促进HBV前C区基因突变,导致病毒持续感染,令肝细胞损伤加重[19]。另外本研究还发现在启东地区HBV 1896位点突变虽有增加肝癌风险的趋势,但尚未达统计学意义。这与本实验室之前对肝癌及非肝癌患者的HBV全基因序列比对分析的结果一致[4]。但也有报道1896位点突变与肝癌发生具有相关性的研究[7]。本研究样本量较小,因此不能排除启东地区G1896A突变与肝癌有相关性。此外,还可能与不同地区HBV流行株的基因型不同有关。Yang等[20]在台湾地区的大规模分子流行病学研究结果显示,与1896位点未发生突变的患者相比,发生1896位点突变患者具有较低的患肝癌风险。在以A基因型为主的西欧及北美地区,1858位点为C,1896位点不易突变;而以B、C基因型为主的亚洲地区,1858位点为U,1896位点易发生突变。台湾及启东地区均是以B、C基因型流行株为主[20]。
越来越多的数据表明HBV联合突变较单点突变与进展性肝脏疾病发生显著相关。已报道的联合突变包括HBV DNA前C区G1896A突变叠加X区中的A1762T/G1764A和C1653T突变时,将进一步增加宿主罹患HCC的风险[21]。另外,Chen等[3]报道,同时发生HBV DNA G1899A、T1762/A1764和前S区缺失时,患者发生肝癌的风险是野生型病毒感染者的16.9倍。基于本项研究中突变位点探针的设计原则和已优化的杂交条件,我们还将建立检测HBV DNA C区、X区和P区肝癌高发相关突变位点的反向杂交技术[4,7,22],建立同张膜检测突变位点的高通量方法,为肝癌高发风险模型的建立提供方法学的基础。
综上所述,反向杂交检测HBV DNA前C区突变灵敏、方便、准确,可检测混合感染中10%以上的突变株,为肝癌预警提供了适合临床大规模推广应用的方法。
[1]CHEN C J, YANG H I, LLOEJE U H, et al. Hepatitis B virus DNA levels and outcomes in chronic hepatitis B [J]. Hepatology, 2009, 49(Suppl 5): 72-84.
[2]SUNG J J, TSUI S K, TSE C H, et al. Genotype-specific genomic markers associated with primary hepatomas, based on complete genomic sequencing of hepatitis B virus [J]. J Virol, 2008, 82(7): 3604-3611.
[3]CHEN C H, CHANGCHIEN C S, LEE C M, et al. Combinedmutations in pre-s/surface and core promoter/precore regions of hepatitis B virus increase the risk of hepatocellular carcinoma: a case-control study [J]. J Infect Dis, 2008, 198(11): 1634-1642.
[4]ZHU Y, JIN Y, GUO X, et al. Comparison study on the complete sequence of hepatitis B virus identifies new mutations in core gene associated with hepatocellular carcinoma [J]. Cancer Epidemiol Biomarkers Prev, 2010, 19(10): 2623-2630.
[5]CHO S W, SHIN Y J, HAHM K B, et al. Analysis of the precore and core promoter DNA sequence in liver tissues from patients with hepatocellular carcinoma [J]. J Korean Med Sci, 1999, 14(4): 424-430.
[6]胡晨波, 方敏, 陈晓蓉. 慢性乙型肝炎前C区和基本核心启动子突变的临床意义[J]. 肝脏, 2013, 18(8): 579-581.
[7]TONG M J, BLATT L M, KAO J H, et al. Basal core promoter T1762/A1764 and precore A1896 gene mutations in hepatitis B surface antigen-positive hepatocellular carcinoma: a comparison with chronic carriers [J]. Liver Int, 2007, 27(10): 1356-1363.
[8]LIAO Y, HU X, CHEN J, et al. Precore mutation of hepatitis B virus may contribute to hepatocellular carcinoma risk: evidence from an updated meta-analysis [J]. PLoS One, 2012, 7(6): 38394.
[9]QIN Y, ZHANG J, MAO R, et al. Prevalence of basal core promoter and precore mutations in Chinese chronic hepatitis B patients and correlation with serum HBeAG titers [J]. J Med Virol, 2009, 81(5): 807-814.
[10]涂文辉, 侯伟, 张瑾, 等. 基于单标记探针实时荧光定量PCR和熔解曲线分析方法定量检测HBeAg阳性患者前C区变异株[J].肝脏, 2013, 18(5): 297-302.
[11]NIE H, EVANS A A, LONDON W T, et al. Quantification of complex precore mutations of hepatitis B virus by simple probe real time PCR and dual melting analysis [J]. J Clin Virol, 2011, 51(4): 234-240.
[12]周秀梅, 曲迅, 刘福利, 等. 应用基因芯片技术检测乙型肝炎病毒前C区/BCP区基因突变的研究 [J]. 解放军医学杂志, 2003, 28(12): 1104-1106.
[13]MICHELON C T, ROSSO F, SCHMID K B, et al. Colorimetric microwell plate reverse-hybridization assay for mycobacterium tuberculosis detection [J]. mem Inst Oswaldo Cruz, 2011, 106(2): 194-199.
[14]PUEHRINGER H, NAJMABADI H, LAW H Y, et al. Validation of a reverse-hybridization strip assay for the simultaneous analysis of common alpha-thalassemia point mutations and deletions [J]. Clin Chem Lab Med, 2007, 45(5): 605-610.
[15]杨秉辉, 夏景林. 原发性肝癌的临床诊断与分期标准[J].中华肝脏病杂志,2001, 9(6): 324.
[16]刘彦辰, 张文露, 胡源, 等. 反向杂交法检测乙型肝炎病毒3种核苷(酸)类似物耐药突变[J]. 中华肝脏病杂志, 2010, 1(6): 414-418.
[17]杨艳, 仇小强, 曾小云, 等. 广西地区人群TNF-α基因启动子区多态性与肝癌的易感性研究[J]. 中国癌症杂志, 2012, 22(1): 35-41.
[18]ZENG X, KONG F, WANG H, et al. Simultaneous detection of nine antibiotic resistance-related genes in streptococcus agalactiae using multiplex PCR and reverse line blot hybridization assay [J]. Antimicrob Agents Chemother, 2006, 50(1): 204-209.
[19]CHUANG W L, OMATA M, EHATA T, et al. Precore mutations and core clustering mutations in chronic hepatitis B virus infection [J]. Gastroenterology, 1993, 104(1): 263-271.
[20]YANG H I, YEH S H, CHEN P J, et al. Associations between hepatitis B virus genotype and mutants and the risk of hepatocellular carcinoma [J]. J Natl Cancer Inst, 2008, 100(16): 1134-1143.
[21]CHEN B F, LIU C J, JOW G M, et al. High prevalence and mapping of pre-S deletion in hepatitis B virus carriers with progressive liver diseases[J]. Gastroenterology, 2006, 130(4): 1153-1168.
[22]BAI X, ZHU Y, JIN Y, et al. Temporal acquisition of sequential mutations in the enhancer Ⅱ and basal core promoter of HBV in individuals at high risk for hepatocellular carcinoma [J]. Carcinogenesis, 2011, 32 (1): 63-68.
Development and application of a reverse hybridization method for detection of hepatitis B virus precore mutation associated with hepatocellular carcinoma
ZHAO Zhi-mei1, ZHU Yu1, WU Yan1, FAN Chun-sun1, CHEN Tao-yang2, GAN Yu1, TU Hong1(1. State Key Laboratory of Oncogenes and Related Genes, Shanghai Cancer Institute, Renji Hospital, Shanghai Jiao Tong University School of Medicine, Shanghai 200032, China; 2.Qidong Liver Cancer Institute, Qidong Tumor Hospital, Qidong Jiangsu 226200, China)
TU Hong E-mail: tuhong@shsci.org
Background and purpose:It was reported that G1896A and G1899A mutation in the hepatitis B virus (HBV) precore region were all signi fi cantly associated with hepatocellular carcinoma (HCC). Simple, sensitive and reliable methods to detect precore mutations are needed in order to prevent the occurrence of HCC in clinical detection. The aim of this study was to develop a simple and sensitive reverse hybridization method for the detection of HBV precore mutation in HBV carriers. This method was applied for exploring the relationship between the precore mutations and HCC in patients of Qidong, Jiangsu Province.Methods:The speci fi c probes of nt.1896 and nt.1899 were designed and synthesized. In order to improve the sensitivity and specificity, reaction conditions of reverse hybridization were optimized. We used reverse hybridization to detect 50 HCC serum samples and compared the results with direct sequencing. Then we used this method to assess the association between HBV precore mutations and HCC in serum samples from 50 HCC patients and 50 non-HCC controls.Results:The detection limit of reverse hybridization for HBV DNA level was 103copy/mL and the sensitivity was 10% within a mixed virus population. The coincidence rate of reverse hybridization analysis was 98% compared with the direct sequencing results. It was showed that G1899A mutation was signi fi cantly associated with HCC compared to non-HCC controls (P=0.000, OR=4.846, 95%CI: 2.240-10.485), while G1896A mutation was not associated with HCC.Conclusion:Reverse hybridization is a simple andaccurate approach for the detection of low amounts of HBV precore mutants among a mixed viral population. It has potential usage in the large-scale screening of precore mutations in chronic hepatitis B carriers.
Hepatitis B virus; Precore domain; Reverse hybridization; Mutation; Hepatocellular carcinoma
10.3969/j.issn.1007-3969.2014.04.005
R735.7
A
1007-3639(2014)04-0266-07
2014-01-26
2014-03-01)
国家十二五重大科技专项(No:2012ZX10002-008-002)。
屠红 E-mail:tuhong@shsci.org

