趋化因子受体CXCR4非肽类抑制剂药效团的设计
符海洋,燕志慧,林润方,刁爱坡,肖冬光
(1. 天津科技大学生物工程学院,天津 300457;2. 南开大学药学院,天津 300071)
趋化因子受体CXCR4非肽类抑制剂药效团的设计
符海洋1,燕志慧1,林润方2,刁爱坡1,肖冬光1
(1. 天津科技大学生物工程学院,天津 300457;2. 南开大学药学院,天津 300071)
趋化因子受体CXCR4 是HIV进入宿主细胞的辅助受体之一.为了研究其抗HIV抑制剂的构效关系,6个结构多样性和拥有高活性的抑制剂被作为训练集来构建药效团模型,然后对由活性抑制剂和非活性抑制剂构成的测试集进行筛选,通过应用受试者工作特征(receiver operator characteristic,ROC)曲线和计算富集因子(enrichment factor,EF)的方法评估每个模型的预测能力与合理性.最终得到一个具有较高的ROC曲线下面积(area under curve,AUC)与富集因子的药效团模型,可用于指导新颖CXCR4抑制剂的设计及相关抗艾滋病药物的研发.
药效团;CXCR4受体;抑制剂;ROC曲线;富集因子
趋化因子受体CXCR4是基质细胞衍生因子1 (stromal cell-derived factor-1,SDF-1)的特异受体,后者是骨髓基质细胞产生的CXC趋化蛋白,系统命名CXCL12(CXC chemokine ligand-12).CXCR4受体与CXCL12蛋白协同表达于多种组织中,如脑、心脏、肾、免疫系统,在它们的运转与发育过程中起着相当重要的作用[1],并参与体内多种生理机制,如HIV-1病毒感染、造血和肿瘤迁移[2–3].CXCR4是由352个氨基酸组成的类视紫红质亚家族G蛋白偶联受体(GPCR),具有7次跨膜结构[4].当HIV-1病毒入侵细胞过程时,病毒刺突中的gp120蛋白与细胞膜表面的CD4复合物作用.在CD4的辅助下,gp120蛋白与CXCR4相互作用,使HIV-1病毒侵入细胞.因此,通过阻断或影响CXCR4受体与配体结合,可以阻碍HIV-1病毒对正常组织的侵染.以CXCR4为靶标的抗HIV抑制剂不断被报道[5–7],包括双环拉胺类抑制剂(AMD3100)、非拉胺类抑制剂(AMD070)和肽类抑制剂(T22).其中大多数抑制剂在体外有较好的抑制活性,但由于肽类抑制剂相对分子质量大,不利于分子的穿膜和吸收,以至于口服活性低.因此,AMD类抑制剂已成为目前最有前景的CXCR4抑制剂.药物之所以能够发挥药理作用是因为药物分子能够以特定的三维结构与体内受体发生结合作用,在三维空间中,药物分子特定的构象是呈现药物活性的必要前提.在计算机辅助药物设计中,药效团模型方法可以最大限度地利用已知的针对某一特定受体的活性分子三维结构信息来筛选并发现新的活性化合物.目前,报道基于AMD类抑制剂的药效团模型很少.为了研究其构效关系,本文选择了具有可靠类药性三维结构特征的6个AMD类抑制剂构建药效团模型,然后通过计算ROC曲线下面积(area under curve,AUC)和富集因子(enrichment factor,EF)对药效团模型的数据库筛选能力进行评价并选出最优的模型.
1 材料与方法
1.1 材料
224个CXCR4抑制剂被用来构建和验证药效团模型[8–22],其中6个抑制活性最佳的分子作为训练集以构建药效团模型[8–12].抑制活性使用pIC50值表征,保证了所构建模型可以表征能够较好地与CXCR4受体结合并产生抑制作用的化合物之物理化学特征,分子的结构与活性值如图1所示.在Schrödinger的软件中[23],构建的6个分子在Ligprep模块中用OPLS 2005力场对其结构进行优化,最后使用MCMM(monte carlo multiple minimum)方法对经过优化的分子进行构象搜索,设定最大分子位置偏差为0.05nm以除去冗余构象.

图1 用于构建药效团的抑制剂化学结构及其抑制活性(pIC50)Fig. 1 Inhibitor structures used for pharmacophore generation and their inhibitory activities(pIC50)
1.2 模型的构建
使用Schrödinger软件中的Phase模块构建药效团模型步骤如下[23]:(1)导入经过前处理的分子集,(2)选择并编辑活性位点,(3)寻找分子间通用的药效团结构,(4)对得到的药效团模型假设进行打分.当给出1个与同一特定受体作用的分子集合后,Phase采用K-point药效团分析法对每一个活性的分子进行分析,统计每个分子的药效团位点,包括氢键受体位点、氢键供体位点、疏水区域和芳香性环.建立模型后,Phase会对每个模型进行打分,在对药效团模型打分过程中,设定假设模型的RMSD上限值为0.11nm,以提高打分效率.
1.3 测试集的生成与验证
为验证模型的准确性与有效性,构建了一个测试分子集.分子集由剩余的218个CXCR4抑制剂[8–22]和由DecoyFinder软件搜索出的5,261个Decoy组成.Decoy是一些针对特定靶标无活性的分子,如不可能与靶标发生作用的分子,它们通常用来计算配体富集因子(ligand enrichment factor)以验证针对特定靶点虚拟筛选流程的准确性.在Decoy Finder软件中导入218个抑制剂,选择搜索目标为Zinc数据库.DecoyFinder可以在数据库中和给定的活性分子集中分别计算MACCS指纹特征和五点物理性质描述符(physical descriptors),通过比对活性分子和数据库中潜在的Decoy间两个特征的差别,搜索出针对CXCR4受体的Decoy分子[24].
使用Phase模块中的Find Matches to Hypothesis对上面获得的分子三维结构数据库进行基于药效团模型的搜索.对于单个化合物,搜索过程中都要对该化合物的每个三维构象进行比对,最小满足位点个数设置为5个,最大允许位点偏离距离设置为0.2nm.搜索完成后,分子库中符合药效团假设的分子被筛选出来,并计算出这些分子的拟合度(fitness).然后通过计算EF、AUC值验证每一个药效团模型预测能力.
EF是评价筛选流程优劣的一个重要指标,它是指数据集中活性分子的命中率与初始活性分子浓度的比值,这个指标用于评价筛选流程中命中有效活性分子的阳性率.富集因子计算公式[25]为EF=m/n.其中:m=NActive Fit/NTotal Fit,NActive Fit指筛选结果中活性分子的个数,NTotalFit指筛选所得分子总数;n=NAll Active Comp/NAll Comp,NAll Active Comp指被筛选分子中活性分子个数,为218个,NAllComp指被筛选分子总个数,为5,479个.
对所得到的数据表进行ROC曲线分析.ROC曲线表征的是敏感度(sensitivity)和特异性(specificity)之间的关系[26–27].评价筛选流程的好坏,则会使用到AUC值,即ROC曲线下面积.当被评价的筛选流程能够筛选出同等浓度的非活性Decoy分子时,筛选出越多的活性分子就代表该流程对活性分子的敏感度更高.AUC值是以具体数值表示ROC曲线下面积,取值范围为[0–1]:“0”表示筛选结果包含全部的Decoy分子,不包含活性分子;“1”表示筛选结果包含全部活性分子,不包含Decoy分子.在曲线的制作过程中,将全部活性分子的化合物名称改为“1”,全部Decoy分子的化合物名称改为“0”,设置化合物名称变量为检验变量,Fitness值为状态变量.选择其中富集因子较大,AUC值较高的模型作为CXCR4受体抑制剂的药效团模型.
将与药效团模型拟合度较高的活性分子使用Schrödinger软件的Glide模块[23]与CXCR4受体结构(PDB 3ODU)[4]进行分子对接,分析配体与受体间作用模式、药效团特征与蛋白结构特征间的联系与吻合程度,进而验证所获得的药效团模型的合理性.
2 结果与讨论
在Schrödinger软件包中[23],使用Phase模块构建CXCR4抑制剂的药效团模型,得到9个五点药效团假设模型.模型由氢键供体(A)、疏水中心(H)、正电中心(P)和芳香环(R)中的3个元素或4个元素组成.9个药效团模型的Survival值范围为2.538~2.803,Selectivity值范围为2.332~2.501,均具有较好的选择性和代表性.为了考察所得药效团模型的数据库筛选能力,用以上所得9个药效团模型为提问结构,搜索由5,479个化合物构成的测试集(218个CXCR4受体抑制剂和5,261个Decoy分子,其中不含构建药效团模型所用的6个抑制剂化合物),并用AUC值和EF值评价9个药效团模型的合理性与筛选效率,结果见表1.
运用药效团进行虚拟筛选的目的是希望具有抑制活性的化合物分子出现在筛选结果中,而对CXCR4受体无抑制作用的Decoy分子则尽可能不被筛选出来.在统计结果上表征为ROC曲线下面积越大越好,EF值更高,借此可以选择药效团模型中最优的一个,以用于指导和筛选抗HIV活性的CXCR4抑制剂.由表1可知,AHPRR.34和AHPRR.38模型虽然能得到更多的活性分子,但是这两个模型的假阳性都比较高,不适合进行数据库的筛选.剩余的7个模型对于活性分子的识别能力几乎都一样,不同的是对非活性分子的识别能力.其中与化合物2拟合最好的模型HPRRR.114(图2)能从5,261个Decoy分子中得到更少的非活性分子(95个),EF值为15.95,AUC值为0.913(ROC曲线如图3所示),表明HPRRR.114模型的筛选结果中含有更大比例的活性分子,且活性预测的偏差较小,证明该模型具有较好的活性预测能力.因此,选择该模型用以进一步研究小分子抑制剂与CXCR4受体的构效关系.

表1 药效团模型筛库能力参数Tab. 1Performance of pharmacophore models in library screening
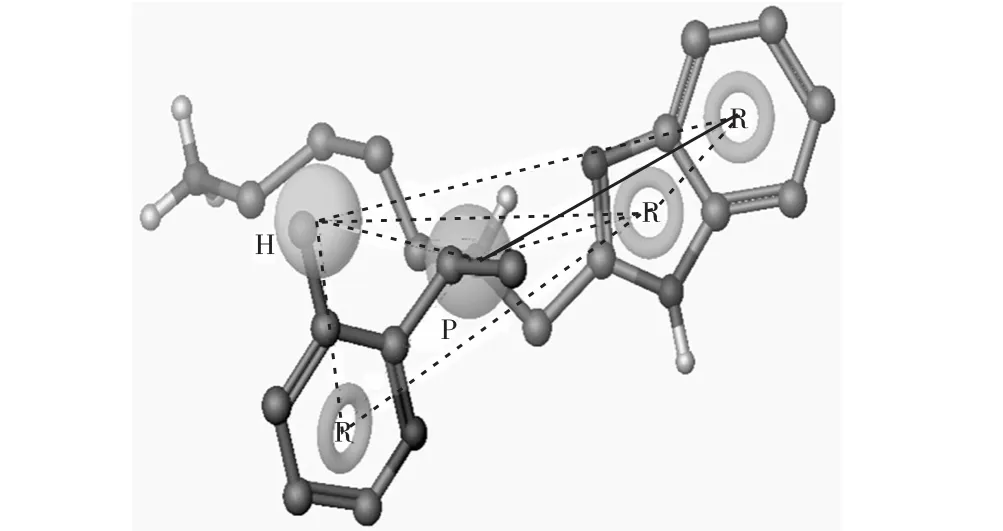
图2 药效团模型HPRRR.114与化合物2的叠合图Fig. 2 Compound 2 mapped into model HPRRR.114

图3 HPRRR.114对测试集筛选的ROC曲线图Fig. 3 The ROC curve of HPRRR.114 in screening tests
将化合物2以其与药效团拟合度最高的构象对接到受体蛋白中,分析受体与配体的相互作用(对接结果如图4(a)所示.图中白色配体为化合物2,化合物2上黑色为氮原子).由图4(a)可以看到化合物2的吡啶环与受体口袋中TRP.94的芳香稠环及HIS.113的咪唑环发生了π-π相互作用,与芳香环(R)特征相符合;配体中心处带有正电荷的氮原子距离受体的ASP.97较近.Wu等[4]的研究表明,ASP.97的侧链可以与配体带正电荷的部分产生盐桥,表明在这一位置存在正电荷药效团特征(P)对于生物分子的活性发挥与结构稳定有很重要的作用.与此同时,对受体的亲水/疏水趋向面进行分析(结果如图4(b)所示,亮白色代表疏水趋向,黑色代表亲水趋向),发现化合物2吡啶基团上的甲基正好深入了活性口袋中的疏水部分,从而与周围疏水性氨基酸残基发生范德华力作用,加强了对接产物的稳定性,而在之前的药效团构建结果中,疏水特征位点(H)恰好拟合于该甲基位点上.由此可见,对接结果显示的配体与受体间相互作用的模式与药效团特征比较吻合.

图4 化合物2与受体蛋白的作用模式以及受体的亲水/疏水趋向面图Fig. 4 The interaction between compound 2 and the receptor,and the hydrophilicity/hydrophobicity surface
本次研究利用Schrödinger软件构建CXCR4抑制剂的药效团模型,得到一个含有5个药效团特征的模型,包括1个疏水中心(H)、1个正电中心(P)、3个芳香环(R).疏水中心匹配在化合物的3–甲基吡啶的甲基上;3个芳香性环分别匹配在了吡啶环上和苯并咪唑的2个稠环上;正电中心匹配在了化合物中心的阳性氨基上,并且各个位点之间的距离与位置均要满足一定的空间限制(药效团模型结构见图2).综合比对得到的药效团模型和CXCR4蛋白结构模型发现,CXCR4受体抑制剂与受体活性位点之间的相互作用形式主要包含氢键相互作用、π-π相互作用和疏水相互作用,并且各个基团之间满足一定的空间限制,从而产生抑制CXCR4受体的活性作用.
3 结 语
本文应用6个结构多样性和拥有高活性的抗HIV的抑制剂来构建药效团模型,然后对测试集进行筛选,通过应用ROC曲线和计算EF值的方法评估每个模型的预测能力与合理性.最终得到1个最优的模型HPRRR.114,为进一步指导和筛选潜在的抗HIV药物分子提供了理论依据.
[1] Horuk R. Physiology:Chemokines beyond inflammation [J]. Nature,1998,393(6685):524–525.
[2] Deng H K,Liu R,Ellmeier W,et al. Identification of a major co-receptor for primary isolates of HIV-1[J]. Nature,1996,381(6584):661–666.
[3] Burger J A,Kipps T J. CXCR4:A key receptor in the crosstalk between tumor cells and their microenvironment [J]. Blood,2006,107(5):1761–1767.
[4] Wu B,Chien E Y,Mol C D,et al. Structures of the CXCR4 chemokine GPCR with small molecule and cyclic peptide antagonists[J]. Science,2010,330(6007):1066.
[5] Clercq E D. The bicyclam AMD3100 story[J]. Nature Revive Drug Discovery,2003,2(7):581–587.
[6] Stone N D,Dunaway S B,Flexner C,et al. Multiple-dose escalation study of the safety,pharmacokinetics,and biologic activity of oral AMD070,a selective CXCR4 receptor inhibitor,in human subjects[J]. Antimicrobial Agents and Chemotherapy,2007,51(7):2351–2358.
[7] Masuda M,Nakashima H,Ueda T,et al. A novel anti-HIV synthetic peptide,T-22([Tyr5,12,Lys7)]polyphemusin II)[J]. Biochemical and Biophysical Research Communications,1992,189(2):845–850.
[8] Skerlj R,Bridger G,McEachern E,et al. Design of novel CXCR4 antagonists that are potent inhibitors of T-tropic(X4)HIV-1 replication[J]. Bioorganic & Medicinal Chemistry Letters,2011,21(5):1414–1418.
[9] Jenkinson S,Thomson M,McCoy D,et al. Blockade of X4-tropic HIV-1 cellular entry by GSK812397,a potent noncompetitive CXCR4 receptor antagonist[J]. Antimicrobial Agents and Chemotherapy,2010,54(2):817–824.
[10] Skerlj R,Bridger G,McEachern E,et al. Synthesis and SAR of novel CXCR4 antagonists that are potent inhibitors of T tropic(X4)HIV-1 replication[J]. Bioorganic & Medicinal Chemistry Letters,2011,21(1):262–266.
[11] Catalano J G,Gudmundsson K S,Svolto A,et al. Synthesis of a novel tricyclic 1,2,3,4,4,a,5,6,10,boctahydro-1,10-phenanthroline ring system and CXCR4 antagonists with potent activity against HIV-1[J]. Bioorganic & Medicinal Chemistry Letters,2010,20(7):2186–2190.
[12] Gudmundsson K S,Boggs S D,Catalano J G,et al. Imidazopyridine-5,6,7,8-tetrahydro-8-quinolinamine derivatives with potent activity against HIV-1 [J]. Bioorganic & Medicinal Chemistry Letters,2009,19(22):6399–6403.
[13] Skerlj R T,Bridger G J,Kaller A,et al. Discovery of novel small molecule orally bioavailable C-X-C chemokine receptor 4 antagonists that are potent inhibitors of T-tropic(X4)HIV-1 replication [J]. Journal of Medicinal Chemistry,2010,53(8):3376–3388.
[14] Ueda S,Kato M,Inuki S,et al. Identification of novel non-peptide CXCR4 antagonists by ligand-based design approach[J]. Bioorganic & Medicinal Chemistry Letters,2008,18(14):4124–4129.
[15] Miller J F,Turner E M,Gudmundsson K S,et al. Novel N-substituted benzimidazole CXCR4 antagonists as potential anti-HIV agents[J]. Bioorganic & Medicinal Chemistry Letters,2010,20(7):2125–2128.
[16] Miller J F,Gudmundsson K S,Richardson L D,et al. Synthesis and SAR of novel isoquinoline CXCR4 antagonists with potent anti-HIV activity [J]. Bioorganic & Medicinal Chemistry Letters,2010,20(10):3026–3030.
[17] Jacobson O,Weiss I. D,Szajek L,et al.64Cu-AMD 3100:A novel imaging agent for targeting chemokine receptor CXCR4[J]. Bioorganic & Medicinal Chemistry,2009,17(4):1486–1493.
[18] Murakami T,Kumakura S,Yamazaki T,et al. The novel CXCR4 antagonist KRH-3955 is an orally bioavailable and extremely potent inhibitor of human immunodeficiency virus type 1 infection:Comparative studies with AMD3100[J]. Antimicrobial Agents and Chemotherapy,2009,53(7):2940–2948.
[19] Ichiyama K,Yokoyama-Kumakura S,Tanaka Y,et al. A duodenally absorbable CXC chemokine receptor 4 antagonist,KRH-1636,exhibits a potent and selective anti-HIV-1 activity[J]. Proceedings of the National Academy of Sciences,2003,100(7):4185–4190.
[20] Thoma G,Streiff M B,Kovarik J,et al. Orally bioavailable isothioureas block function of the chemokine receptor CXCR4 in vitro and in vivo[J]. Journal of Medicinal Chemistry,2008,51(24):7915–7920.
[21] Rosenkilde M M,Gerlach L O,Hatse S,et al. Molecular mechanism of action of monocyclam versus bicyclam non-peptide antagonists in the CXCR4 chemokine receptor[J]. Journal of Biological Chemistry,2007,282(37):27354–27365.
[22] Habashita H,Kokubo M,Hamano S,et al. Design,synthesis,and biological evaluation of the combinatorial library with a new spirodiketopiperazine scaffold. Discovery of novel potent and selective low-molecularweight CCR5 antagonists[J]. Journal of Medicinal Chemistry,2006,49(14):4140–4152.
[23] McRobb F M,Capuano B,Crosby I T,et al. Homology modeling and docking evaluation of aminergic G Protein-Coupled Receptors[J]. Journal of Chemical Information and Modeling,2010,50(4):626–637.
[24] Cereto-Massagué A,Guasch L,Valls C,et al. Decoy-Finder:An easy-to-use python GUI application for building target-specific decoy sets[J]. Bioinformatics,2012,28(12):1661–1662.
[25] Güner O F. Pharmacophore Perception,Development,and Use in Drug Design[M]. La Jolla:International University Line,2000:422.
[26] Hevener K E,Zhao W,Ball D M,et al. Validation of molecular docking programs for virtual screening against dihydropteroate synthase[J]. Journal of Chemical Information and Modeling,2009,49(2):444–460.
[27] Truchon J F,Bayly C I. Evaluating virtual screening methods:Good and bad metrics for the “early recognition” problem[J]. Journal of Chemical Information and Modeling,2007,47(2):488–508.
责任编辑:郎婧
Pharmacophoric Design for Non-peptide Inhibitors of Chemokine Acceptor CXCR4
FU Haiyang1,YAN Zhihui1,LIN Runfang2,DIAO Aipo1,XIAO Dongguang1
(1. College of Biotechnology,Tianjin University of Science & Technology,Tianjin 300457,China;
2. College of Pharmacy,Nankai University,Tianjin 300071,China)
CXCR4 is one of several chemokine receptors that HIV can use to infect host cells. To study the structure-activity relationship between chemokine receptor CXCR4 and its inhibitors,six inhibitors with structural diversity and high-activity were used to develop pharmacophore models,and then a molecular library consisting of active and decoy ligands was applied to evaluate the predicting capability and rationality of every model by receiver operator characteristic(ROC)curve and enrichment factor(EF)value. The result should that a pharmacophore model with higher area under ROC curve and EF value was obtained,which can be used to guide the design of novel CXCR4 inhibitors and the development of the related anti-HIV drugs.
pharmacophore;CXCR4 receptor;inhibitor;ROC curve;EF value
O642.3
A
1672-6510(2014)02-0006-05
10.13364/j.issn.1672-6510.2014.02.002
2013–11–15;
2013–12–23
国家高技术研究发展计划“863计划”资助项目(2012AA023408)
符海洋(1989—),男,河南内乡人,硕士研究生;通信作者:刁爱坡,教授,diaoaipo@tust.edu.cn.
- 天津科技大学学报的其它文章
- 氨式碳酸铜溶液中铜离子含量的测定方法

