真菌中DNA重复序列诱导点突变的研究进展*
冯凤鹃,曲志才,田李,王转斌
(曲阜师范大学生命科学学院,山东 曲阜 273165)
真菌在生物界中占据很大的一个类群,现在已经发现的有七万多种,估计只是所有存在的一小部分.重复序列诱导的点突变(repeat-induced point mutation,RIP)是真菌所特有的一种真核生物基因沉默机制,自1987年在粗糙脉孢菌(Neurosporacrassa)中首次发现RIP以来[1],以及在其他丝状子囊菌中RIP的研究,逐渐揭示了真菌的表观遗传学机理及其现象,使其能够更好地为人类服务,尤其是在工农业生产、医药、环境等领域对人类产生的影响更为深刻.此外,对粗糙脉孢菌基因组序列的分析还显示,RIP通过基因重复对基因组进化有着显著影响[2].这些研究结果也促进了高等动植物表观遗传学的发展.
1 重复序列诱导点突变
1.1 重复序列诱导点突变的概念
重复序列诱导点突变(RIP)是以同源性为基础的使重复DNA序列发生突变的过程,并通过DNA甲基化导致突变序列表观遗传沉默[3].最初的研究结果显示只有在有性周期作用下,RIP能够识别那些重复序列长度超过约400 bp(或不连锁的重复序列超过约1kb)、相似性大于80%的片段,从而触发RIP的发生[4].通过对粗糙脉孢菌全基因组序列分析,发现该基因组的甲基化绝大多数与RIP有关,在发生RIP的序列中,80%以上的胞嘧啶被甲基化[5].通过RIP,粗糙脉孢菌序列中约30%的G-C碱基对突变成为A-T对,其主要发生在CpA二核苷酸区域,使CpA转换成为TpA,从而形成富T+A 片段[6].RIP突变的序列在营养细胞中常常引起DNA甲基化导致基因沉默,有时这种甲基化不仅包括重复序列自身,还会延伸至重复序列之外[7].此外,粗糙脉孢菌的着丝粒区存在着丰富的与转座子相关的序列,但是这些序列大多数已被突变[8].粗糙脉胞菌中重复序列诱导点突变(RIP)的过程,如图1所示.
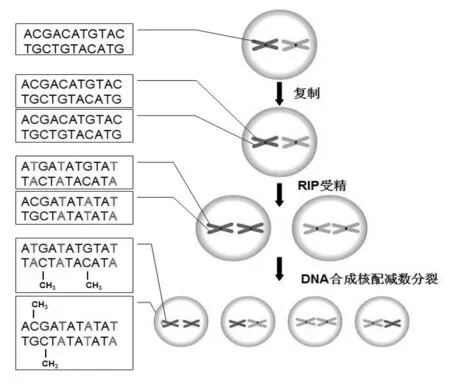
图1 粗糙脉胞菌中RIP的过程
随着对真菌中RIP研究的日渐关注,此后数年在其他真菌中也先后发现存在RIP,如子囊菌鹅柄孢壳菌(Podosporaanserina)、稻瘟病菌(Magnaporthegrisea)、十字花科小球腔(LeptosphaeriaMaculans)、尖孢镰刀菌(Fusariumoxysporum)、米曲霉(A.oryzae)、黑曲霉(A.niger)、构巢曲霉(A.nidulans)、产黄青霉(Penicilliumchrysogenum)和谷类炭疽病(Colletotrichumcereale)[9],以及担子菌花药黑粉菌(Microbotryumviolaceum)等.与粗糙脉孢菌相比,其他真菌的RIP比较温和,许多重复序列具有活性或即使没有活性也很少发生突变,如鹅绒委陵菜柄孢壳菌中重复序列的检测效率较低,突变就少[10];黑曲霉RIP仅限于使少数转座子序列的开放阅读框中止;而产黄青霉中受RIP影响的序列则较多[2].
1.2 RIP的产生机制
用生化的方法来对RIP的机制进行研究是十分困难的,因为这个过程发生在微观的产子囊的组织中.虽然到目前为止RIP的机制还不是很清楚,但普遍认可的模型是RIP需要一个RIP缺陷型基因,该基因被预测为可以编码一个DNA甲基转移酶(DMT)或类似DMT的酶,称之为RID,其拥有六个最高度保守的基因组序列,这一特性是真核DMT的特性[11].RID首先使胞嘧啶甲基化,随后在脱氨酶作用下使胞嘧啶脱氨基生成胸腺嘧啶.脱氨步骤可能是由DMT催化和对甲基供体的低水平细胞参与作出的RIP反应诱变共同完成的,但实际的步骤仍然是不确定的,因为有性阶段的胞嘧啶甲基化尚未见报道[12].目前符合这一模式的除了粗糙脉孢菌中的RID,还有构巢曲霉中的DMTA以及米曲霉中的RID同源物DMTAO[13].RID缺陷突变体显示RIP隐性缺陷,但不显示任何DNA甲基化的明显缺陷或对可育性、生长或发育有影响.
参与RIP的其他因素还有待发现.例如,RIP在整个基因组中如何有效地检测小范围内重复是不成立的.RIP的特性意味着DNA与DNA直接配对.对含有两个或更多拷贝的给定基因所在细胞核的RIP分析显示,RIP以完全配对的方式起作用[14].也许RIP发生的过程,类似于同源重组这一众所周知的功能,但是值得注意的是如果取消染色体配对和减数分裂重组,以及降低串联重复的切除,这些事件都会损害RIP[15].
1.3 RIP与DNA甲基化的关系
与高等动植物相比,大多数真菌基因组DNA中胞嘧啶甲基化程度很低或不存在甲基化,只有少数真菌胞嘧啶甲基化程度较高,如粗糙脉孢菌、多头绒泡菌、布拉克须霉、黄曲霉等.因此,甲基化对于真菌可能并非是必需的[16].丝状真菌的甲基化主要发生在转座子和其他重复序列,大部分基因组并不存在甲基化,包括CpG二核苷酸.粗糙脉孢菌基因组中的重复DNA序列相对比较少,大约占8%,其中约81%发生甲基化[17].粗糙脉孢菌等真菌基因组中的重复序列的甲基化是与RIP密切相关的,RIP将重复序列中的CpA突变生成TpA,产生富T+A片段,这些富T+A片段就作为甲基化信号,用来引发DNA的重新甲基化.在粗糙脉孢菌的基因组中约2%的胞嘧啶发生甲基化,并且该甲基化已被证明能导致基因沉默.体内重组实验表明, (TAAA)n和(TTAA)n能够刺激绝大多数的甲基化,当富T+A片段长度在75 bp及以上时即可有效地行使甲基化信号功能.然而,RIP本身的引发只需要重复序列,其重新和维持性甲基化并不依赖多拷贝的富T+A片段[18].
最近的全基因组分析表明,大多数发生DNA甲基化的部位与显示RIP特征的序列相关联,即通过RIP引起的序列突变通常是甲基化的,但它仅仅是轻度突变以至于无甲基化现象显示 .有趣的是,当以转化的方式重新进入生物体时,大多数RIP突变的序列行使的是可重复重新甲基化.迄今的结果表明,重新甲基化是由于识别富含A-T短片段的小沟而触发的.与重新甲基化相对应的是维护甲基化,它是通过某些用来触发重新甲基化的RIP产物甲基化的失败来证明的[19].在粗糙脉孢菌中,除了RID,还存在DIM-2缺陷型,它是用来负责这个生物体中所有已知的甲基化的.与RID不同的是,DIM-2并不是引发RIP所必需的[16].尽管在RIP发生期间并没有直接的证据证明DNA甲基化,但在有性阶段已经观察到.现在待研究的就是去确定RID是否是一个真正的DMT和阐明RIP与DNA甲基化之间的关系.
2 真菌中重复DNA序列诱导点突变(RIP)的研究进展
2.1 粗糙脉孢菌中RIP的研究进展
丝状真菌粗糙脉孢菌因其生长速度快,在特定介质上容易传播,并且遗传操作如诱变、互补实验和基因定位都很简单等优点,首先发展成为生物实验的模式菌种,并被提出著名的“一个基因一个酶”的Beadle 和Tatum所选定用来研究生物化学和遗传学之间的联系[20].因此对粗糙脉孢菌中RIP的研究开展的最早,也最全面.
Selker和Stevens(1985),首次在粗糙脉孢菌中长约1.6 kb的Z- H(泽塔-ETA)区发现甲基化特征的存在,该区域包含一个0.8 kb DNA片段的串联重复,内含5S rRNA基因.通过与缺乏重复序列的菌株相应染色体区域的比较,最初提出来重复序列能以某种方式诱导DNA甲基化的想法,并最终导致了基因组防御系统RIP的发现[21].RIP能够诱导粗糙脉孢菌基因组中重复序列的甲基化,通过对粗糙脉孢菌染色体的ζ-η区(ζ和η分别为5S RNA的基因,二者序列高度相似且位于同一染色体上)DNA序列的研究,结果发现在脉孢菌生活史的有性阶段,存在着二个不同交配型的单核菌丝,在减数分裂核融合前,会发生一个瞬时双核体阶段,此时染色体ζ-η区的重复序列就会变得很不稳定,从而容易缺失或发生重新甲基化[22].在粗糙脉孢菌中,也并不是所有的重复基因都发生RIP,如分散的5S rRNA和tRNA基因,因为它们的长度小于RIP的阈值[23].
RIP在粗糙脉孢菌处于受精和减数分裂的有性阶段时,对富含CpA的片段具有偏爱性,致使CpA发生甲基化,在发生RIP的序列中,80%以上的胞嘧啶被甲基化(Cambareri等,1989;Selker等,1987).RIP能够检测的DNA重复大于400 bp,包括节段性重复,并使重复序列中大约30%的G-C碱基对发生突变最终变为A-T碱基对,这一特性被认为是为转座因子的传播提供的一种防御机制(Galagan和Selker,2004).早期的研究结果,为反转录转座子Tad和DNA型转座子Punt中RIP介导的灭活提供了明确的证据,通过对位于连锁群七区的着丝粒区域序列的分析,发现了其他类型转座子的遗迹[24].另外,对与DNA甲基化有关的基因组序列进行探寻,揭示了一大批RIP灭活转座子的集合点[25].最近,对粗糙脉孢菌野生型菌株的基因组序列的分析,为RIP和可移动元件之间的关系提供了一个全面的视图.虽然众多的附加转座子被确定,但是在基因组序列中并没有一个单一完整的移动元件被鉴定.到目前为止,在着丝粒区已发现了高密度的灭活转座子,除了最普遍的与反转录元件相关的序列片段,DNA型转座子遗迹也很丰富[26].
近年来,对RIP的研究发现它可能对粗糙脉孢菌的演变有着显著影响.粗糙脉孢菌最显著的特点之一是其基因组序列几乎没有完全高度相似的成对基因,通过对粗糙脉孢菌中10082编码蛋白质的基因进行预测,发现只有6对它们的编码序列能够共享大约80%的核苷酸或氨基酸的特性[2].如前所述,RIP能变异那些DNA重复大于400 bp、相似性大于80%的核苷酸序列.对于粗糙脉孢菌中高度相似基因对的缺乏,存在一个简约的解释即RIP通过重复序列来防止功能基因的出现.然而,它也有可能是一些被RIP突变后的重复基因继续保持功能并积极地分化所造成的.虽然一些重复基因对可以存活几个性周期,但在进化的过程中绝大多数这样的基因对似乎被RIP灭活.连同高度相似的基因对缺乏,这些数据表明在粗糙脉孢菌中,大多数的旁系同源重复和RIP出现前分化以及通过基因复制产生的新基因的演变几乎被阻止[27].但在粗糙脉孢菌中发现单轮RIP相当温和,加倍化基因的一个或多个拷贝不仅保持其功能并有新的等位基因生成,因此,短暂加倍的基因通过RIP可在群体水平上增加基因多样性,并有可能会加快现有基因的演变[28].RIP除了参与基因进化以外,对真菌的表观遗传学现象及其机理的揭示和了解也具有很大的意义,这些研究结果也促进了高等动植物表观遗传学的发展.
目前,在粗糙脉孢菌中仍然存在的问题包括:是不是所有的重复序列都能发生RIP突变?RIP在何种程度上影响基因组进化?是否有迹象显示RIP可以通过变异产生新的功能,来加速某些基因的进化?RIP的产物是被迅速淘汰或是被保留[25]?这些问题的提出,对后人在粗糙脉孢菌RIP的研究中特别具有启发性.
2.2 其他丝状子囊菌中RIP的研究进展
RIP除了存在于粗糙脉孢菌中,也广泛存在于其他丝状子囊菌中.Clutterbuck(2010)运用两种不同的方法对49种丝状子囊菌基因组进行检测,并证明了RIP机制的存在.第一种方法是对真菌基因组中的转座元件和重复序列进行鉴定,并按照公式对显示RIP机制的CpG双突变和CpC二核苷酸的数量进行分析,从而定量突变率; 第二种方法是通过对全基因组核苷酸频率分布异常的评估,发现在允许非编码基因组进行定量的区域存在着广泛的定向突变[29].这两种方法的结果显示,无论是突变的程度还是核苷酸方面C到T的转换数量,都说明不仅存在RIP,而且具有多样化.只有球毛壳菌,没有证据显示定向突变.下面重点介绍几种常见真菌的RIP研究进展.
2.2.1 稻瘟病菌
Nakayashiki(1999)等人在稻瘟病菌中发现存在营养甲基化; Ikeda(2002)等报道了稻瘟病菌中反转录转座子MAGGY区域C到T的突变,使其最终成为一个富含T+A 片段,但在MAGGY区域不同的稻瘟病菌菌株RIP的变化程度很低,只有0.06%~0.72%,而Pyret区域高达32.8%[30].同年,Shiflett等人在LTR(一种能复制的反转录转座子)的LTRs部位发现了与MAGGY区域相似的特性[31].Thon(2004)等人通过研究指出MAGGY元件的TS/TV比值高达6.8,但在其他的TE家族中TS/TV的比值普遍偏低,如MGL约4.2,而POT2甚至只有2.3.这些数据的差异是由不同菌株中序列的特异性引起的缺乏这些二核苷酸.最新的研究表明,与鹅绒委陵菜柄孢壳相比,在稻瘟病菌基因组中富含T+A的DNA片段较少,约有3%的基因组中明显缺乏这些二核苷酸[29].目前已经证明,稻瘟病菌存在有性阶段,但只在实验室条件下观察得到.
2.2.2 鹅绒委陵菜柄孢壳
Graïa(2001)等人通过重复实验证明鹅绒委陵菜中存在RIP,而且与粗糙脉孢菌中的RIP相比比较温和[32].Hamann(2000)发表报告称发现了两种具有RIP机制的转座子:Pat,表明CpA和CpT二核苷酸转换的频率相等,与de-RIP共有序列相关;Yeti,是一种反转录转座子,缺乏CpA二核苷酸.这些元件大部分的长度可以保持一致,但在de-RIP共有区存在差异,一共有13个颠换和308个C到T的转换[33].虽然鹅绒委陵菜柄孢壳中二核苷酸转换的频率与粗糙脉孢菌的相似,但鹅绒委陵菜柄孢壳的基因组相比于粗糙脉孢菌含有较少的富T+A碱基对的DNA片段.Bouhouche(2004)和Arnaise(2008)团队也在鹅绒委陵菜中发现RIP机制[34].其实,早在1987年就有人提出在鹅绒委陵菜柄孢壳中存在串联重复序列,但是至今也只有几个RIP事件的报道,可见在这个物种中RIP是罕见的.
2.2.3 尖孢镰刀菌,串珠镰刀菌,禾谷镰刀菌,茄病镰刀菌
Hua-Van(2001)等在尖孢镰刀菌中发现几个能够呈现出RIP特性的黑斑羚因子[35].Daboussi(2002)等在镰刀菌中发现转座因子Fot1,在其转座序列中RIP的频率约为28%.通过对尖孢镰刀菌存在的黑斑羚、Fot1的基因组以及曲霉中Mariner、Gypsy、Helitron的蛋白序列进行BLAST,发现他们的序列几乎相同,这意味着他们没有被损坏[36].Coleman(2009)等人发现在大型茄病镰刀菌的基因组中还包含三个谱系特异性,以及TE丰富的染色体,但与尖孢镰刀菌不同的是其没有丰富的C到T突变的证据.对Gypsy元件进行调整,能够确定多个转换的存在,主要集中在相同的CpA和CpG二核苷酸区,约4%的基因组受此影响.串珠镰刀菌和禾谷镰刀菌因其基因组较小在类似的核苷酸环境下呈现出一种GC比例较少甚至衰竭的表象,但在串珠镰刀菌中,对CpG的突变稍微优先于CpA[37].Ma(2010)等人所在的研究组通过对尖孢镰刀菌、串珠镰刀菌、禾谷镰刀菌基因组的分析,发现在它们的基因组中转座元件所占的比例分别是3.98%,0.14%和0.03%,但每个菌种中总的重复DNA约是这些比例的7倍.尖孢镰刀菌中转座元件所占的比例之所以大,是因为其基因组具有四个额外的与其他两个物种没有共线性的染色体,这些染色体包含了大量的TEs和一些可以在混合培养中转移到其他菌株的谱系特异性致病基因[38].目前已对尖孢镰刀菌复合体的成员进行了广泛研究,并一致认为其繁殖方式是无性,这一结论符合只有极少数TEs区能够显示RIP迹象的事实.
2.2.4 曲霉属
曲霉属在自然界分布极广,是引起多种物质霉腐的主要微生物之一,主要包括米曲霉、黑曲霉、黄曲霉、烟曲霉、构巢曲霉等.其中黄曲霉具有很强毒性,绿色和黑色的具有很强的酶活性,如黑曲霉可以用来生产柠檬酸.以米曲霉为主的霉菌,大都和酒、豆酱、酱油等的酿造有密切关系.因此对曲霉属中RIP的研究有利于更好地了解曲霉属,从而避开那些有害的,善用那些对人有利的菌种.
Neuveglise(1996)研究组首次在烟曲霉中发现了具有RIP特性的逆转录转座子Afut1;Nielsen(2001)等人通过实验在曲霉菌属的反转录转座子Dane1和Dane2观察到了C-G到T-A的转换;Clutterbuck(2004)等人通过对构巢曲霉基因组中的五个MATE转座子进行研究,证实了构巢曲霉重复序列中的胞嘧啶突变不如粗糙脉孢菌中的活泼,但进一步研究发现,构巢曲霉中还存在着许多只有426个碱基对的非独立性元件hAT-N1,这些小元件主要集中在CpG和CpA区,部分CpT区也有着数量可观的转换[39].Montiel(2006)等人开展了对真菌曲霉属中DNA甲基化的探究,从黄曲霉基因组中发现了一段具有多拷贝的并与米曲霉基因组存在同源性的核苷酸序列,这些序列显示RIP特性[40].Clutterbuck(2008)等又发现构巢曲霉和烟曲霉的光谱分裂和胞嘧啶耗竭是相似的,虽然它们各自包含一套独特的TES,并对构巢曲霉和烟曲霉基因组中的这些TEs进行了系统编目[41].目前烟曲霉中已发现的具有RIP特性的转座因子有:Afut1,Aft2和Aft1.构巢曲霉包括:Dane和MATE因子.通过对编码构巢曲霉和烟曲霉TE的蛋白质序列进行BLAST,发现在这两个物种中发生转换最频繁的区域是CpG,CpA二核苷酸也经常发生,但烟曲霉与构巢曲霉相比含有较多的富AT片段[42].经实验证明,构巢曲霉具有有性周期,但烟曲霉更倾向于无性繁殖,尽管有证据显示其具有潜在的有性繁殖倾向(Dyer etal.2003; Paoletti.2005).
Machida(2005)等人发现,米曲霉基因组的重复序列中存在着许多富含AT的片段,而这些片段中23%的重叠区域确定包含有TEs,这一事实与富含AT的DNA片段容易发生突变相关联,但是这些重叠序列一般都不太长[43].同年,Galagan等人发表报道称米曲霉中转座元件和重复序列约占整个基因组的1.4%,因此,尚存在还没有被发现的能显示RIP突变的其他转座子.Montiel(2006)在米曲霉转座子Fot1中发现了RIP突变的证据,但未检测到甲基化的结果;在米曲霉中CpA是最经常发生突变的,对RIP而言, CpA过渡到TpA被认为是增加了引入终止密码子TAA和 TAG的机会.这种情况是单独的突变导致的基因失活,无甲基化的参与[40].Ogasawara(2009)描述了工业米曲霉菌株中至少16种Tc1/Mariner元件明显不存在突变序列,这说明在米曲霉中胞嘧啶甲基化进而突变为胸腺嘧啶的概率是非常小的[44].
Braumann(2007)等人在产黄青霉中发现多个受RIP影响的TE序列.Braumann(2008)等通过对黑曲霉和产黄青霉具有RIP遗迹的转座子序列进行研究发现,产黄青霉中的RIP受多个突变序列的影响;而黑曲霉仅限于少数的序列,并且RIP只发生在开放阅读框已被中断了的转座子序列中,这种现象在其他真菌中尚未被报道.另外,他们还在工业黑曲霉CBS-513.88菌株中发现RIP状突变的ANiTa家族,但同样这个家族也是由ATCC-1015序列上几个单独的LTR代表的.在CBS-513.88序列上有许多近乎相同而且显然未突变的拷贝,而且显示出对核苷酸突变的偏好性:CpG> CpA> CpC> CpT[45].虽然黑曲霉和产黄青霉已被证明是无性繁殖,但它们可能存在一种未被发现的有性阶段,或具有有性生殖的潜力.
2.2.5 十字花科小球腔
Idnurm(2003)通过实验证实,在十字花科小球腔中存在RIP特性[46];Attard(2005)等人在十字花科小球腔中发现了类似反转录转座子的Pholy区域,它能够显示RIP的特性,如在重复序列中能够发生C-G到T-A的突变[47].Isabelle(2009)等研究了十字花科小球腔中毒力和RIP的关系,发现RIP可作为十字花科小球腔中毒力进化的一种替代机制[48].Rouxel(2011)等报道了植物病原子囊菌十字花科小球腔的基因组序列和表征蛋白质效应,发现在叶黄斑部位的基因组有一个不寻常的鸟嘌呤和胞嘧啶交替的二分结构,和富含AT的同质核苷酸组成的区域.富含AT的区域有1/3的基因组包含效应物基因和转座元件,这两者都受RIP的影响[49].
3 观点和结论
重复诱导点突变(RIP)广泛存在于各种真菌中已经是毋庸置疑的事实,起初认为它只存在于有性周期,后来发现在营养细胞中也存在RIP特性,而且不同的真菌其RIP特性均有所不同.通过对真菌RIP的研究,对真菌基因组防御功能、基因表达的调控以及真菌系统进化有了更深层次的了解.虽然基因重复机制已被认为是生物体产生新基因的主要手段,但目前在一些过程上还存在着比较大的争议:如基因复制的程度以及产生有益突变的频率,重复基因的丢失以及新功能的演变速度等.
转座因子转座是真核生物基因组进化的主要动力之一.Felix Horns(2012)等人对担子菌类转座元件中的RIP进行了系统的研究.通过对真菌转座子基因组的研究,逐渐扩展了有关基因组变异和演化方面的知识,而且真菌基因组序列数据的日益普及提供了前所未有的机会去探索和理解转座子的分布、功能以及多样性[50].从上文的介绍中可以获悉:转座因子是真核基因组的基本组成部分,是促使基因组防御系统发展的主要推动力,并且可以以各种方式对基因组可塑性和演变作贡献;RIP是一个同源依赖的基因组防御机制,通过RIP机制,真菌在减数分裂过程中使多拷贝DNA发生突变,能够最大限度地使转座子失去活性,导致基因沉默,从而保持基因组的稳定性.虽然RIP导致重复序列沉默阻碍了基因加倍化使其失去进化产生新基因的机会,但在粗糙脉孢菌中单轮RIP相当温和,加倍化基因的一个或多个拷贝不仅能够维持其旧的功能,而且还能够产生新的等位基因.由此引发的一个重要问题就是粗糙脉孢菌的这一特性是如何实现的.如前所述,在进化的过程中这种重复最终会被RIP灭活,但是如果这对重复基因中的一个丢失(由于异位拷贝的隔离或减数分裂前串联重复序列的删除所导致),剩下的基因将不再受RIP影响.因此,短暂加倍的基因通过RIP可在群体水平上增加基因多样性[28].
到目前为止,真核生物基因组进化的机制,自私DNA的影响和基因组防御机制有许多还尚待了解,随着粗糙脉孢菌全基因组序列测序成功,它可以作为一种模式物种来更好地应对这些问题.例如,在实验室中构建野生型菌株的突变体,进而研究突变型菌株与进化之间的关系.此类实验的结果有助于在近代进化史中找出那些与粗糙脉孢菌基因组重排最接近的近缘种.综上所述,对真菌中RIP进行深入研究,既有助于阐明真菌的系统进化关系,也有利于分析真菌的转基因沉默现象.
参考文献:
[1]Selker E U,Cambareri E B,Jensen B C,et al. Rearrangement of duplicated DNA in specialized cells ofNeurospora [J].Cell, 1987, 51(5): 741-752.
[2]Galagan J E, Selker E U. RIP: the evolutionary cost of genome defense [J].Trends Genet, 2004, 20(9): 417-423.
[3]Selker E U. Premeiotic instability of repeated sequences inNeurosporacrassa[J].Rev Genet, 1990, 24(1): 579-613.
[4]Watters M K, Randall T A, Margolin B S, et al. Action of repeat-induced point mutation on both strands of a duplex and on tandem duplications of various sizes inNeurospora[J].Genetics, 1999, 153(2):705-714.
[5]Cambareri E B, Singer M J, Selker E U. Recurrence of repeat-induced point mutation (RIP) inNeurosporacrassa[J].Genetics, 1991, 127(4):699-710.
[6]Cambareri E B, Jensen B C, Schabtach E, et al. Repeat-induced G-C to A-T mutations inNeurospora[J]. Science, 1989, 244(4912):1571-1575.
[7]Irelan J T, Hagemann A T, Selker E U. High frequency repeat-induced point mutation (RIP) is not associated with efficient recombination inNeurospora[J]. Genetics, 1994, 138(4):1093-1103.
[8]Borkovich K A, Alex L A, Yarden O, et al. Lessons from the genome sequence ofNeurosporacrassa: tracing the path from genomic blueprint to multicellular organism [J]. Mol. Biol. Rev, 2004, 68(1):1-108.
[9]Crouch J A, Glasheen B M, Giunta M A, et al. The evolution of transposon repeat-induced point mutation in the genome ofColletotrichumcereale: reconciling sex, recombination and homoplasy in an “asexual” pathogen [J].Fungal Genet Biol., 2008, 45(3): 190-206.
[10]Ikeda K, Nakayashika H, Kataoka T, et al. Repeat-induced point mutation (RIP) inMagnaporthegrisea: implications for its sexual cycle in the natural field context [J]. Mol. Biol., 2002, 45(5):1355-1364.
[11]Freitag M, Williams R L, Kothe G O, et al. A cytosine methyltransferase homologue is essential for repeat-induced point mutation inNeurosporacrassa[J]. Proc. Natl. Acad. Sci. USA., 2002, 99(13):8802-8807.
[12]Mautino M R, Rosa A L. Analysis of models involving enzymatic activities for the occurrence of C/T transition mutations during repeat-induced point mutation (RIP) inNeurosporacrassa[J]. J. The. Biol., 1998, 192(1):61-71.
[13]Machida M, Asai K, Sano M, et al. Genome sequencing and analysis ofAspergillusoryzae[J]. Nature, 2005, 438(4):1157-1161.
[14]Foss H M, Selker E U. Efficient DNA pairing in aNeurosporamutant defective in chromosome pairing [J]. Mol. Gen. Genet., 1991, 231(1):49-52.
[15]Bowring F J, Catchesaid D E. The effect of rec-2 on repeat induced point-mutation (RIP) and recombination events that excise DNA sequence duplications at the his-3 locus inNeurosporacrassa[J]. Genetics, 1993, 23(5-6):496-500.
[16]Kouzminova E A, Selker E U. dim-2 encodes a DNA methyltransferase responsible for all known cytosine methylation inNeurospora[J].EMBO J, 2001, 20(15): 4309-4323.
[17]Watters M K, Randall T A, Margolin B S, et al. Action of repeat induced point mutation on both strands of a duplex and on tandem duplications of various sizes inNeurospora[J]. Genetics, 1999, 153(2): 705-714.
[18]Tamaru H, Selker E U. Synthesis of signals fordenovoDNA methylation inNeurosporacrassa[J].Mol. Cell. Biol, 2003, 23(7):2379-2394.
[19]Singer M J, Marcotte B A, Selker E U. DNA methylation associated with repeat induced point mutation inNeurosporacrassa[J]. Mol. Cell, Biol., 1995, 15(10):5586-5597.
[20]Aramayo R, Selker E U.Neurosporacrassa, a Model System for Epigenetics Research [J]. Cold Spring Harbor Biology, 2013, 5:a017921.
[21]Selker E U, Stevens J N. DNA methylation at asymmetric sites is associated with numerous transition mutations [J]. Proc. Natl.Acad. Sci. USA., 1985, 82(23):8114-8118.
[22]邱立友,余翠,戚元成,等.真菌表观遗传学研究进展[J]. Chinese Journal of Cell Biology, 2009, 31(2): 212-216.
[23]Selker E U. Pinstability of repeated sequences inNeurosporacrassa[J].Rev Genet, 1990, 24(1): 579-613.
[24]Cambareri E B, Aisner R, Carbon J. Structure of the chromosome VII centromere region inNeurosporacrassa: degenerate transposons and simple repeats [J]. Mol. Cell. Biol., 1998, 18(9): 5465-5477.
[25]Selker E U, Cross S H, Murphy J G, et al. The methylated component of theNeurosporacrassagenome [J].Nature, 2003, 422:893-897.
[26]Borkovich K A, Alex L A, Yarden O, et al. Lessons from the genome sequence ofNeurosporacrassa: tracing the path from genomic blueprint to multicellular organism [J]. Mol. Biol. Rev., 2004, 68(1):1-108.
[27]Galagan J E, Calvo S E, Borkovich K A, et al. The genome sequence of the filamentous fungusNeurosporacrassa[J]. Nature, 2003, 422:859-868.
[28]Selker E U. Epigenetic phenomena in filamentous fungi: useful paradigms or repeat-induced confusion [J]. Trends Genet, 1997, 13(8): 296-301.
[29]Clutterbuck A J. Genomic evidence of repeat-induced point mutation (RIP) in filamentous ascomycetes [J].Fungal Genetics and Biology, 2011, 48(3):306-326.
[30]Ikeda K I, Nakayashika H, Kataoka T, et al. Repeat-induced point mutation (RIP) inMagnaporthegrisea: implications for its sexual cycle in the natural field context [J]. Mol. Biol., 2002, 45(3):1355-1364.
[31]Shiflett A M, Enkerli J, Covert S F, et al. Nht2, a copia LTR retrotransposon from a conditionally dispensible chromosome inNectriahaematococca[J]. Genetics, 2002, 41(2):99-106.
[32]Graia F, Lespinet O, Rimbault B, et al. Genome quality control: RIP (repeat-induced point mutation) comes toPodospora[J].Mol. Biol., 2001, 40(3): 586-595.
[33]Hamann A, Feller F, Osiewacz H D. The degenerate DNA transposon Pat and repeat-induced point mutation (RIP) inPodosporaanserine[J]. Mol. Gen. Genet., 2000, 263(6): 1061-1069.
[34]Arnaise S, Zickler D, Bourdais A, et al. Mutations in mating-type genes greatly decrease repeat-induced point mutation process in the fungusPodosporaanserine[J]. Fungal Genet. Biol., 2008, 45(2):207-220.
[35]Hua-van A, Langin T, Daboussi M J. Evolutionary history of the impala transposon inFusariumoxysporum[J]. Mol. Biol. Evol., 2001, 18(10):1959-1969.
[36]Daboussi M J, Daviere J M, Grazione S, et al. Evolution of the Fot1 transposons in the genus Fusarium: dicontinuous distribution and epigenetic inactivation [J]. Mol. Biol. Evol., 2002, 19(4): 510-520.
[37]Coleman J, Rounsley S D, Wasmann C, et al. The Genome ofNectriahaematococca: contribution of supernumerary chromosomes to gene expansion [J]. PloS Genet., 2009, 5:1000618.
[38]Ma L J, Borkovich K A, Coleman J, et al. Comparative genomics reveals mobile pathogenicity chromosomes inFusarium[J]. Nature, 2010, 464: 367-373.
[39]Clutterbuck A J. MATE transposable elements inAspergillusnidulans: evidence of repeat-induced point mutation [J]. Fungal Genet, Biol., 2004, 41(3): 308-316.
[40]Montiel M D, Lee H A, Archer D B. Evidence of RIP (repeat-induced point mutation) in transposase sequences ofAspergillusoryzae[J].Fungal Genet, Biol., 2006, 43(6):439-445.
[41]Clutterbuck A J, Kapitonov V, Jurka J, et al. Transposable elements and repeat induced point mutation inAspergillusnidulans,AspergillusfumigatusandAspergillusoryzae[J].Fungal Genet, Biol., 2008, 36(2):343-355.
[42]Hey P, Robson G, Birch M, et al. Characterization of Aft1 a Fot/Pogo type transposon ofAspergillusfumigatus[J].Fungal Genet, Biol., 2008, 45(6):117-126.
[43]Machida M, Asai K, Sano M, et al. Genome sequencing and analysis ofAspergillusoryzae[J]. Nature, 2005, 438:1157-1161.
[44]Ogasawara H, Obata H, Hata Y, et al. Crawler, a novel Tc1/Mariner-type transposable element inAspergillusoryzaetransposes under stress conditions [J]. Fungal Genet, Biol., 2009, 46(6-7): 441-449.
[45]Braumann I, Van- den B M, Kempken F. Repeat induced point mutation in two asexual fungi,AspergillusnigerandPenicilliumchrysogenum[J].Cur. Genet., 2008, 53(5): 287-297.
[46]Idnurm A, Howlett B J. Analysis of loss of pathogenicity mutants reveals that repeat-induced point mutations can occur in theDothideomyceteLeptosphaeriamaculans[J]. Fungal Genet, Biol., 2003, 39(1):31-37.
[47]Attard A, Gout L, Ross S, et al. Truncated and RIP-degenerated copies of the LTR retrotransposon Pholy are clustered in a pericentromeric region of theLeptosphaeriamaculansgenome [J]. Fungal Genet, Biol., 2005, 42(1):30-41.
[48]Isabelle F, Simon R, Hortense B, et al. Repeat-Induced Point Mutation (RIP) as an Alternative Mechanism of Evolution Toward Virulence inLeptosphaeriamaculans[J]. The American Phytopathological Society, 2009, 22(8):932-941.
[49]Rouxel T, Grandaubert J, Hane J K, et al. Effector diversification within compartments of theLeptosphaeriamaculansgenome affected by Repeat-Induced Point mutations [J]. Nature, 2011, 15(10):1 -10.
[50]Horns F, Petit E, Hood M E, et al. Patterns of Repeat-Induced Point Mutation in Transposable Elements ofBasidiomyceteFungi [J].Genome Biol. Evol., 2012, 4(3):240-247.

