HLAMatchmaker软件分析Eplets的配型在临床肾移植配型中的应用*
张 行,潘晓东,徐慧英,吴存造,蔡 勇,夏 鹏,郑少玲,杨亦荣,陈必成,△
(1.温州医科大学附属第一医院外科实验室,2.移植中心,浙江温州325000)
HLA(human leukocyte antigen)是人类最复杂的基因多态性系统,同种异体移植术前需进行供受者间HLA配型。HLA抗原零错配能获得较高的移植成功率和较长的术后生存期,但是回顾性研究中发现某些存在多个HLA抗原错配的移植亦能取得较好的效果[1]。因各 HLA间存在交叉反应组抗原(CREGs),具有相同或相似的抗原决定簇结构或氨基酸残基,组内有较好的匹配,但也可产生此组抗供者抗体的交叉反应。在此基础上的CREGs配型策略,认为可接受性错配抗原(permissible mismatched antigen)为零错配,扩大了可接受的供体选择。但CREGs的缺点是根据经验结果总结,不能进行精确的评估可接受错配和不可接受的错配。
HLAMatchmaker(Duquesnoy教授基于微软公司的 Excel软件开发,http://www.hlamatchmaker.net/)软件可以通过计算机比对HLA的接受程度,它基于分子免疫学的以下两个原理:(1)每一个HLA分子是由一系列能各自产生特异性抗体的Eplets(决定抗原决定簇特异性的关键氨基酸组成,指位于HLA分子表面3.0~3.5埃(1埃 =1×10-8cm)大小的空间多态性位点);(2)移植受者不会产生针对自身HLA Eplets的抗体。为此设计的HLAMatchmaker可计算供受者间HLA氨基酸序列Eplets的差异,能够定量地分析出Eplets错配数,给出不兼容的错配位点,找到可接受的错配(acceptable mismatch)从而寻找合适的供者,提高移植术后受者的生存率[2]。运用此软件分析可避开基因错配位点,让群体反应性抗体高敏感性受者亦能接受移植[3]。本研究中我们回顾性对239例移植受者运用3种现有配型方法即HLA六抗原无错配配型、交叉反应组配型和基于HLAMatchmaker软件的Eplets配型,分析 Eplets配型优越性。
1 对象与方法
1.1 研究对象
2011年1月份至2012年12月份期间在温州医科大学第一医院移植中心进行239例肾移植的受者,其中男性 169例(70.7%),女性 70例(29.3%),最大年龄 70,最小年龄17岁,平均年龄(45.23±11.70)岁,O型103例,A型 66例,B型 48例,AB型22例,供者共139例。移植供受者的ABO血型匹配。PRA(panel reactive antibody)≥10%阳性病人17例(7.11%)。
1.2 HLA检测方法
采用美国 One Lambda公司的 Micro SSP HLA Class and ABDR DNA Typing Tray通过低分辨率的顺序特异引物聚合酶链反应(polymerase chain reaction with sequence-specific primers,PCR-SSP)方法检测出HLA的配型,用 HLAMatchmaker中的(4-digit Convert)将血清学的HLA转化成基因型的HLA,另外有部分标本采用GenDX公司Sanger法基因测序试剂盒检测HLA的基因型。
1.3 配型方法
1.3.1 HLA配型 采用美国器官分配联合网(United Network for Organ Sharing,UNOS)在 1990年制定的HLA六抗原无错配标准(Zero HLA-A,B,DR Antigen Mismatch,0 Ag Mismatch):供受者HLA存在一个抗原错配得一分,抗原错配数累积,分值在0~6之间,分数越低匹配度越高。
1.3.2 CREGs配型 Thompson提出的 HLA氨基酸残基配型,又称交叉反应组配型(HLA-cross reactive groups matching,CREGs)[4]。在此基础上我们进行了得分累积的改进:供受者HLA六抗原位点完全匹配为0,不匹配但如属于同一交叉反应组则得分为0.5,此外的不匹配为1分,分值在0~6之间,分数越低匹配度越高。
1.3.3 HLAmatchmaker软件配型 供受者 HLA-Ⅰ基因型使用(ABC Matching for 500 donor-recipient pairs)和 HLA-Ⅱ基因型(DRDQMatching for 500 donorrecipient pairs),HLAmatchmaker软件对 HLA-Ⅰ 和HLA-Ⅱ分别进行配对,寻找出错配数及错配Eplets,在Eplets表达式中,如66 RNM代表一个天冬氨酸,在氨基酸序列第66位用字母N表示,而相邻的第65位精氨酸和第67位蛋氨酸分别用字母R和M表示。如果前后的氨基酸同中间的氨基酸相同,则表达式中不再重复显示,如9F代表氨基酸序列第9位的苯丙氨酸,62QE代表氨基酸序列第62位的甘氨酸和第63位为谷氨酸。然后在通过EXCEL合计错配数,分数越低匹配度越高[5]。
1.4 统计学分析
三种方法分组人数之间比较采用多分类列表概率分布检验计算,三种方法所得错配数通过两两比较采用秩转换结合随机取组设计的方差分析检验计算。实验数据所有统计均由SPSS 16.0软件完成。
2 结果
2.1 HLAMatchmaker结果显示形式
在HLAMatchmaker软件中输入相对应的供受者的基因型,HLAMatchmaker软件会自动分析供受者之间Eplets错配的数量和位点。如:受者的HLA-Ⅰ的基因型为 A*0301、A*3001、B*1302、B*2705,而供者 HLA-Ⅰ的基因型 A*0201、A*3001、B*1302,根据软件分析结果显示,共计存在11个Eplets错配,而这些错配分别用不同位点的氨基酸表示62GE65RKA70AHS116Y127K142MT144TKH151HV207 S184A193AV。另外运用相同的方法获得该供受者HLA-ⅡEplets的错配(表 1)。
2.2 三种方法错配数得分
239例肾移植的三种配型方法所获得的错配情况,其中6例在表2中列出,如Case 3中传统的HLA抗原配型组显示HLA-Ⅰ、Ⅱ分别为3分和2分,合计为5分;在CREGs配型组中,HLA-Ⅰ、Ⅱ分别存在2分和 1.5分,合计 3.5分;HLAMatchmaker软件配型的结果显示,HLA-Ⅰ、Ⅱ均为18分,合计36分。三种方法计算的239例供受者配型计错配数得分,秩转换并经SPSS软件统计计算,显示 HLAMatchmaker软件配型的方法与传统HLA配型、CREG交叉反应抗原组配型有显著性的差异(表2,P<0.01)。
2.3 三种配型方法的分布
HLA对239供受者HLA-Ⅰ、Ⅱ抗原配型所得分数,根据HLA抗原错配结果将受者分为7组,分别:0个 Mismatch(MM)组 1例;1个 MM组 2例;2个MM组16例;3个MM组31例;4个MM组81例;5个MM组72例;6个MM组36例。
CREGs对239供受者HLA-Ⅰ、Ⅱ抗原配型所得分数,0个MM组1例;0<n≤1个MM组4例;1<n≤2个MM组为27例;2<n≤3个MM组为73例;3<n≤4个MM组为89;4<n≤5个MM组为39例;5<n≤6个MM组为6例(表3)。
HLAMatchmaker对239供受者 HLA-Ⅰ、Ⅱ抗原配型所得分数,根据 Valentini[6]建立了一套相对完善的标准,0个 Eplets Mismatch(EMM)组1例;0<n≤10个EMM组10例;10<n≤20个 EMM组43例;20<n≤30个 EMM组73例;30<n≤40个EMM组66例;50<n个EMM组8例(表3)。

Tab.1 Examples of donor-recipients HLA-Ⅰ mismatch with HLAMatchmaker at the Eplets level

Tab.2 Examples of donor-recipients HLA the number of mismatch with three methods

Tab.3 Results of HLA matching,CREGmatching and HLAMatchmaker typing matching in grouping
根据表3的结果将 HLA、CREGs和 HLAMatchmaker三种方法所得分数结果按照3个组别进行分组,分别为低错配组、中错配组和高错配组(图1)。在低错配组中,HLA、CREG和 HLAMatchmaker分别为 19(7.9%)、32(13.4%)、54(22.6%);中错配组中分别为 112(46.9%)、162(67.7%)、139(58.1%);高错配组中,分别为 108(45.2%)、45(18.8%)、46(19.2%)。从结果上分析,三种方法存在显著差异(χ2=66.68 P<0.01),HLAMatchmaker配型方法相比较其他两种方法在0~2低错配组别的人数较明显的增加,而3~4中错配组别HLAMatchmaker配型方法的人数介于传统HLA和CREGs两种配型方式之间,5~6高错配的高错配组别 HLAMatchmaker人数与CREGs方法人数相近,且这两种方法的人数明显少于传统的HLA配型方法。从表4中可以看出,HLAMatchmaker相对于其他两种方法可以明显提高供受者的相配率。

Tab.4 Comparison among three methods of HLA matching group in 239 donor-recipients
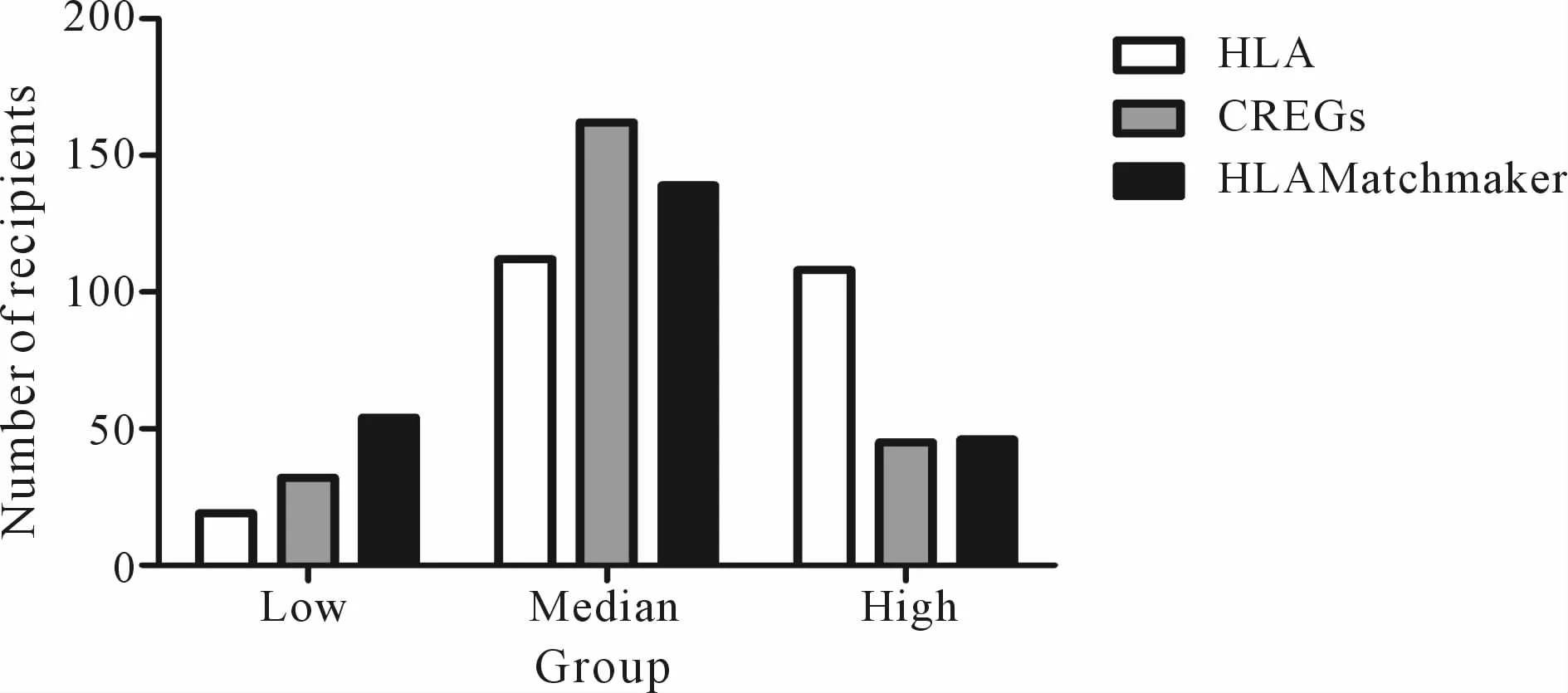
Fig.1 Number of recipients gruoping by low-high MM
3 讨论
器官移植中,不管国际或者国内均普遍采用HLA六抗原无错配为标准的配型,但由于HLA在基因上的高度多态性及复杂的蛋白空间结构,供受者间存在HLA-Ⅰ、Ⅱ抗原零错配的概率很低,致使患者需等待较长时间才得以移植手术。但是随着移植免疫学的发展,提出了CREGs的配型方法,是指特异性血清学反应格局的公共抗原为同一组零错配,该方法通过HLA抗原分子公共抗原表位在一定程度上提高了HLA无错配和低错配的几率,并且在之前的研究中显示出了较好的临床应用价值,CREGs抗原相合的肾移植术后效果要优于CREGs抗原不相合的[7]。但是CREGs同样存在缺陷,亦无法精确地预测HLA分子的立体化学空间结构和氨基酸序列间的差异,依然将配型停留在抗原水平,而现在的HLA分型则大多开始采用分子生物学的技术,如PCR和测序等,使得HLA分型的准确性和精确度都得到了较大的提高。而根据CREGs的这种配型方式已无法满足HLA基因型多样性和对新基因型的配型需求。此外,生物信息学的迅速发展,使蛋白质三维结构模拟的技术更加成熟,但是一直缺乏一种能在空间立体结构层面比较HLA分子的配型方法,为此Duquesnoy教授引入了Eplets这样全新的概念并开发出HLAmatchmaker软件以解决此问题。而我们在此软件的基础上,首次将该软件应用于临床中肾移植手术前的配型和对移植术后的抗体预判。
HLAMatchmaker软件配型的方法则是基于E-plets位点间的比对,给出不兼容的错配。本研究将本中心两年间的239例次肾移植受者,按照传统HLA配型、CREGs配型和基于HLAMatchmaker软件配型进行比较,HLAmatchmaker配型方法的低错配人数明显少于其他两种方法,并有显著性差异(P<0.01),说明 HLAMatchmaker能明显提高相配率。同时HLAMatchmaker软件能在抗原结构相容性上更精确地发现错配,即HLA抗原型相同,也可能存在个别Eplets的不同;但其优势是在供受者HLA分型不同时对比Eplets组合找到最少的错配。由于个体携带的Eplets库差异,即使供、受者HLA 6抗原型完全不相同,亦有找到低错配的可能;此外,在传统HLA配型和 CREGs配型认为较好的配率,但是 HLAMatchmaker却能筛选出较多存在的错配Eplets,反之亦然。从结果中显示三种方法的错配存在着明显的统计学差异(P<0.01)。采用 HLAMatchmaker软件的配型方法不但扩大了供体选择的范围同时也更容易找到优质供者。
在以往的研究中运用HLAMatchmaker软件进行Eplets配型可显著降低肾移植术后的致敏性。Duquesnoy教授回顾性研究了1987~1999年间美国器官分配和联合网络(UNOS)和欧洲器官库(Eurotransplant)数据中的HLA-DR抗原零错配的肾移植情况下,结果发现肾移植受者HLA-A、B在0~4个E-plet错配的情况下5年移植物生存率为(76.30±3.11)%,而传统 HLA-A、B抗原零错配的生存率为76.8%,两者未显示出差异,说明了即使存在少数的EMM的情况下同HLA-A、B抗原零错配长期生存率基本一致,而且此结果在不同肤色种族及是否为高致敏受者并不受影响。同时HLAMatchmaker的配型方式能够显著降低肾移植术后的致敏性,改善移植物的存活率[2,8]。此外亦能根据每个受者那些EMM(Eplets mismatch)位点再次通过 HLAMatchmaker系列的软件进行直接分析得出受者移植术后预判具体HLA特异性抗体及其产生的几率[9,10]。以上均显示了HLAMatchmaker软件在移植配型有非常突出的优势和广泛应用的潜力。
使用HLAMatchmakers软件来进行配型,不但可以处理供者和大量潜在受者之间的配型,提高工作效率。同时该方法扩大了移植受者的可接受群体,有机会考虑影响移植结果的其他因素。由于完全基于计算机的计算和统计,避免了传统HLA配型和使用CREGs配型容易造成的人为误配。HLA配型是决定移植肾和受者存活的重要因素,应用 HLAMatchmaker软件配型的方法,能够有效提高受者和肾的存活率,同时降低肾移植后急性排除的发生率。良好的配型可减少免疫抑制剂的用药量,有效避免药物浓度过高引起的毒性反应,也节省了费用。在T细胞免疫可被控制的移植时代,HLAMatchmaker软件配型符合精确、高效的要求,并对避免和分析体液免疫提供了全新的工具。
[1] Duquesnoy RJ.HLAMMATCHMAKER:a molecularly based donor selection algorithm for highly alloimmunized patients[J].Transplant Proc,2001,33(1-2):493-497.
[2] Duquesnoy RJ,Takemoto S,de Lange P,et al.HLAmatchmaker:a molecularly based algorithm for histocompatibility determination.III.Effect of matching at the HLA-A,Bamino acid triplet level on kidney transplant survival[J].Transplantation,2003,75(6):884-889.
[3] Dankers MK,Witvliet MD,Roelen DL,et al.The number of amino acid triplet differences between patient and donor is predictive for the antibody reactivity against mismatched human leukocyte antigens[J].Transplantation,2004,77(8):1236-1239.
[4] Thompson JS,Thacker LR.CREGmatching for first cadaveric kidney transplants(TNX)performed by SEOPFcenters between October 1987 and September 1995.Southeastern Organ Procurement Foundation[J].Clin Transplant,1996,10(6 Pt 2):586-593.
[5] 张 行,潘晓东,陈文伟,等.HLAMatchmaker软件在HLA配型中的应用原理及前景[J].中华移植杂志(电子版),2013,7(1):42-47.
[6] Valentini RP,Nehlsen-Cannarella SL,Gruber SA,et al.Intravenous immunoglobulin,HLA allele typing and HLAMatchmaker facilitate successful transplantation in highly sensitized pediatric renal allograft recipients[J].Pediatr Transplant,2007,11(1):77-81.
[7] Crowe DO.The effect of cross-reactive epitope group matching on allocation and sensitization[J].Clin Transplant,2003,17(Suppl 9):13-16.
[8] Mizutani K,Terasaki P,Hamdani E,et al.The importance of anti-HLA-specific antibody strength in monitoring kidney transplant patients[J].Am J Transplant,2007,7(4):1027-1031.
[9] Nambiar A,Duquesnoy RJ,Adams S,et al.HLAMatchmaker-driven analysis of responses to HLA-typed platelet transfusions in alloimmunized thrombocytopenic patients[J].Blood,2006,107(4):1680-1687.
[10]Duquesnoy RJ.Antibody-reactive epitope determination with HLAMatchmaker and its clinical applications[J].Tissue Antigens,2011,77(6):525-534.

