牛源性Escherichia coli O157∶H7分离鉴定、耐药性及耐药基因分析
刘云,卢婷,张力国,赵明礼,孙冬冬,孙思超,梁伟峰,周国辉,师东方,于力
(1.东北农业大学动物医学学院,哈尔滨 150030;2.中国农业科学院哈尔滨兽医研究所,哈尔滨 150001)
牛源性Escherichia coli O157∶H7分离鉴定、耐药性及耐药基因分析
刘云1,卢婷1,张力国1,赵明礼1,孙冬冬1,孙思超1,梁伟峰1,周国辉2,师东方1,于力2
(1.东北农业大学动物医学学院,哈尔滨 150030;2.中国农业科学院哈尔滨兽医研究所,哈尔滨 150001)
了解某牛场犊牛群发腹泻后继发胸膜炎、腹膜炎并造成犊牛大量死亡的主要病原菌及其耐药性情况。采集病死犊牛肺、腹膜和肠系膜等脏器,进行病原菌分离、鉴定,并对分离的主要致病菌株进行药敏试验及耐药基因检测。结果表明,引起此次犊牛腹泻及胸膜炎、腹膜炎的主要致病菌为E.coli O157:H7;药敏试验结果表明分离菌对青霉素、四环素和氨苄西林等12种抗生素耐药,对头孢哌酮、头孢他啶、丁胺卡那等8种抗生素敏感,表现为多重耐药。该分离菌同时携带tetB、strB、aadB、aphA、floR和TEM等耐药基因,耐药基因型和耐药表型不完全相同。研究可为E.coli O157:H7的防治和耐药性控制提供依据。
E.coli O157:H7;药敏试验;耐药基因
肠出血性大肠杆菌(EHEC)血清型主要包括E.coliO157∶H7,O157∶NM,O26∶H8,O125∶NM和O45∶H2等,其中以O157∶H7血清型为主,约占90%以上,是引起出血性肠炎主要病原体[1]。1982年,E.coliO157∶H7第一次在被污染的汉堡中分离得到,牛和其他反刍动物(羊和鹿等)已成为自然宿主,其中牛是主要宿主[2]。Sanchez等研究表明,猪也是E.coliO157∶H7的宿主[3]。E.coliO157: H7分泌的志贺菌素能够引起人类疾病,如出血性肠炎(Haemorrhagic colitis,HC)、溶血性尿毒综合症(Haemolytic curaemic syndrome,HUS)和血栓性血小板减少性紫癜(Thrombotic thrombocytopenic pur⁃pura,TTP)等。世界卫生组织已将E.coliO157:H7列为新的食源性病原菌,E.coliO157:H7感染已成为全球性的公共卫生问题[4]。
动物大肠杆菌疾病,如仔猪黄白痢、猪的水肿病、犊牛痢疾及禽类大肠杆菌引起的败血症、腹膜炎、输卵管炎、滑膜炎等,因大肠杆菌种类繁多,又有不同的血清型,同一血清型有不同亚型,其毒力和致病力不一。E.coliO157:H7是引起人出血性和致死性胃肠炎的主要病原之一,但动物关于E.coliO157:H7引发的疾病研究不多。2011年10月,黑龙江省某牛场犊牛出生后出现严重水样腹泻,发病率100%,发病后犊牛陆续死亡。剖检其主要病理变化为心、肝、脾、肺、肾、肠管和腹膜等处均有不同程度出血,出血性胸膜炎、腹膜炎症状明显,临床疑似为大肠杆菌感染。为了解引起该牛场牛发病的病源特性,本试验采集病死牛的病料,肺脏、肠系膜、胸膜及腹膜,进行病原菌分离、鉴定,对其进行药敏试验及耐药基因检测,为进一步研究其生物学特性奠定基础。
1 材料与方法
1.1 材料
1.1.1 菌株
大肠杆菌临床分离株:分离自黑龙江省某牛场病死犊牛肺、腹膜和肠系膜等脏器。采集病料放入冰盒内,12 h内带回东北农业大学动物医学学院外科实验室。大肠杆菌质控菌株ATCC 25922和标准菌株E.coliCICC 21530购自中国工业微生物菌种保藏管理中心。
1.1.2 培养基与试剂
麦康凯琼脂、伊红美蓝琼脂、脑心浸液肉汤(BHI)等培养基及肠杆菌科生化鉴定管等购自杭州天和微生物试剂有限公司;TaKaRa ExTaq(5 U·μL-1)、DL2000 DNA Marker购自TaKaRa公司;所有PCR引物均由上海英俊试剂公司合成。
1.1.3 试验药物
头孢哌酮(CFP)、头孢曲松(CRO)、头孢噻肟(CTX)、头孢他啶(CAZ)、氨苄西林(AM)、青霉素(P)、丁胺卡那(AN)、红霉素(E)、卡比西林(CB)、呋喃妥因(FT)、四环素(TC)、链霉素(S)、庆大霉素(GM)、卡那霉素(K)、氯霉素(C)、利福平(RA)、妥布霉素(TC)、环丙沙星(CIP)、克林霉素(CLI)和复方新诺明(SXT)等20种药敏纸片均购自康泰生物制品有限公司。
1.2 方法
1.2.1E.coli菌株的分离与生化试验
将采集的样品接种于麦康凯培养基,于37℃恒温培养18~24 h,挑取单个光滑、粉红色菌落接种于伊红美蓝培养基,37℃恒温培养18~24 h,挑取具有金属光泽的菌落进行革兰氏染色、镜检,并接种于BHI液体培养基进行增菌。选择生化微量管葡萄糖、乳糖、木糖、甘露糖、麦芽糖、棉子糖、蕈糖、果糖、山梨醇、硝酸盐、VP、卫矛醇、蔗糖、H2S、靛基质和枸橼酸盐进行常规生化检测,置于37℃培养箱内培养24 h,观察结果。
1.2.2E.coli菌株的DNA扩增
分离出的E.coli菌株接种在5 mL BHI液体培养基中,37℃震荡12~18 h。吸取1.5 mL BHI培养基,以10 000 r·min-1离心2 min。菌泥加入100 μL无菌蒸馏水孵育10 min,放置在冰盒上冷却5 min,混合物以10 000 r·min-1离心2 min。移除悬液并于-20℃储存,作为DNA模板用于之后的PCR分析[5]。
1.2.3 分离菌株的鉴定
分离菌株用16S rRNA通用引物[6]进行PCR扩增,扩增反应体系(25 μL):10×buffer 2.5 μL,4 μL dNTPs,0.5 UTaqDNA酶,2 mmol·L-1MgCl2,每个引物1 μL,模板DNA 2 μL,用灭菌蒸馏水补到25 μL,标准菌株E.coliATCC 25922作为阳性对照。扩增产物在1.0%琼脂糖凝胶进行电泳(1×TAE,EB染色,20 min,20 V),凝胶影像用凝胶成像仪进行照相,利用胶回收试剂盒回收产物进行测序,测序结果经Blast比对。
利用编码E.coli菌体抗原O157的基因rfbEO157和编码E.coli结构性鞭毛抗原H7的基因fliCH7检测分离菌株的血清型[7],扩增反应体系(25 μL):模板DNA 2 μL,每个引物1 μL,dNTPs 4 μL,10× Buffer 2.5 μL,Tap酶0.5 μL,用灭菌蒸馏水补足25 μL,标准菌株E.coliCICC 21530作为阳性对照。
1.2.4E.coliO157:H7菌株的药敏试验
使用Mueller-Hinton琼脂通过纸片扩散法(K-B法)实施。将细菌接种于BHI培养基中,置于37℃摇床培养10 h,用生理盐水校正至与0.5麦氏单位标准比浊管相同,在15 min内,用无菌棉签拭子蘸取菌液均匀涂布于Mueller-Hinton琼脂平板整个表面。对头孢哌酮、头孢曲松、头孢噻肟、头孢他啶、氨苄西林、青霉素、丁胺卡那、红霉素、卡比西林、呋喃妥因、四环素、克林霉素、链霉素、庆大霉素、卡那霉素、氯霉素、利福平、妥布霉素、环丙沙星和复方新诺明共20种药物进行药敏试验。
在需氧环境下37℃培养18~24 h,使用E.coliATCC 25922作为质控菌株,通过游标卡尺测量抑菌环直径,并根据NCCLS标准判断分离株对受试抗菌药物的敏感性[8]。
1.2.5 分离菌株的耐药基因检测
根据大肠杆菌对β-内酰胺类、氨基糖苷类、四环素类、链霉素类、磺胺类、喹诺酮类、氯霉素类抗生素的耐药机制,选取SHV、floR、aacA4等14种耐药基因用于分析E.coliO157:H7携带的耐药基因型。
利用提取的分离菌株DNA模板分别对14种耐药基因进行PCR检测。扩增产物在1.0%琼脂糖凝胶进行电泳(1×TAE,EB染色,20 min,120 V),凝胶影像用凝胶成像仪进行照相,反应引物及退火温度见表1。
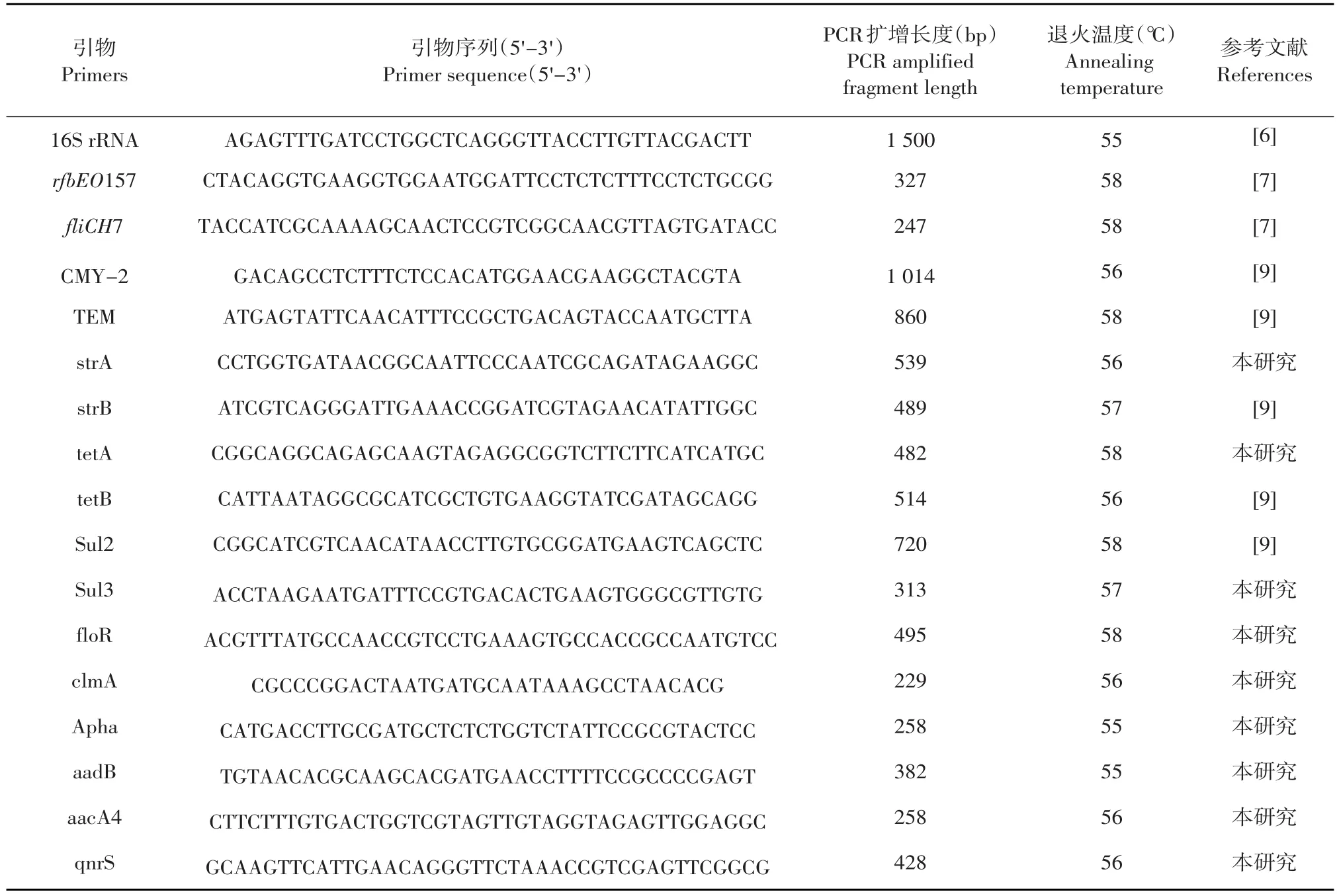
表1 本研究中所使用的引物Table 1 Primers used in the study
2 结果与分析
2.1 菌株分离与生化试验结果
通过麦康凯与伊红美蓝培养基的筛选,分离菌株在麦康凯琼脂培养基上呈圆形,稍隆起的粉红色菌落光滑湿润,边缘整齐。在伊红美蓝平板上呈现出带有金属光泽的紫黑色菌落。革兰氏染色为阴性(见图1)。经微量生化管进行检测(见表2),参照《伯杰细菌鉴定手册》[10],分离菌株符合E. coli菌株的典型生化特征。

图1 分离菌株镜检形态Fig.1 Morphology of isolates in the microscope
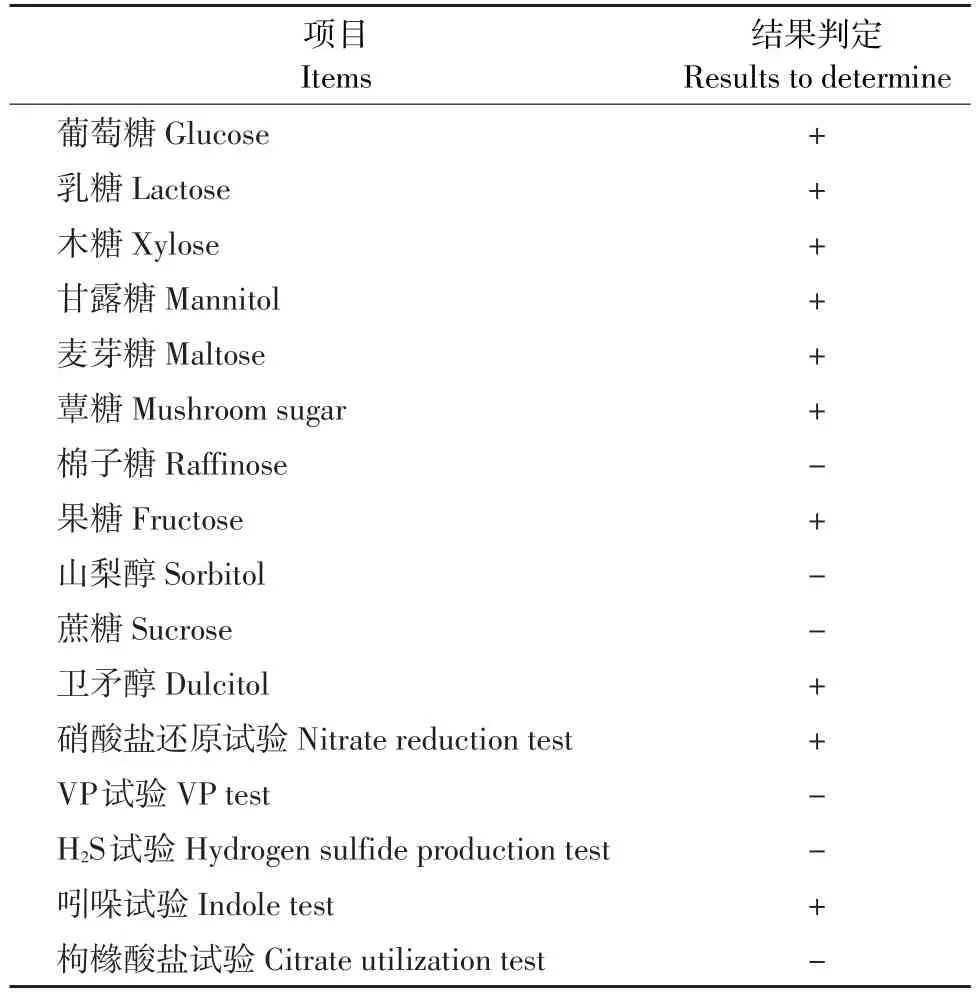
表2 分离菌株的生化试验结果Table 2 Biochemical experimental results of the isolate
2.2 分离菌株的PCR鉴定
分离菌株经16S rRNA通用引物进行扩增,产物测序结果与GenBank中已知E.coli16S rRNA序列同源性达99%,鉴定结果表明该分离菌株为E.coli。通过检测rfbEO157基因与fliCH7基因,证明分离菌株的血清型为O157∶H7(见图2)。
2.3 药敏试验结果
E.coliO157∶H7分离株对抗生素敏感性检测结果见表3。结果可知,分离菌株对头孢他啶、头孢哌酮、庆大霉素、卡那霉素、丁胺卡那、妥布霉素、氯霉素和呋喃妥因共8种抗生素敏感,对青霉素、氨苄西林、卡比西林、头孢噻肟、头孢曲松、环丙沙星、链霉素、红霉素、四环素、复方新诺明、克林霉素和利福平12种抗生素表现为耐药。

图2 分离菌株E.coli O157:H7 rfbEO157基因与fliCH7基因的扩增结果Fig.2 rfbEO157 gene and fliCH7 gene amplificated in E.coli O157:H7 isolate
2.4 分离菌株耐药基因的PCR检测结果
以分离菌株的DNA为模板进行PCR扩增,扩增出tetB、strB、aadB、aphA、floR和TEM耐药基因的特异性条带,未检测出qnrs,sul3,CMY-2,cl⁃mA、strA、aacA4、sul2和tetA耐药基因。PCR扩增凝胶电泳结果见图3。
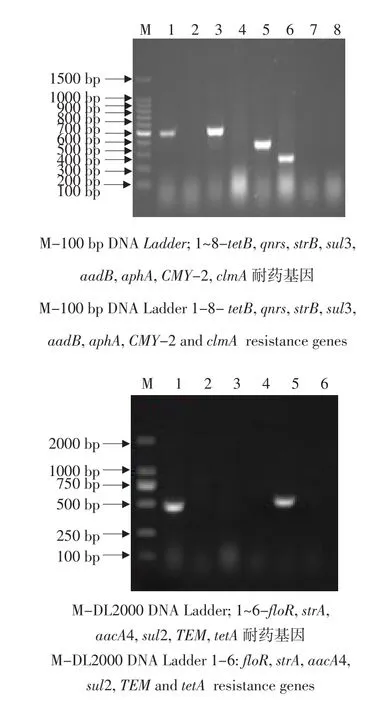
图3 分离菌株E.coli O157:H7耐药基因的扩增结果Fig.3 Resistance genes detected in E.coli O157:H7 isolate
3 讨论
本研究从患有腹泻和胸膜炎犊牛的病料中分离出病原菌,经麦康凯培养基和伊红美兰培养基选择性培养后,再进行革兰氏染色,生化试验和多重PCR方法分离鉴定出一株E.coliO157:H7,为致病性大肠杆菌。Jame等对奶制品和牛肉的调查发现,动物中血清型O157:H7的检出率在0~2.8%,而最高的检出率来源于犊牛而非成年牛[11]。与本研究从患有腹泻和胸腹膜炎的犊牛中分离出O157∶H7结果一致。目前,EHEC O157∶H7检测有血清学检测、免疫学检测、分子生物学检查等方法,本研究采用王革华多重PCR方法鉴定分离菌株的血清型,利用标准质控菌株CICC 21530作为阳性对照,检测编码E.coli菌体抗原O157的基因rfbEO157和编码E.coli结构性鞭毛抗原H7的基因fliCH7。此方法能够快速检测出血清型O157∶H7,操作简单、检测周期短、价格低廉且敏感性高,适合EHEC O157:H7实验室诊断分析。
大肠杆菌普遍存在于环境中和动物机体内,在兽医临床抗生素选择压力下耐药菌株增多。由于耐药基因可以在细菌间水平传播,新耐药表型不断产生,很多细菌已由单一耐药发展到多重耐药,不仅给预防和治疗动物疾病带来困难,而且加剧抗生素在畜禽体内的残留,严重危害动物性食品安全。本研究表明该牛场致犊牛爆发胸膜炎、腹膜炎的E.coliO157:H7菌株多重耐药,对第一、二代头孢类抗生素严重耐药,对头孢哌酮、头孢他啶敏感;对氨基糖苷类抗生素普遍敏感,仅对链霉素耐药,对四环素、利福平、复方新诺明、环丙沙星和红霉素有较强耐药性,并对其他抗生素也有不同程度的耐药。本研究结果与刘明春等[12]、李军等[13]、赵秋华等[14]、张秀英等[15]报道基本一致。
大肠杆菌耐药性的产生主要与两个因素有关:耐药基因的存在和药物的选择性压力。质粒介导的抗生素耐药基因不仅可垂直传给子代,更重要的是可在不同微生物间进行水平传播。根据Mathur等的报道[16-19],本研究分别针对β-内酰胺类、氨基糖苷类、四环素类、链霉素类、磺胺类、喹诺酮类、氯霉素类设计14对引物。耐药基因PCR扩增结果显示,该分离菌株携带tetB、strB、aadB、aphA、floR和TEM耐药基因。故该分离菌株的耐药基因型为四环素类,链霉素类,氨基糖苷类,氯霉素类和β-内酰胺类。
本研究发现,该菌株耐药表型和耐药基因型不完全匹配,分离菌携带氯霉素和氨基糖苷类的耐药基因,但并不表现出这两类的耐药表型。这可能是由于不同耐药基因表达的酶不同,各种酶的耐药谱不同,因此耐药表型和耐药基因型间存在差异。建议在今后E.coliO157:H7治疗过程中慎重用药,避免新耐药性产生,防止细菌间耐药基因水平传播。
[1]冉陆.肠出血性大肠杆菌(EHEC)流行趋势[J].中国食品卫生杂志,1999,11(3):31-34.
[2]Ozgur C,Belgin S,Inat G,et al.The prevalence ofEscherichia coliO157 and O157:H7 in ground beef and raw meatball by immuno⁃magnetic separation and the detection of virulence genes using multiplex PCR[J].Meat Science,2010,84:553-556.
[3]Sanchez S,Martinez R,Garcia A,et al.Detection and character⁃ization of O157:H7 and non-O157 shiga toxin-producingEsche⁃richiacoli in wild boars[J].Veterinary Microbiology,2010,143: 420-423.
[4]孟祥升,辛崇兴,邵晞.肠出血性大肠杆菌O157:H7研究进展[J].中国动物检疫,2011,28(11):69-71.
[5]Yuan W,Chai T J,Miao Z M.ERIC-PCR identification of the spread of airborneEscherichia coliin pig houses[J].Science of the Total Environment,2010,408:1446-1450.
[6]Claudia M,Jaime R,Romilio T E.Polymorphism in repeated 16S rRNA genes is a common property of type strains and environmen⁃tal isolates of the genusVibrio[J].Microbiology,2002,148:1233-1239.
[7]Wang G,Clifford G C,Frank G R.Detection inEscherichia coliof the genes encoding the major virulence factors,the genes defining the O157:H7 Serotype,and components of the type 2 shiga toxin family by multiplex PCR[J].Journal of clinical microbiology,2002 (10):3613-3619.
[8]John H L.Antimicrobial resistance ofEscherichia coliO26 and O111 isolates from cattle and their characteristics[J].Veterinary Microbiology,2009,135:401-405.
[9]Patrick B,Rebeccah T,Carlton L,et al.Antimicrobial resistance and virulence genes ofEscherichia coliisolates from swine in on⁃tario[J].Applied and Encironmental Microbiology,2005,71(11): 6753-6761.
[10]布坎南R E,吉本斯N E.伯杰细菌鉴定手册[M].8版,北京:科学出版社,1984:385-388.
[11]James P N,James B K.DiarrheagenicEscherichia coli[J].Clinical Microbiology Reviews,1998(1):142-201.
[12]刘明春,佟恒敏,何剑斌,等.16种抗菌药物对大肠杆菌的体外抑菌研究[J].东北农业大学学报,2003,34(2):176-178.
[13]李军,禤雄标,谢宇舟,等.广西畜禽大肠杆菌O157:H7流行病学调查[J].中国人畜共患病学报,2011,27(12):1151-1155.
[14]赵秋华,王少辉,刘萍萍,等.上海市闵行区猪场中大肠杆菌O157:H7的分离鉴定及生物学特性研究[J].中国动物传染病学报,2013,21(3):38-44.
[15]张秀英,王磊杰,徐国锋,等.哈尔滨地区猪源多重耐药大肠杆菌接合性质粒和整合子检测[J].东北农业大学学报,2013,44 (9):40-45.
[16]Mathur S,Singh R.Antibiotic resistance in food lactic acid bacte⁃ria——a review[J].Int J Food Microbiol,2005,105(3):281-295.
[17]Singh A,Shahid M,Sobia F,et al.Occurrence and molecular epi⁃demiology of blaCTX-M,including co-occurrence of blaTEM and blaSHV genes,and sul1 association in Indian Enterobacteriaceae [J].Int J Antimicrob Agents,2012,39(2):184-185.
[18]Perreten V,Boerlin P.A new sulfonamide resistance gene(sul3) inEscherichia coliis widespread in the pig population of Switzer⁃land[J].Antimicrob Agents Chemother,2003,47(3):1169-1172.
[19]Friedman S M,Lu T,Drlica K.Mutation in the DNA gyrase A gene ofEscherichia colithat expands the quinolone resistancedetermining region[J].Antimicrob Agents Chemother,2001,45 (8):2378-2380.
Isolation and identification ofEscherichia coliO157∶H7 from cattle and analysis of resistant gene
LIU Yun1,LU Ting1,ZHANG Liguo1,ZHAO Mingli1,SUN Dongdong1,SUN Sichao1,LIANG Weifeng1,ZHOU Guohui2,SHI Dongfang1,YU Li2
(1.School of Veterinary Medicine,Northeast Agricultural University,Harbin 150030,China;2.Harbin Veterinary Research Institute,Chinese Academy of Agricultural Sciences,Harbin 150001,China)
To investigate pathogenic bacteria which caused serious calf diarrhea,theEscherichia coliO157∶H7 isolate was isolated from diseased tissue and identified by PCR method.The result of drug sensitivity test showed that the strain exhibited certain degrees of drug resistance to 12 kinds of antibiotics resistance,such as penicillin,tetracycline and ampicillin.While the strain was susceptible to eight kinds of antibiotics resistanc,such as cefoperazone,ceftazidime,amikacin.The strain possessedtetB,strB,aadB,aphA,floRandTEMgenes.The drug resistant genotype and the resistance phenotype was not identical.
E.coliO157∶H7;drug sensitivity test;resistance genes
S858.23
A
1005-9369(2014)11-0083-06
2014-03-25
兽医生物技术国家重点实验室开放课题基金项目(SKLVBF201303);国家科技部支撑项目(2012BAD12B03,2012BAD12B05)
刘云(1964-),女,教授,博士,博士生导师,研究方向为肿瘤分子生物学及奶牛疾病等。E-mail:abliuyun@yeah.net
时间2014-11-21 16:39:00[URL]http://www.cnki.net/kcms/detail/23.1391.S.20141121.1639.008.html
刘云,卢婷,张力国,等.牛源性E.coliO157∶H7分离鉴定、耐药性及耐药基因分析[J].东北农业大学学报,2014,45(11):83-88.
Liu Yun,Lu Ting,Zhang Liguo,et al.Isolation and identification ofEscherichia coliO157∶H7 from cattle and analysis of resistant gene[J].Journal of Northeast Agricultural University,2014,45(11):83-88.(in Chinese with English abstract)

