杜仲MVA途径相关基因全长cDNA序列特征研究
王 淋,乌云塔娜,刘慧敏,叶生晶,杜红岩
(1.中南林业科技大学 a.经济林育种与栽培国家林业局重点实验室;b.林学院,湖南 长沙 410004;2.中国林业科学研究院 经济林研发中心,河南 郑州 450003)
杜仲MVA途径相关基因全长cDNA序列特征研究
王 淋1a,b,乌云塔娜2,刘慧敏1a,b,叶生晶1a,b,杜红岩2
(1.中南林业科技大学 a.经济林育种与栽培国家林业局重点实验室;b.林学院,湖南 长沙 410004;2.中国林业科学研究院 经济林研发中心,河南 郑州 450003)
杜仲幼果和成熟果转录组测序的基础上,分离鉴定了幼果和成熟果MVA途径ACOT、HMGS、HMGR基因全长cDNA序列,并对基因的核苷酸序列及其相应氨基酸序列的组成成分、跨膜结构域、疏水性/亲水性、蛋白质二、三级结构、分子系统进化等进行分析。EuACOT基因全长cDNA为1 248 bp,编码416个氨基酸,属于稳定疏水性蛋白,无明显的跨膜区,在进化上分属于不同的分类群,与烟草和番茄的ACOT蛋白间的亲缘关系最为接近;EuHMGS基因全长cDNA为1 356 bp,编码452个氨基酸,属于不稳定的疏水性蛋白,无明显的跨膜区,在进化上分属于不同的分类群,与茶树的HMGS蛋白间的亲缘关系最为接近;EuHMGR基因全长cDNA为1 770 bp,编码590个氨基酸,属于不稳定的疏水性蛋白,有两个较明显的跨膜区,在进化上分属于不同的分类群,与杜仲的HMGR蛋白间的亲缘关系最为接近;以上三个蛋白的二级结构均是以α-螺旋和螺环结构为主的混合型结构。
杜仲;萜类物质; MVA途径;序列分析;生物信息学
杜仲Eucommia ulmoidesOliv. 为落叶乔木,单科单属,又名丝楝树皮、胶树等,是我国特有新生代第三纪孑遗植物[1]。杜仲的药用价值较高,是名贵的中药材;除此之外,杜仲的叶子、果实,树皮等组织均含有大量的白色丝状物质-杜仲胶,杜仲胶是天然橡胶之一,其化学成分为都是反-1,4-聚异戊二烯,而是巴西橡胶分顺-1,4-聚异戊二烯,两者互为同分异构体,但是物理性质却有大不相同,杜仲胶具有塑胶二重性,是目前最有开发前景的温带胶资源,具有较高的工业应用价值[2-3]。
乙酰CoA酰基转移酶 (AOCT) 是萜类化合物生物合成甲羟戊酸 (MVA)途径的起始酶, 催化使2个分子的乙酰CoA 缩合为乙酰乙酰CoA,AOCT催化蛋白质的酰基化和去酰基化,在真核生物中蛋白质翻译后修饰中酰基化和去酰基化是普遍的方式,AOCT属于硫解酶,能催化蛋白质的酰基化和去酰基化,所以AOCT具有调节蛋白质活性、基因表达等功能[4-5]。羟甲基戊二酰辅酶A合酶基因(HMGS)是甲羟戊酸途径中的重要的代谢酶,在MVA途径中,在HMGS催化下,乙酰CoA和乙酰乙酰CoA能生成羟甲基戊二酰CoA[6],进一步产生IPP,IPP为萜类物质合成提供前体,因此,HMGS在萜类物质的合成中扮演着重要的角色。不同基因编码有不同的基因亚细胞定位以及基因的表达也不同,HMGS基因家族成员有细胞质和线粒体两种形式。HMGS在细胞质中催化还原底物,为各种类异戊二烯化合物提供前体化合物IPP的合成,而位于线粒体中HMGS形式目前很少在植物中发现,与细胞质中的HMGS功能不同,位于线粒体的HMGS主要为酮体产生前体[7-8]。羟甲基戊二酰CoA还原酶(HMGR) 是天然橡胶类异戊二烯合成途径中关键酶之一,作为MVA途径首个限速酶, HMGR催化3-羟基-3-甲基戊二酰辅酶A从而形成甲羟戊酸,对甲羟戊酸代谢“碳流”的调控起决定性作用[9]。根据所含异戊二烯数目不同,萜类物质可以分单萜、陪半萜、双萜、三萜和多萜等等,例如在马铃薯中,HMGR1基因与合成甾类化合物密切相关,而倍半萜植保素合成却与HMGR1 同一家族的HMGR2和HMGR3基因有关[10-11]。HMGR不仅与天然橡胶合成有关,同时也是类胡萝卜素、叶绿素、赤霉素等合成的相关酶之一[12]。
对杜仲幼果和成熟果实MVA途径ACOT、HMGS、HMGR基因进行详细的生物信息学分析,可为进一步深入研究杜仲萜类物质合成相关酶奠定了理论基础。
1 材料与方法
1.1 数据来源
对杜仲胶合成时期转录组测序及转录组数据组装及基因功能注释[13-14],其中萜类物质合成MVA途径共涉及30条Unigene,其中鉴定了ACOT、HMGS、HMGR基因分别有7条、3条、11条。
1.2 方 法
根据NCBI(National Center for Biotechnology Information, http: //www. ncbi. nlm.nih.gov/)中 核酸及蛋白质数据库ACOT、HMGS、HMGR基因的核酸序列及其对应的氨基酸序列进行相似性检索,并利用ORF Finder程序查找出基因cDNA 开放阅读框;利用ExPASyProtParam( http: / /cn.expasy.org) 程序与ScanProsite程序对氨基酸残基数、氨基酸组成、蛋白质相对分子量、理论等电点以及蛋白质的亲疏水性等性质进行了分析;利用TMHMM程序(http://www.cbs.dtu.dk/services/TMHMM-2.0/)预测蛋白质跨莫结构域;利用PSIPRED 程 序 ( http: / /bioinf.cs.ucl.ac.uk /psipred) 预测蛋白质的二级结构;利用ESyPred3D Web Server程序,根据基因氨基酸序列进行蛋白同源建模,准确预测蛋白质的三级结构;利用MEGA 5 软件构建基因系统进化树,对蛋白的亲缘关系进行相关的分析。
2 结果与分析
2.1 EuACOT基因的生物信息学分析
2.1.1 EuACOT基因的理化性质预测与分析
EuACO(Unigene1729)氨基酸序列与烟草 Nicotiana tabacum(AAU95619.1)、 番 茄 红素 Solanum lycopersicum(XP_004236650.1)、 桃Prunus persica(EMJ12994.1)的ACOT氨基酸序列的相似性均为81%(见图1)。EuACOT1基因全长cDNA长度为1 248 bp,编码416个氨基酸,其中甘氨酸(11.5%)>丙氨酸(11.5%)>亮氨酸(10.1%)三种氨基酸含量最高,EuACOT蛋白相对分子量为43.18 kD,理论等电点为9.01;蛋白不稳定系数为31.84,属于稳定蛋白质。
2.1.2 EuACOT基因疏水性/亲水性的预测
Expasy Protscale程序分析EuACOT蛋白亲水性/疏水性的结果如图2所示,EuACOT蛋白氨基酸残基中以A375疏水性最强(3.000),以A183亲水性最强(-2.211),疏水氨基酸的数量的数量大于亲水氨基酸,故推断 EuACOT为疏水性蛋白。
2.1.3 EuACOT编码蛋白的疏水/亲水性预测与分析
EuACOT蛋白的疏水/亲水性预测的结果如图2所示,EuACOT蛋白氨基酸残基中A375(3.000)疏水性最强,以A183(-2.211)亲水性最强,A375(3.000)>A183(-2.211)故EuACOT蛋白的疏水性大于亲水性,EuACOT为疏水性蛋白。

图1 EuACOT与同源序列的比对Fig.1 Alignment homologous sequence of ACOT
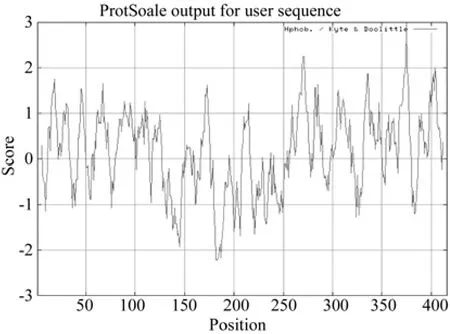
图2 EuACOT蛋白的亲水/疏水性的预测Fig.2 Prediction on hydrophobicity/hydrophilicity of EuACOT
2.1.4 EuACOT基因跨膜结构域的预测
利用在线工具TMHMM-2.0对EuACOT氨基酸序列的跨膜结构域进行预测见图3,EuACOT蛋白N端在细胞膜内的可能性为 0.014 10,预测EuACOT蛋白无跨膜区,蛋白全部在膜外。
2.1.5 EuACOT基因二级结构的预测
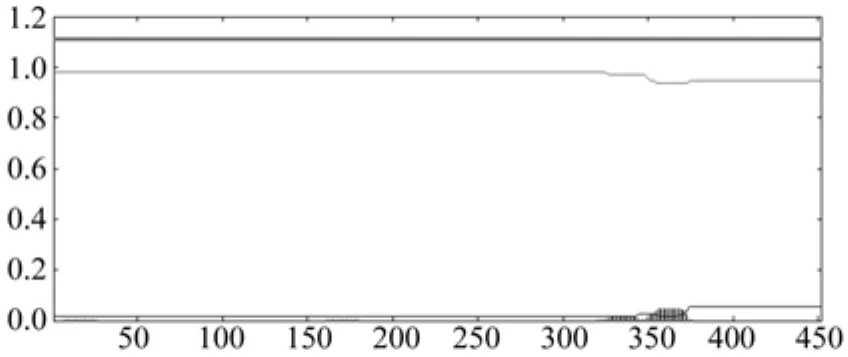
图3 EuACOT蛋白的跨膜区预测Fig.3 Predicted transmembrane of deduced protein of EuACOT
由图4可知,EuACOT蛋白二级结构中,以螺环结构(45.43%)和α-螺旋(41.35%)为主,其中螺环结构(45.43%)>α-螺旋(41.35%)>β-折叠(13.22%),故推测EuACOT为混合型蛋白质。
2.1.6 EuACOT的三级结构的预测
采用auto mode建模方式以蛋白模板(2ib8D)对EuACOT蛋白进行同源建模(见图5),EuACOT蛋白的三维空间模型。推导EuACOT蛋白模型分值为0.56,蛋白序列的相似性为46.99%。
2.1.7 EuACOT氨基酸序列分子系统进化分析

图4 预测EuACOT蛋白的二级结构Fig.4 Prediction on secondary structure of EuACOT
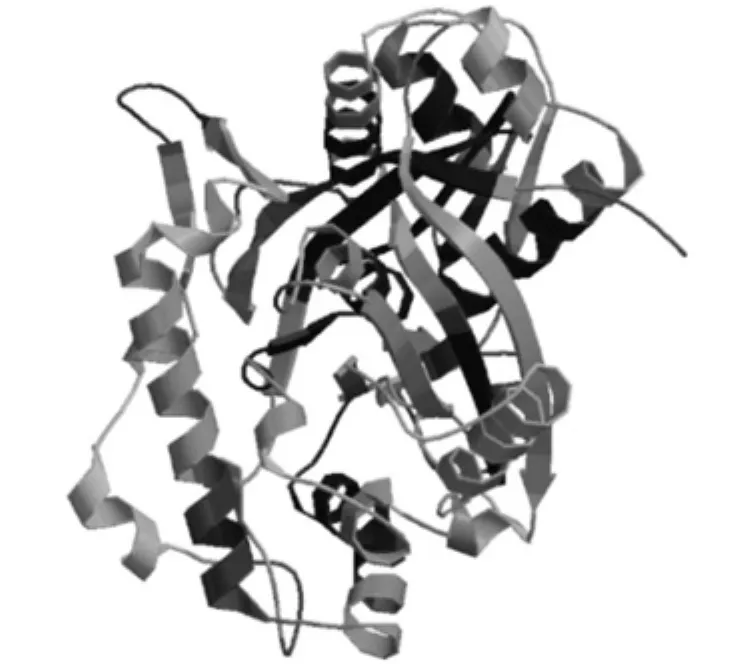
图5 EuACOT蛋白三级结构预测Fig.5 Prediction on tertiary structure of EuACOT
系统进化分析是研究物种进化和系统分类的一种方法,研究对象为携带遗传信息的生物大分子序列,通过序列同源性的比较,采用特定的数理统计算法来计算生物间的生物系统发生的关系。利用MEGA5比较不同物种的ACOT基因的氨基酸序列,构建了系统进化树(见图6、7),在聚类分析中可以看出,EuACOT蛋白与烟草和番茄的ACOT蛋白在聚类分析图的距离较近,说明在进化遗传中杜仲、烟草和番茄的亲缘关系较近,由系统发育树图值,ACOT基因进化关系复杂多样,不同物种的ACOT基因于多为不同的类群。
2.2 EuHMGS基因的生物信息学分析
2.2.1 EuHMGS基因的理化性质的预测与分析
EuHMGS(Unigene17165)氨基酸序列与茶树Camellia sinensis(AFC34137.1)、喜树Camptotheca acuminata(ACD87446.1)、龙蕃茄红素Solanum lycopersicum(XP_004252572.1)的HMGS氨基酸序列的相似性分别为90%、88%、88%(见图8)。EuHMGS基因全长cDNA长度为1 356 bp,编码452个氨基酸,其中丝氨酸(9.7%)>丙氨酸(9.2%)>亮氨酸(7.7%) 3个氨基酸含量最高,EuHMGS蛋白相对分子量为50.20 kD,理论等电点为6.04;蛋白不稳定系数为40.17,故属于不稳定蛋白质。
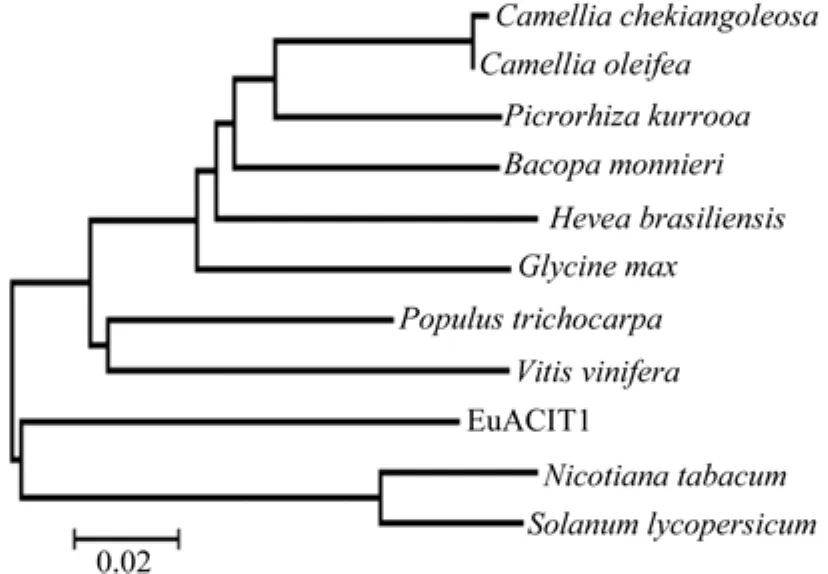
图6 EuACOT蛋白的聚类分析Fig.6 Cluster analysis of EuACOT
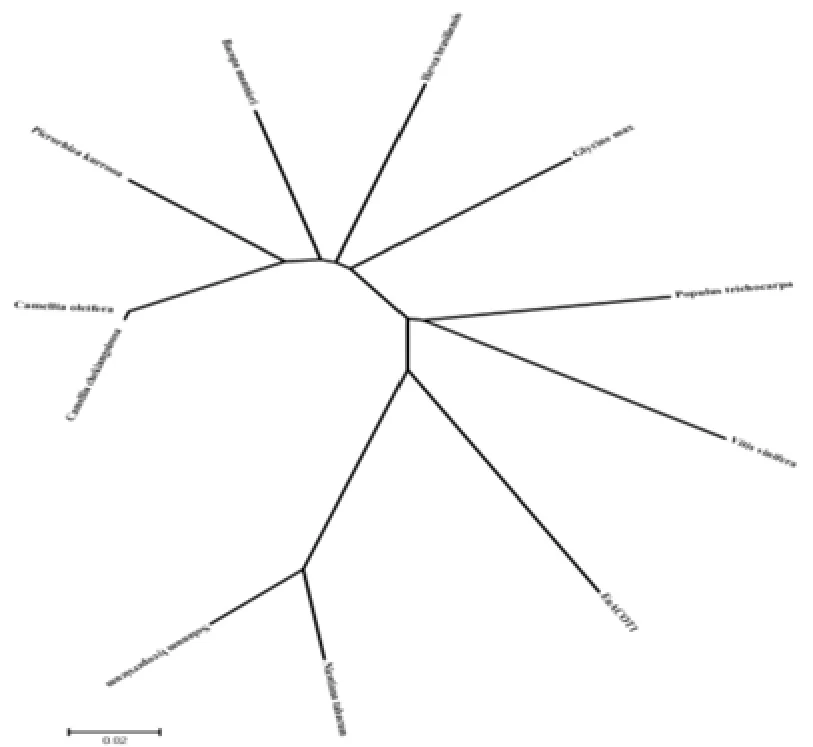
图7 EuACOT系统发育树Fig.7 Phylogenetic tree of EuACOT
2.2.2 EuHMGS基因疏水性/亲水性的预测
Expasy Protscale程序分析EuHMGS蛋白亲水性/疏水性的结果如图9所示,EuHMGS蛋白氨基酸残基中以A169疏水性最强(2.389),以A396亲水性最强(-2.566),疏水氨基酸的数量的数量大于亲水氨基酸,故EuHMGS推断为疏水性蛋白。
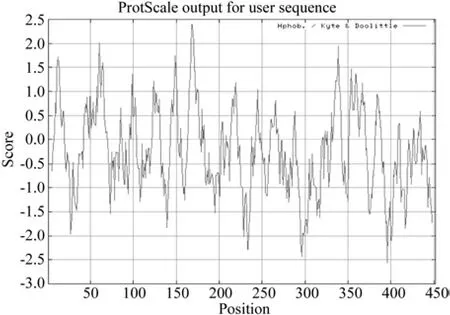
图9 EuHMGS蛋白的亲水/疏水性的预测Fig.9 Prediction on hydrophobicity/hydrophilicity of EuHMGS
2.2.3 EuHMGS编码蛋白的疏水/亲水性分析
EuHMGS蛋白的疏水/亲水性预测的结果如图9所示,EuHMGS蛋白氨基酸残基中A169(2.389)疏水性最强,以A396(-2.566)亲水性最强,A169(2.389)>A396(-2.566)故疏水性大于亲水性,EuHMGS为疏水性蛋白。
2.2.4 EuHMGS基因跨膜结构域的预测
利用在线工具TMHMM-2.0对EuHMGR氨基酸序列的跨膜结构域进行预测见图10,EuHMGS蛋白N端在细胞膜内的可能性为 0.019 21,EuHMGS蛋白无跨膜区,蛋白全部在膜外。
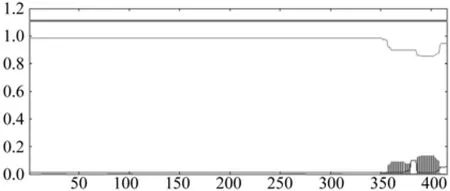
图10 EuHMGS蛋白的跨膜区预测Fig. 10 Predicted transmembrane of deduced protein of EuHMGS
2.2.5 EuHMGS基因二级结构的预测
由图11可知,EuHMGS蛋白二级结构以螺环结构(49.78%)和α-螺旋占(38.05%)为主,其中螺环结构(49.78%)>α-螺旋占(38.05%)>β-折叠占(12.17%),属于混合型结构的蛋白质。

图11 EuHMGS蛋白的二级结构的预测Fig.11 Prediction on secondary structure of EuHMGS
2.2.6 EuHMGS的三级结构的预测
采用auto mode建模方式以蛋白模板(2f82A)对EuHMGS蛋白进行同源建模(见图12),EuHMGS蛋白的三维空间模型。推导EuACOT蛋白模型分值为0.7,蛋白序列的相似性为83.96%。
2.2.7 EuHMGS氨基酸序列分子系统进化分析
利用MEGA5比较不同物种的ACOT基因的氨基酸序列,构建了系统进化树(见图13、14),在聚类分析中可以看出,EuHMGS蛋白与茶HUMGS蛋白在聚类分析图的距离较近,说明在进化遗传中杜仲与茶的亲缘关系较近,由系统发育树图值,HUMGS基因进化关系复杂多样,不同物种的ACOT基因于多为不同的类群。
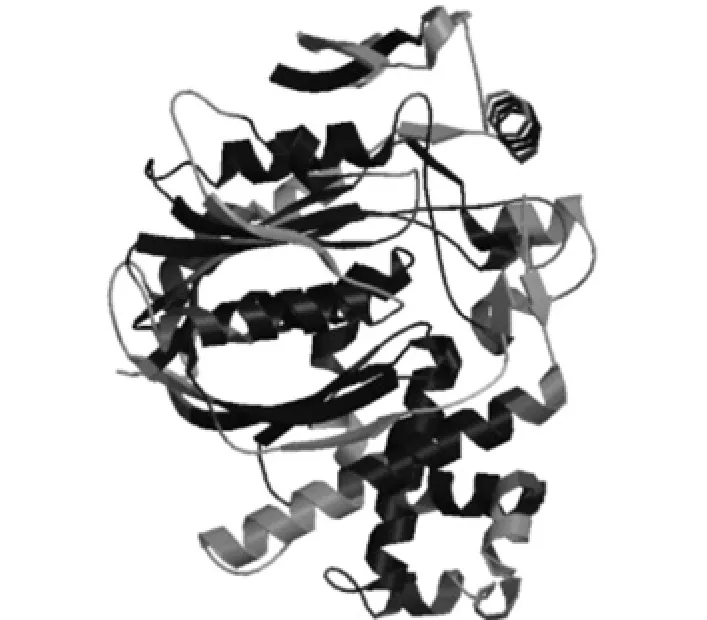
图12 EuHMGS蛋白三级结构预测Fig.12 Prediction on tertiary structure of EuHMGS
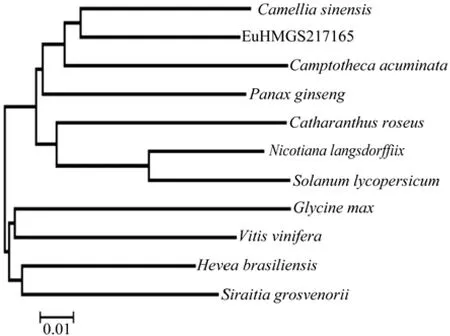
图13 EuHMGS蛋白的聚类分析Fig.13 Cluster analysis of EuHMGS
2.3 EuHMGR基因的生物信息学分析
2.3.1 EuHMGR基因的理化性质的预测与分析
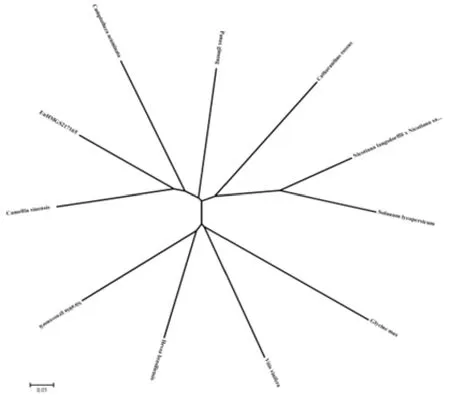
图14 EuHMGS系统发育树Fig.14 EuHMGS phylogenetic tree
EuHMGR(Unigene5369)氨基酸序列与杜仲Eucommia ulmoides(AY796343.1 )、 葡 萄Vitis vinifera(XM_002275791.2)、刺五加Eleutherococcus senticosus(AFM77981.1 )基酸序列的相似性分别为99%、82%、79%(见图15)。EuHMGR9基因全长cDNA为1 770 bp,编码590个氨基酸,氨基酸组成中亮氨酸(9.8%)>丝氨酸(9.8%)>丙氨酸(9.5%)这3种氨基酸含量较高,EuHMGR蛋白的相对分子量为63.30 kD,理论等电点为6.83,蛋白不稳定系数为44.64,属于不稳定蛋白质。

图15 EuHMGR与同源序列的比对Fig.15 Alignment homologous sequence of HMGR
2.3.2 EuHMGR基因疏水性/亲水性的预测
EuHMGR蛋白的疏水/亲水性预测的结果如图16所示,EuACOT蛋白氨基酸残基中A398(2.822)疏水性最强,以A73(-2.667)亲水性最强,A398(2.822)>A73(-2.667)故EuACOT蛋白的疏水性大于亲水性,EuACOT1为疏水性蛋白。
2.3.3 EuHMGR基因跨膜结构域的预测
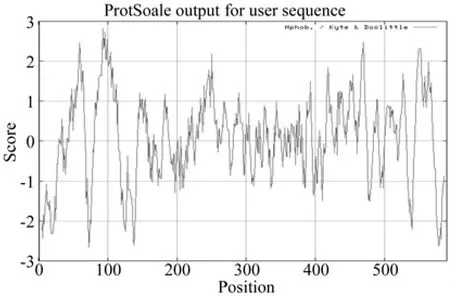
图16 EuHMGR蛋白的亲水/疏水性的预测Fig.16 Prediction on hydrophobicity/hydrophilicity of EuHMGR
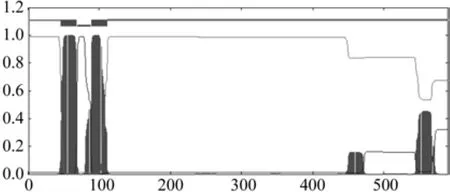
图17 EuHMG蛋白的蛋白的跨膜区预测Fig.17 Predicted transmembrane of the deduced protein of EuHMGR
利用在线工具TMHMM-2.0对EuHMGR氨基酸序列的跨膜结构域进行预测见图17,EuHMGR蛋白N端在细胞膜内的可能性为0.009 97,在N-末端附近有两个跨膜区共有两个较明显的跨莫螺旋结构(见图6)。其中EuHMGR的跨膜区为TMhelix1(46-68)、TMhelix2(89-111)均含有 22个氨基酸残基。
2.3.4 EuHMGS基因二级结构的预测
如图18,EuHMGR蛋白二级结构中,其中螺环结构(44.24%)和α-螺旋(42.88%)为主,螺环 结 构 (44.24%)> α-螺 旋 (42.88%)> β-折 叠(12.88%),故推测EuACOT为混合型蛋白质。

图18 预测EuHMGR蛋白的二级结构Fig.18 Prediction on secondary structure of EuHMGR
2.3.5 EuHMGR的三级结构的预测
采用auto mode建模方式以蛋白模板(2q6bA)对EuHMGR蛋白进行同源建模(见图19),EuHMGR蛋白的三维空间模型,推导EuACOT蛋白模型分值为0.63,蛋白序列的相似性为55.37%。
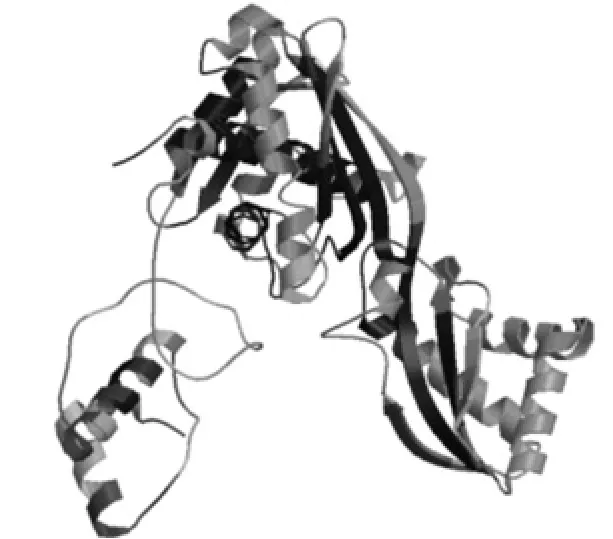
图19 EuHMGR蛋白三级结构预测Fig.19 Prediction on tertiary structure of EuHMGR
2.3.6 EuHMGR氨基酸序列分子系统进化分析
利用MEGA5中Clustal W法对不同物种HMGR氨基酸序列进行比对,并用Neighborjoining法构建了系统进化树(见图17、图18)。结果表明不同来源的HMGR蛋白序列在进化上分属于不同的分类群,说明其进化关系较为复杂。EuHMGR蛋白与杜仲的HMGR蛋白间的亲缘关系最为接近。
利用MEGA5比较不同物种的HMGR基因的氨基酸序列,构建了系统进化树(见图20、21)。在聚类分析中可以看出,EuHMGR蛋白与杜仲的HMGR蛋白在聚类分析图的距离较近,说明在进化遗传中与杜仲的亲缘关系较近,由系统发育树图值,HMGR基因进化关系复杂多样,不同物种的ACOT基因于多为不同的类群。
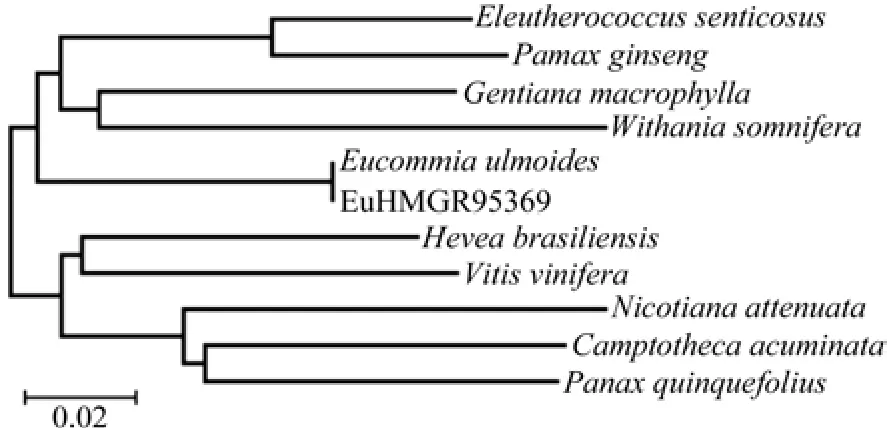
图20 EuHMGR蛋白的聚类分析Fig.20 Cluster analysis of EuHMGR
3 讨论与结论
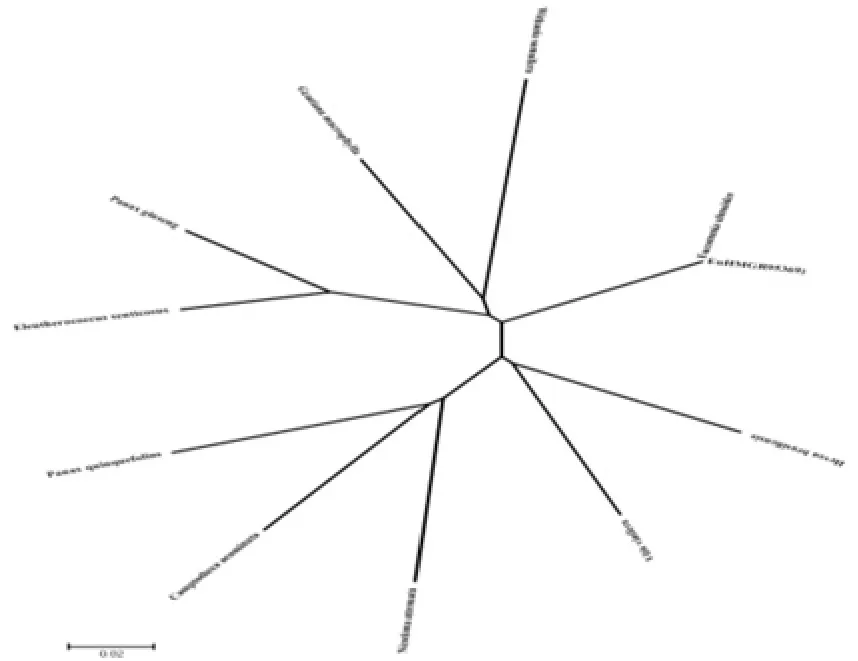
图21 EuHMGR系统发育树Fig.21 EuHMGR phylogenetic tree
乙 酰CoA酰 基 转 移 酶 (acetyl-CoA C-acetyltransferase, ACOT) 是萜类化合物生物合成甲羟戊酸 (MVA)途径的起始酶,对EuACOT基因序列进行同源性分析和分子进化分析表明EuACOT是一种与植物类异戊二烯MVA途径相关的蛋白;生物信息学分析表明EuACOT基因全长cDNA为1 248 bp,编码416个氨基酸,属于稳定的疏水性蛋白,无明显的跨膜区,二级结构以α-螺旋和螺环结构为主的混合型结构,进化关系多样复杂,不同来源的ACOT蛋白属于不同的类群,其中与杜仲ACOT关系较为亲近的为烟草和栽培番茄ACOT基因。
HMG-COA合酶的催化涉及到许多反应,如乙酰化反应、缩合反应和水解反应等,这些反应的模式类似Bi- Bi-Ping-Pong的形式[14-15]。HMGCOA合酶氨基酸有天冬酰胺、半胱氨酸、组氨酸的保守的氨基酸[16]。 羟甲基戊二酰辅酶A合酶基因(HMGS)是MVA 途径中的一个非常重要的代谢酶,目前,已经在橡胶树和拟南芥等植物中对HMGS基因进行了分离克隆和表达分析以及功能鉴定,但关于杜仲HMGS基因的相关信息却尚未见公开报道[17-18]。文章对甲羟戊酸途径的关键酶基因HMGR进行生物信息学分析:EuHMGS基因全长cDNA为1 356 bp,编码452个氨基酸,属于不稳定的疏水性蛋白,无跨膜区,二级结构是以α-螺旋和螺环结构为主的混合型结构,不同物种的HMGS蛋白序列属于不同类群。羟甲基戊二酰CoA还原酶( HMGR) 调控类异戊二烯物质合及代谢过程,在确定“碳流”和萜类物质合成产物比例分配调节中HMGR合酶起到了至关重要的作用[19-20]。在目前现有的研究中,提高萜类物质有效成分的含量主要是通过植物转基因技术,即通过基因枪、叶盘法、花粉通道法等将基因转入到目地的植物中,比如将橡胶中的HMGR基因转入烟草中,可使烟草HMGR活性提高,同时,烟草中的甾醇含量也有所增加[21]。本文对甲羟戊酸途径的关键酶基因HMGR进行生物信息学分析,结果表明EuHMGR基因全长cDNA为1 770 bp,编码590个氨基酸,不稳定的疏水性蛋白,有两个较明显的跨膜区,二级结构均是以α-螺旋和螺环结构为主的混合型结构,EuHMGR蛋白与杜仲的HMGR蛋白间的亲缘关系最为接近。
类萜生物合成途径是生物体内主要的代谢途径之一,本研究采用转录组测序方法,对杜仲MVA途径相关基因通过生物信息学方法分别从核苷酸和蛋白质序列水平上, 分析了该基因的特征,为预测杜仲胶合成过程中的功能提供了指导, 也为揭示杜仲胶合成规律和杜仲的分子育种提供了理论依据和科学基础。
[1] 赵德刚,韩玉珍,傅永福,等.杜仲胶生物合成蛋白质的研究[J].中国农业大学学报, 1999 ,4(1):114.
[2] 杜红岩.我国的杜仲胶资源及其开发潜力与产业发展思路[J].经济林研究,2010, 28(3): 1-6.
[3] 杜红岩,王俊鸿,杜兰英,等.杜仲感技术产品的研究与开发[J]. 经济林研究,2001,19(2): 18-21.
[4] 张 琳,谭晓风,胡 娇,等.油茶乙酰CoA酰基转移酶基因cDNA克隆及序列特征分析[J].中南林业科技大学学报,2011, 31(8):108-112.
[5] 崔光红,王学勇,冯 华,等.丹参乙酰CoA酰基转移酶基因全长克隆和SNP分析[J]. 药学学报 ,2012, 45(6):85-790.
[6] 周宪林.甲基茉莉酸对喜树中HMGS基因表达的影响初探[J].井冈山医专学报, 2009, (16) :6.
[7] Ayte J, Gil-Gomez G, Haro D,et al.Rat mitochondrial and cytosolic 3-hydroxy-3-methylglutaryl-CoA synthases are eneoded by two differentgenes [J]. Proe Natl Acad Sci USA.1990, 87(10):3874-3878.
[8] Masearo C,Buesa C, Ortiz J A,et al.Moleeular cloning and tissue expression of human mitoehondrial 3-hydroxy-3-methylglutaryl-CoA synthases[J]. Arch Bioehem BioPhys, 1995, 10(2): 385-390.
[9] Suwanlnaneel P, Slrinupong N, Suvachlttanont W. Regulation of the expression of 3-hydroxy-3-methylglutaryl-CoA synthases inHevea brasiliensis(B.H.K.) Mull. Arg[J]. Plant Sci, 2004, 166:531-537.
[10] 陈大华,叶和春,李国凤,等.马铃薯HMGR基因的克隆、序列分析及其表达特征[J].植物学报, 2000 ,42(7):724-727.
[11] Korth L K, Stermer B A, Bhattacharyya M K,et al.HMG_CoA reductase gene families that differentially accumulate transcripts in potato tubers are developmentally expressed in floral tissues[J]. Plant Mol Biol. ,1997 , 33: 545-551.
[12] Chot D. Differential inductin and suppression of potato 3-hydroxy-3-methylglutaryl coenzyme A reductace genes in respose to Phytophora infestans and to its ehenor acachidonic acid [J]. Plant Cell ,1993, 4:1333-1344.
[13] 李铁柱,杜红岩,刘慧敏,等.杜仲幼果和成熟果实转录组数据组装及基因功能注释[J].中南林业科技大学学报,2012,32(10): 9-17.
[14] 李铁柱,杜红岩,刘慧敏,等.杜仲果实和叶片转录组数据组装及基因功能注释[J].中南林业科技大学学报, 2012, 32(11):22-130.
[14] Middleton B. The kinetic mechanism of 3-hydroxy-3-methylglutaryl-CoA synthases from baker’s yeast [J]. Bioehem Journal, 1972, 126(l):35-47.
[15] Miziorko HM, Clinkenbeard KD, Reed WD, et al. 3-hydroxy-3-methylglutaryl-CoA synthases Evidence for anacetyl-S-enzyme intermediate and ideniifieation of a cysteinyl sulthydryl as the site of acetylation [J]. J. Biol. Chem., 1975 10(15):5768-73.
[16] Priee A C, ChoiK-H, Health R J, et al. Inhibition of b-ketoacylacyl carrier protein synthases by thiolaetomyein and cerulenin Structure and meehanism[J]. J. Biol. Chem., 2001,276:6551-6559.
[17] 费小雯, 邓晓东, 胡新文,等.橡胶延伸因子基因及3-端非编码区的克隆与序列分析[J]. 生命科学研究,1999 ,(4):15-18.
[18] 王凤华,刘文锋.拟南芥HMGs家族成员全基因组分析[J].安徽农业科学, 2012,40(36) : 17478-17481.
[19] Dai Z B, Cui G H, Zhou S F, et al. Clong and characterixation of a novel 3-hydroxy-3-methylglutary coenzyme A reductase gene from Salvia miltiorrhiza involved in diterpenoid transhinone accumulation[J]. J. Plant Physiol, 2001, 168:148-157.
[20] 武 莹,李 威,祝传书.雷公藤HMGR基因克隆、序列分析及诱导表达[J].西北农业科技大学学报:自然科学版,2012,40(11): 103-111.
[21] 王启超, 刘实忠, 校现周.橡胶HMGR基因的克隆及表达分析[J].植物研究,2012, (1):61-68.
Study on full length cDNA of MVA pathway related genes in Eucommia ulmoides
WANG Lin1a,b, WUYUN Ta-na2, LIU Hui-min1a,b, YE Sheng-jing1a,b, DU Hong-yan2
(1a. Key Lab of Non-wood Forest Product of Forestry Ministry; b.School of Forestry, Central South University of Forestry & Technology,Changsha 410004, Hunan, China; 2.Non - timber Forestry Research and Development Center, CAF, Zhengzhou 450003, Henan, China)
This analysis was based on data of the transcriptome sequencing ofEucommiayoung fruit and mature fruit. The terpene synthesis of MVA pathway involving of 30 Unigene. We identif i ed 3 full-length cDNA in MVA pathway of young fruit and mature fruit, These full-length cDNA were analyzed and predicted by the tools of bioinformatics in the following aspects: composition of the nucleotide sequence and corresponding amino acid sequence, transmembrane structure, hydrophobicity or hydrophilicity, secondary and tertiary structure of protein, molecular phylogenetic evolution and so on.The results are as follows: Full-length cDNA of EuACOT gene has 1 248 bp, and coded 416 aa, EuACOT is stable hydrophobic protein without transmembrane domain, and belong to different taxa in evolution, high consistency with theNicotiana tabacumandLycopersicon esculentumin ACOT protein sequences and high structure.Full-length cDNA of EuHMGS gene has 1 356 bp, and coded 452 aa, EuHMGS is unstable hydrophobic proteins without transmembrane domain, and high consistency with theCamellia sinensisin HMGR protein sequences and high structure. Full-length cDNA of EuHMGR gene has 1 770 bp, and coded 590 aa, EuHMGR is unstable hydrophobic proteins with two transmembrane domanin, and high consistency with theEucommia ulmoidesin HMGR protein sequences and high structure. The secondary structures of above three proteins are predominantly hybrid structure of alpha helical and spiral ring structure and belong to different taxa in evolution.
Eucommia ulmoidesOliv; terpenes; MVA pathway; sequence analysiss; bioinformatics
S727.34
A
1673-923X(2014)01-0094-09
2013-05-25
国家林业公益性行业科研专项(201004029);国家“十二五”科技支撑计划项目(2012BAD21B0502)
王 淋(1986-),女,辽宁鞍山人,硕士研究生,主要从事生经济林遗传育种研究
乌云塔娜(1975-),女,内蒙古通辽人,教授,主要从事经济林育种栽培的研究;E-mail:tanatanan@sina.com
[本文编校:吴 毅]

