5'-腺苷磷酰硫酸激酶及其R68K 突变体对5'-腺苷一磷酸3'羟基的磷酸化
杨 洋,黄园波,马建辉,孙梅好
(浙江师范大学化学与生命科学学院,浙江金华321004)
硫作为构成生命体的大量元素之一,是生命活动的必需元素,其主要存在于氨基酸、氨基酸代谢物及各种磺酸类化合物中[1-2],参与细胞的代谢及调控。硫酸根离子是细胞的基本硫元素来源,化学性质非常稳定,其通过膜转运蛋白进入细胞后,在ATP硫酸化酶(ATP sulfurylase,ATPS)的催化下接受ATP的腺苷一磷酸基团形成腺苷-5'-磷酰硫酸(adenosine 5'-phosphosulfate,APS,图1-A)。APS中的磷硫键具有非常高的化学能,ATPS催化的APS合成反应非常不容易进行,反应平衡常数大约为10-8~10-7[1,3]。在大肠杆菌中,ATPS和GTP水解酶(GTPase)组成异源二聚体形成的硫活化酶复合体(Sulfate Activating Complex,SAC)促进硫的活化。此SAC通过蛋白构象变化偶联ATPS和GTPase催化的2个反应即APS的合成和GTP的水解,提高APS合成速度116倍[1,4]。APS的3'羟基可被APS激酶(APSkinase,APSK)磷酸化形成3'-磷酸腺苷-5'-磷酰硫酸(3'-phosphoadenosine 5'-phosphosulfate,PAPS)[5-6],PAPS可在PAPS还原酶等酶的催化下还原,最后整合到半胱氨酸及其他还原性硫化合物中[1,7-8],也可作为硫酸转移酶(sulfotranferase,ST)的底物进行化合物的硫酸化,参与化合物的生理功能调节及蛋白之间的互作[1,9-10]。3',5'二磷酸核苷酸酶(3,5-bisphosphatenucleotidase,ND)[11-14]可催化3',5'二磷酸腺苷(3'-phosphoadenosine 5'-phosphate,PAP)3'-磷酸基团的水解产生AMP,其KmPAP为0.7~17μmol/L[14-16]。AMP可在肌激酶(myokinase,MK)以及丙酮酸激酶(pyruvatekinase,PK)的作用下磷酸化完成ATP的再生。
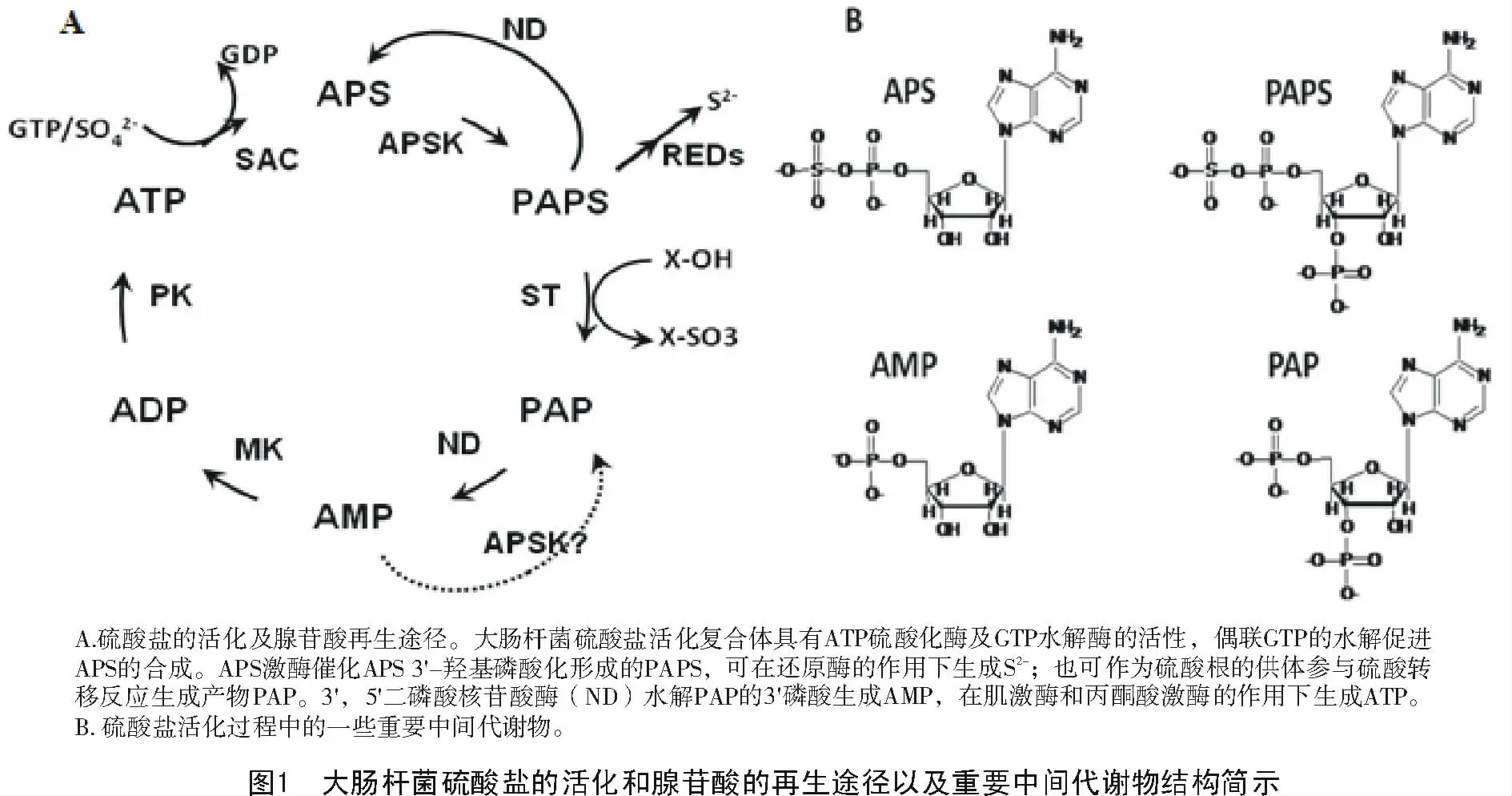
研究表明,ND活性降低引起的胞内PAP累积[12-15,17]可通过影响ST活性[18-19]、酰基载体蛋白合酶[20]和RNA加工酶[17,21]等的活性,影响细胞的生长发育。ND除水解PAP外,也水解PAPS的3'磷酸基团生成APS和无机磷酸[13,15,22-24]。
APS激酶催化APS磷酸化形成PAPS,是E.coli 硫代谢的必需酶。较APS少一个硫酸根离子(图1-B)的AMP是否可作为APS类似物被APSK磷酸化其3'羟基,生物体内是否存在AMP 3'羟基激酶(AMP 3'-hydxroxyl kinase,AMP3K),尚没有相关报道。本研究主要利用APSK及其突变体,分析了其活性。
1 材料和方法
1.1 菌株与载体
菌株Escherichia coli DH5α 及BL21(DE3),pSKB4(由pGEX-6P-1改造的双标签表达载体,其融合蛋白标签GST的N端具有6 Histines)由浙江省重中之重现代农业生物防治技术与作物病害防控实验室保存。pMD18-T简易载体购自TaKaRa公司。
1.2 酶与生化试剂
兔肌肉丙酮酸激酶(pyruvate kinase,PK)、兔肌肉乳酸脱氢酶(lactate dehydrogenase,LDH)购自罗氏公司;还原型辅酶I(NADH)、还原型谷胱甘肽(GSH)购自Sigma公司;镍柱亲和层析树脂(Ni-NTA) 购自Novagen公司,PreScission蛋白酶、谷胱甘肽硫转移酶(GST)亲和层析树脂为GEHealthcare产品;溶菌酶(lysozyme)、二硫苏糖醇(Dithiothreitol,DTT)、异丙基-β-D-硫代吡喃半乳糖苷(Isopropylβ-D-1-Thiogalactopyranoside,IPTG)、抑肽素(Pepstatin A)购自Amresco公司;磷酸烯醇式丙酮酸(phosphoenolpyruvate,PEP)、三磷酸腺苷(ATP)、苯甲基磺酰氟(Phenylmethanesulfonyl fluoride,PMSF)为BBI产品;QuikChangeRXL Site-Directed Mutagenesis Kit购自Stratagene公司;超滤管购自Millipore公司;pfu Taq DNA聚合酶和限制性内切酶分别购自Takara和New England Biolabs。腺苷5'-磷酰硫酸按照发表的方法[25]合成。其他为购自国内公司分析纯试剂。
1.3 方法
1.3.1 大肠杆菌APSK基因克隆、突变及蛋白表达、纯化 以E. coli DH5α 基因组为模板及PCR引物(APSKF和APSKR,表1)扩增APSK基因。扩增产物经琼脂糖凝胶电泳割胶回收、纯化及双酶切(Nde I/Xho I)后连接入表达载体pSKB4构建表达载体pSKB4-APSK。按照Stratagene的QuikChangeRXL Site-Directed Mutagenesis Kit突变方法(表1)进行R68K突变,所获载体为pSKB4-APSKR68K。分别转化pSKB4-APSK,pSKB4-APSKR68K入大肠杆菌BL21(DE3)感受态细胞,挑取单菌落于LB液体培养基(100 mg/L Amp)中37℃过夜培养。第2天重新接种250 mL LB液体培养基(100 mg/LAmp),37℃振荡培养至OD600为0.6。将菌液冷却至16℃,利用IPTG(0.5 mmol/L)16℃诱导培养16 h后,离心收集菌体。进一步参照组氨酸-GST双标签融合蛋白纯化方法[26]纯化APSK,其中所用缓冲液如下。
细胞裂解液:0.40 mol/L KCl,5.0 mmol/Lβ-巯基乙醇,1 mmol/L PMSF,1 mmol/L Pepstaine A,10 mg/L溶菌酶,50 mmol/L K2HPO4,pH值8.0。镍柱洗涤液:0.40 mol/L KCl,5.0 mmol/Lβ-巯基乙醇,10 mmol/L咪唑,50 mmol/L K2HPO4,pH值7.3。镍柱洗脱液:0.40 mol/L KCl,5.0 mmol/Lβ-巯基乙醇,250 mmol/L咪唑,50 mmol/L K2HPO4,pH值7.3。GST洗涤液:0.40 mol/L KCl,5.0 mmol/Lβ-巯基乙醇,50 mmol/L K2HPO4,pH值8.0。GST洗脱液:10 mmol/L GSH,100 mmol/L Tris-HCl,pH值8.0。透析酶切液:25 mmol/L Hepes,pH值7.5,50 mmol/L KCl,2.0 mmol/L DTT。收集目的蛋白,经浓缩、考马斯亮蓝定量后储存于-80℃冰箱待用。经SDS-PAGE电泳分析,发现所获蛋白纯度大于95%。

表1 克隆及突变引物
1.3.2 酵母YND的克隆及表达纯化 以酵母(Saccharomyces cerevisiae)基因组为模板及PCR引物(YNDf和YNDr,表1)扩增3',5'-核苷酸酶(yeast 3',5'-bisphosphate nucleotidase,YND)[12-13]基因。经琼脂糖凝胶电泳割胶回收、纯化及双酶切(Eco R I/Hin d III)连接入表达载体pET24a构建表达载体pETND。转化pETND入大肠杆菌BL21(DE3)感受态细胞,挑取单菌落于LB液体培养基(50 mg/L卡那霉素)中37℃过夜培养。第2天重新接种250 mL LB液体培养基(50 mg/L卡那霉素),37℃振荡培养至OD600为0.6。将菌液冷却至16℃,经16 h的IPTG(0.5 mmol/L)诱导蛋白表达后,离心收集菌体。蛋白的纯化参照Sun等[26]的方法进行,蛋白的定量参照方法1.3.1,蛋白经SDS-PAGE电泳分析,发现纯度大于95%。
1.3.3 APSK及突变体R68K的APS磷酸化活性测定 APSK或者R68K突变体催化APS磷酸化生成PAPS和ADP,生成的ADP可通过PK/LDH体系[27]中的PK磷酸化生成ATP和丙酮酸,丙酮酸可进一步被LDH通过氧化NADH(ε339nm=0.622 mmol/(L·cm))而还原为乳酸。反应的另一产物PAPS可被YND水解为无机磷酸和APS,完成底物APS的再生。利用瓦里安Cary4000分光光度计的kinetic程序,记录NADH的氧化速度,从而计算酶的活性。反应测定体系包括:50 mmol/L Hepes(pH值8.0);50 mmol/L KCl;1.0 mmol/L ATP;2 mmol/L MgCl2;0.25 mmol/L NADH ;10 U/mL PK;20 U/mL LDH;5 U/mL YND;0.3μmol/L大肠杆菌APSK或R68K;利用不同浓度APS(0.2,0.4,0.6,1.0,1.2,1.4μmol/L)起始反应,反应温度为25℃。平均3次试验结果,利用KaleidaGraph软件(Synergy software,USA)和双倒数作图方程Y=(Km/Kcat)X+1/Kcat拟合数据,获得KmAPS以及Kcat。
1.3.4 APSK及突变体R68K的AMP3K活性测定 APSK或者R68K突变体对AMP 3'羟基的磷酸化,产生PAP的同时生成1分子ADP。生成的PAP在YND的催化下可重新脱磷酸化再生成底物AMP。利用上述PK/LDH体系可计算AMP3K的活性。反应测定体系包括:50 mmol/L Hepes(pH值8.0);50 mmol/L KCl;1.0 mmol/L ATP;2mmol/LMgCl2;0.25mmol/LNADH;10 U/mL PK;20 U/mL LDH;5 U/mL YND;0.3μmol/L大肠杆菌APSK或者R68K;利用不同浓度AMP(1.5,2.5,5.0,7.5,10.0,12.5,15.0,17.5μmol/L)起始反应,反应温度为25℃。数据处理同1.3.3。
1.3.5 YND活性及KmPAP,Kcat的测定 YND催化PAP的水解生成AMP和无机磷酸。在肌激酶(myokinase,MK)的催化下,ATP磷酸化AMP生成2分子ADP,利用PK/LDH体系可计算ADP的生成速度,减半为AMP生成速度即PAP的水解速率。测定条件为:50 mmol/L Hepes(pH值8.0);50 mmol/L KCl;0.1μmol/L YND;1.0 mmol/L ATP;2 mmol/L PEP;2 mmol/L MgCl2;0.2 mmol/L NADH;5 U/mL MK;10 U/mL PK;20 U/mL LDH;0.5 mmol/LPAP起始反应,反应温度为25℃。
为测定YND水解PAP的动力学常数,可利用AMP3K活性磷酸化产物AMP,再生成底物PAP,从而维持YND活性测定体系中底物浓度稳定,同时消除产物的抑制效应。PK/LDH体系和AMP3K的联合应用,可较为方便进行反应初速度测定。R68K作为AMP3K加入YND的动力学常数测定体系,反应条件如下:50 mmol/LHepes(pH值8.0);50 mmol/L KCl;1.0 mmol/L ATP;2 mmol/LPEP;2 mmol/LMgCl2;0.2 mmol/LNADH;14μmol/L R68K;10 U/mL PK;20 U/mL LDH;5.5 nmol/L YND;利用不同浓度的PAP(0.1,0.2,0.4,0.6,1.0,1.4μmol/L)起始反应,反应温度为25℃。数据处理同1.3.3。
1.3.6 大肠杆菌APSK结构的模拟 将大肠杆菌APSK氨基酸序列递交至SWISS-MODEL workspace网站(http://swissmodel.expasy.org/),选择利用自动模拟方式,也可提供不同的APSK晶体结构(PDB:1D6J,1M7G,1M7H,3CR7,2GKS,3CR8,4FXP,3UIE,2OFX,2OFW 和1XNJ)作为模板。我们发现,利用不同模板,大肠杆菌APSK模拟结果基本一致。本研究以1XNJ为模板,模拟分析大肠杆菌APSK的三维结构。
2 结果与分析
2.1 APSK 对APS 和AMP 的磷酸化
APSK催化APS磷酸化生成PAPS和ADP,利用YND[12-13]作为偶联酶水解产物PAPS再生底物APS,利用PK磷酸化产物ADP再生底物ATP,方便APS磷酸化的反应初速度测定。大肠杆菌APSK的KmAPS和KmATP分别为0.2,10μmol/L[5-6]。在饱和ATP(1 mmol/L)的条件下,APSK的KmAPS和KcatAPS分别为1.78(0.33)μmol/L和22.9(3.1)/min(图2、表2)。
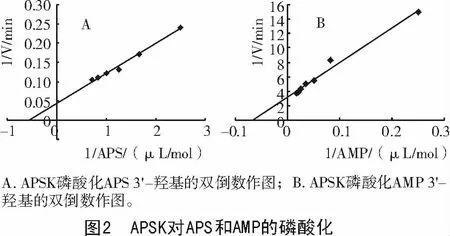
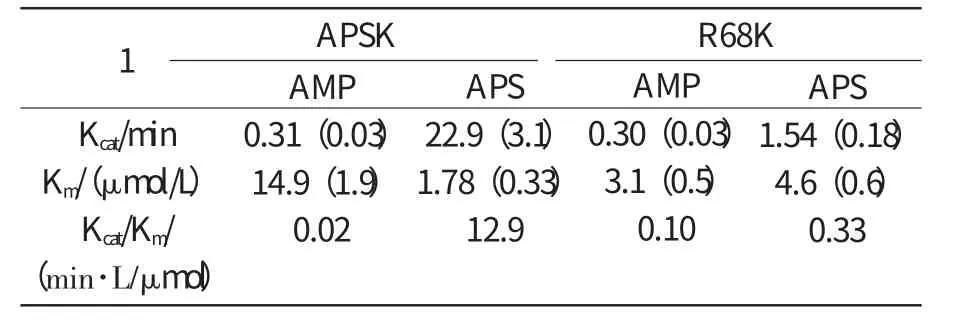
表2 APSK 和突变体R68K 催化磷酸化反应的Km 和Kcat 比较
利用YND水解PAP的3'磷酸基团再生底物AMP,以及PK/LDH测定体系分析APSK是否可催化AMP磷酸化生成PAP。在1 mmol/L ATP的条件下,APSK的KmAMP和KcatAMP分别为14.9(1.9)μmol/L,0.31(0.03)/min(图2、表2)。APSK的KcatAPS是KcatAMP的74倍,KmAPS是KmAMP的0.12倍。APS是APSK的最适底物,其催化效率是AMP的645倍(表2)。
2.2 R68K 突变的选择
通过分析已发表的APSK晶体结构(www.pdb.org),发现不同来源APSK序列中高度保守的R68可能和APS的硫酸根离子相互作用(图3),影响APS的亲和力。利用R68K的点突变,可能消除R68对APS结合的促进作用,而较短侧链的K侧链氨基则可能只与AMP或者APS的α-磷酸基团相互作用,从而将APSK转变为一种AMP 3'羟基激酶。利用突变引物(表1),对R68K进行突变,并经测序确认。经原核表达纯化获得相应R68K突变蛋白。

2.3 R68K 对APS 和AMP 的磷酸化
参照APSK磷酸化APS和AMP的测定条件,R68K的KmAPS和KcatAPS分别为4.6(0.6)μmol/L和1.54(0.18)/min(图4、表2)。R68K突变的KmAPS为对照的3倍,而KcatAPS降至对照的0.067倍。在1 mmol/LATP的条件下,分别测定R68K的KmAMP和KcatAMP,结果表明,KmAMP为3.1(0.5)μmol/L,KcatAMP为0.30(0.03)/min(图4、表2)。R68K突变导致其对AMP的亲和力是对照的5倍,而基本没有影响KcatAMP。因KmAPS增大至KmAMP的1.5倍,导致AMP成为R68K的最适底物。

2.4 YND 水解PAP 的动力学常数分析
YND催化PAP 3'-磷酸的水解产生AMP,而R68K的AMP3K活性可再生PAP(图1-A),在消除YND产物的同时,维持底物PAP浓度一定。我们利用R68K作为偶联酶,对YND的PAP水解活性进行测定(图5),获得与已发表YND数据[15]一致的结果,KmPAP和KcatPAP分别为0.25(±0.028)μmol/L和6.79(±0.44)/s。

3 讨论和展望
APSK催化APS的磷酸化形成PAPS,在硫酸盐同化代谢过程中具有重要的功能。前人已经对多个物种APSK的酶学特性进行了深入研究,青霉菌、拟南芥以及人的APSK的晶体结构已经解析(http://www.pdb.org)。不同来源的APSK动力学常数具有较大差异[5,27-31],如KmAPS为0.15~4.2μmol/L;KcatAPS为0.1~50/s。本研究发现,大肠杆菌APSK的KmAPS,KcatAPS分别为1.78μmol/L和22.9/min。
AMP作为APS的类似物,较APS少一个5'硫酸根离子。目前,尚没有APSK磷酸化AMP的报道。本研究结果表明,大肠杆菌APSK可磷酸化AMP,但其KcatAMP只有KcatAPS的0.013倍,亲和力也较APS低,说明AMP并非APSK的最适底物。细胞内AMP浓度为0.15~1 mmol/L[32-33],KmAMP为15μmol/L说明胞内AMP浓度对于AMP3K活性是饱和的。是否在胞内合成PAP?研究表明,PAP在胞内的累积会影响细胞的生长发育[12-15,17],并且AMP3K活性的大量存在会和ND活性形成一个浪费能量的无效循环,所以,APSK的AMP3K活性可能没有生物学功能,或者APSK的表达受到严紧调控。
分析模拟的APSK三维结构表明,R68通过和APS的α 磷酸根离子和β 硫酸根离子形成氢键,R82通过和APS嘌呤环的N1以及硫酸根离子形成氢键,以及F156和F76与APS嘌呤环形成芳香环交互作用(π-stacking),将APS分子固定在酶结合位点[29,34]。R68可同时和APS的磷酸根离子、硫酸根离子形成氢键,K侧链基团比R短2.4Å,将导致不能和距离较远的硫酸根离子相互作用,从而减弱APS的亲和力,而增加与磷酸根离子的相互作用,从而可能提高AMP的亲和力。通过初步的酶学分析表明,R68K突变体降低KmAMP至对照的0.2倍,同时APS磷酸化能力降低为对照的0.067倍。此位点不在催化中心,推测APS活化能力的降低可能由于K与APS的相互作用,导致其3'羟基的空间位置不是ATPγ 磷酸转移的最适位置所致。
R68K突变体使AMP磷酸化的催化效率是对照的5倍,AMP相对于APS成为最适底物。YND催化PAP水解产物为AMP,其Km值较低,约为0.7μmol/L[15]。AMP3K活性可作为YND底物PAP的再生偶联酶,维持稳态动力学分析中的底物浓度,同时消除其代谢产物,将反应状态一直维持在低产物形成的状态(反应初速度测定条件),有利于准确测定其反应速度。本研究利用R68K作为偶联酶,成功测定了YND的KmPAP为0.2μmol/L,与已有的研究结果[15]是一致的。
APSK具有AMP3K的活性,且通过R68K突变体提高其对AMP的催化效率,但仍然较低(0.1μL/(mol·min),所以R68K可认为是非高效的AMP3K。通过进一步分析其结构,将可能分子设计新的突变位点,提高蛋白对AMP的催化效率,为较低Km的YND动力学分析提供更加有效的偶联酶工具。
[1]Leyh T S.The physical biochemistry and molecular genetics of sulfateactivation[J].Crit Rev Biochem Mol Biol,1993,28(6):515-542.
[2]Mitchell S.Biological interactions of sulfur compounds[M].London:Taylor and Francis,1996:20-41.
[3]Foster B A,Thomas SM,Mahr JA,et al.Cloning and sequencing of ATP sulfurylase from Penicillium chrysogenum:Identification of a likely allosteric domain[J].JBiol Chem,1994,269(31):19777-19786.
[4]Sun M,Leyh T S.Anatomy of an energy-coupling mechanism:the interlocking catalytic cycles of the ATP sulfurylase-GTPase system[J].Biochemistry,2005,44(42):13941-13948.
[5]Satishchandran C,Markham GD.Adenosine-5'-phosphosulfate kinase from Escherichia coli K12.Purification,characterization,and identification of aphosphorylated enzymeintermediate[J].J Biol Chem,1989,264(25):15012-15021.
[6] Satishchandran C,Markham G D.Mechanistic studies of Escherichia coli adenosine-5'-phosphosulfate kinase[J].Arch Biochem Biophys,2000,378(2):210-215.
[7]Mendoza-Cozatl D,Loza-Tavera H,Hernandez-Navarro A,et al.Sulfur assimilation and glutathione metabolism under cadmium stress in yeast,protists and plants[J].FEMS Microbiol Rev,2005,29(4):653-671.
[8]王艳兴,杨玲,孙梅好.硫酸盐活化复合体的分类及其功能[J].生命的化学,2011,31(2):252-257.
[9]Wang Y,Yang L,Sun M.Classification and functions of sulfate activating complexes[J].Chemistry of Life.2011,31(2):252-257.
[10]Liu MC,Yasuda S,Idell S.Sulfation of nitrotyrosine:biochemistry and functional implications[J].IUBMB Life,2007,59(10):622-627.
[11] Neuwald A F,Krishnan B R,Brikun I,et al.cysQ,a gene needed for cysteine synthesis in Escherichia coli K-12 only duringaerobic growth[J].JBacteriol,1992,174(2):415-425.
[12]Murguia JR,Belles JM,Serrano R.Theyeast HAL2 nucleotidase is an in vivo target of salt toxicity[J].J Biol Chem,1996,271(46):29029-29033.
[13]Murguia J R,Belles J M,Serrano R A.Salt-sensitive 3'(2'),5'-bisphosphate nucleotidase involved in sulfate activation[J].Science,1995,267:232-234.
[14]Albert A,Yenush L,Gil-Mascarell M R,et al.X-ray structure of yeast Hal2p,a major target of lithium and sodium toxicity,and identification of framework interactions determining cation sensitivity[J].JMol Biol,2000,295(4):927-938.
[15]Spiegelberg B D,Xiong JP,Smith JJ,et al.Cloning and characterization of a mammalian lithium-sensitive bisphosphate 3'-nucleotidase inhibited by inositol 1,4-bisphosphate[J].J Biol Chem,1999,274(19):13619-13628.
[16]Aggarwal M,Kishan K V,Mondal A K.Creation of salt-insensitive 3'(2'),5'-bisphosphate nucleotidase by modeling and mutagenesis approach[J].Arch Biochem Biophys,2008,469(2):174-183.
[17] Mechold U,Ogryzko V,Ngo S,et al.Oligoribonuclease is a common downstreamtarget of lithium-induced pAp accumulation in Escherichia coli and human cells[J].Nucleic Acids Res,2006,34(8):2364-2373.
[18]Whittemore RM,Pearce L B,Roth JA.Purification and kinetic characterization of a dopamine-sulfating form of phenol sulfotransferase from human brain[J].Biochemistry,1985,24(10):2477-2482.
[19]Ramaswamy S G,Jakoby W B.(2')3',5'-Bisphosphate nucleotidase[J].JBiol Chem,1987,262(21):10044-10047.
[20]Yang L M,Fernandez M D,Lamppa G K.Acyl carrier protein(ACP)import into chloroplasts.Covalent modification by a stromal holoACPsynthase is stimulated by exogenously added CoA and inhibited by adenosine 3',5'-bisphosphate[J].Eur J Biochem,1994,224(2):743-750.
[21]Dichtl B,Stevens A,Tollervey D.Lithium toxicity in yeast is duetotheinhibition of RNA processingenzymes[J].EMBOJ,1997,16(23):7184-7195.
[22]Lu SY,Zhao GR,Wu A M,et al.Molecular cloningof acotton phosphatase gene and its functional characterization[J].Biochemistry(Mosc),2010,75(1):85-94.
[23] Lopez-Coronado J M,Belles J M,Lesage F,et al.A novel mammalian lithium-sensitive enzyme with a dual enzymatic activity,3'-phosphoadenosine 5'-phosphate phosphatase and inositol-polyphosphate 1-phosphatase [J].J Biol Chem,1999,274(23):16034-16039.
[24]Peng Z,Verma D P.A rice HAL2-like gene encodes a Ca(2+)-sensitive 3'(2'),5'-diphosphonucleoside 3'(2')-phosphohydrolase and complements yeast met22 and Escherichia coli cysQmutations[J].JBiol Chem,1995,270(49):29105-29110.
[25]Wei J,Tang QX,Varlamova O,et al.Cysteine biosynthetic enzymesarethepiecesof ametabolic energy pump[J].Biochemistry,2002,41(26):8493-8498.
[26]Sun M,Leyh TS.Channelingin sulfateactivatingcomplexes[J].Biochemistry,2006,45(38):11304-11311.
[27]Malcovati M,Valentini G.AMP-and fructose 1,6-bisphosphate-activated pyruvate kinases from Escherichia coli [J].Methods Enzymol,1982,90:170-179.
[28] Lillig C H,Schiffmann S,Berndt C,et al.Molecular and catalytic properties of Arabidopsis thaliana adenylyl sulfate(APS)-kinase[J].Arch Biochem Biophys,2001 392(2):303-310.
[29]Lansdon EB,Fisher A J,Segel IH.Human 3'-phosphoadenosine 5'-phosphosulfate synthetase(isoform 1,brain):kinetic propertiesof theadenosine triphosphatesulfurylaseand adenosine 5'-phosphosulfate kinase domains [J].Biochemistry,2004,43(14):4356-4365.
[30]Hanna E,MacRae IJ,Medina D C,et al.ATPsulfurylase from the hyperthermophilic chemolithotroph Aquifex aeolicus[J].Arch Biochem Biophys,2002,406(2):275-288.
[31]Gay SC,Fribourgh JL,Donohoue PD,et al.Kinetic properties of ATPsulfurylaseand APSkinase from Thiobacillus denitrificans[J].Arch Biochem Biophys,2009,489(1/2):110-117.
[32] Bochner B R,Ames B N.Complete analysis of cellular nucleotides by two-dimensional thin layer chromatography[J].J Biol Chem,1982,257(16):9759-9769.
[33]Polakis ES,Bartley W.Changes in theintracellular concentrations of adenosine phosphates and nicotinamide nucleotides during the aerobic growth cycle of yeast on different carbon sources[J].Biochem J,1966,99(3):521-533.
[34]Ravilious GE,Jez JM.Nucleotide binding site communication in Arabidopsis thaliana adenosine 5'-phosphosulfate kinase[J].JBiol Chem,2012,287(36):30385-30394.

