细角螺微卫星DNA富集文库构建及特征分析
李清荟, 陈晓姣, 黎中宝, 曹媛钰, 陈丽娜, 戴 刚
(1. 集美大学 水产学院, 福建 厦门 361021; 2. 福建省海洋渔业资源与生态环境重点实验室, 福建 厦门 361021)
细角螺(Hemifusus ternatanus)俗称响螺, 单壳类海产底栖动物, 属腹足纲(Gastropoda), 新腹足目(Neogastropoda), 盔螺科(Galeodidae), 角螺属(Hemifusus)。主要分布于中国东南沿海, 新加坡和日本海域。其肉肥, 美味, 具有滋补功能, 经济价值很高, 是高档海珍品。近年来, 海域生态环境的恶化以及人类过度捕捞, 细角螺资源日趋衰减。为了防止细角螺进一步衰减, 人们将焦点转向细角螺的人工育苗及繁殖生物学的研究上, 并且取得初步成功[1], 但仍处在实验探索阶段[2-3]。由于目前对细角螺种质资源现状了解甚少, 因此对细角螺进行种质资源结构、遗传多样性等方面的研究, 这将对保护该物种的野生群体, 开展苗种繁育、良种培育等工作具有重要的意义。
微卫星(microsatellites)DNA, 又称简单序列重复(simple sequence repeats, SSR)或短串联重复序列(short tanderm reapeats, STR), 由中间的核心序列与其两端的保守侧翼序列组成, 核心序列为1~6个核苷酸为重复单元的串联重复DNA序列。由于微卫星分子标记具有多态性丰富, 稳定性好, 符合孟德尔遗传和具有共显性[4]等特点, 自1984年被Tautz等[5]首次提出可用于群体遗传学研究后, 该技术就被广泛应用于动植物的遗传多样性, 遗传变异, 遗传连锁分析和遗传图谱构建等方面。如斜带石斑鱼(Epinephelus coioides)[6], 大珠母贝(Pinctada maxima)[7]及 许 氏 平 鲉 (Sebastes schlegeli)[8]等 都 得 到很好的应用。细角螺作为重要的经济贝类之一, 目前有关细角螺微卫星分子标记的研究尚未见报道。本研究采用磁珠富集法构建细角螺微卫星富集文库, 并分析微卫星序列的特征, 为进一步筛选大量的微卫星引物奠定基础, 进而为研究细角螺遗传多样性, 遗传图谱构建, 人工育苗等提供一定的基础资料。
1 材料和方法
1.1 实验材料
细角螺样品采自福建省泉州市惠安县。
1.2 基因组DNA提取与酶切
根据上海生工生物工程技术有限公司的基因组小量抽提试剂盒(SK1252)提供的步骤, 稍做修改提取细角螺基因组DNA。所提DNA经1%的琼脂糖凝胶电泳检测后于-20℃保存备用。用MseI酶切基因组DNA, 选取长度为300~1200bp的酶切片段进行连接。
1.3 接头制备与连接
等体积混合与MseI酶切部位相匹配的Linker A(5'-GACGATGAGTCCTGAG-3')及LinkerB (5'-TAC TCAGGACTCAT-3'), 95℃变性10 min, 37℃10min, 最后冰置10min, 形成终浓度为10 μmol/L的双链接头待用。
连接体系30 μL: 酶切产物20μL, 接头5μL(10 μmol/L), T4连接酶(5 U/μL)1μL, 10×T4 buffer 4 μL于37℃连接3.5h或22℃连接过夜。
1.4 探针杂交
杂交反应前, 先将2×Hyb buffer(杂交缓冲液)预热至 55℃备用, 将接头连接产物95℃变性10 min。取变性后的连接产物与 bio-(CT)15、bio-(GT)15混合探针进行杂交反应, 杂交反应体系为50 μL,其中包括25 μL 2×Hyb buffer, 10 μL接头连接产物, 5 μL Bio-(CT)15(10μmol/L), 5 μL Bio-(GT)15(10 μmol/L) 以及5 μLddH2O。61℃反应1h后, 放置4℃备用。
1.5 磁珠的平衡
取150 μL磁珠原液于1.5 mL离心管中, 混匀后置于磁力架上捕获磁珠, 小心吸走上清液。用150μL(等比例)0.5×SSC洗涤磁珠两次, 然后将磁珠重悬于150 μL的6×SSC, 0.1% SDS中。
1.6 磁珠富集及精制
将50 μL杂交产物与重悬磁珠混匀, 室温下温育30 min, 期间以每 1~2 min翻转1次, 以使生物素和链霉亲和素结合, 30 min后将离心管置于磁力架上, 吸去上清液。然后将离心管放置于磁力架上洗涤。室温下, 用400 μL洗液I(2×SSC, 0.1% SDS)洗脱2次, 400 μL洗液Ⅱ(1×SSC, 0.1% SDS)在室温下洗脱2次, 再用洗液Ⅱ于50℃洗脱两次, 将不含微卫星的序列除去, 然后用200 μL的0.1×TE于95℃温育10 min后迅速吸取上清液至新管。加入400 μL经-20℃预处理的无水乙醇, 并加入 20 μL NaAc/EDTA(pH8.0)螯合剂, 冰上静置15 min后冷冻离心, 弃乙醇, 加入500 μL经-20℃预处理的75%乙醇, 冰上放置15 min后冷冻离心10 min, 弃乙醇, 风干样品。最后加入25 μL TE溶液, 放置4℃过夜, 使微卫星DNA充分溶解于TE溶液中。
1.7 PCR扩增及纯化
以寡聚核苷酸链MseIadapterA为引物对上述精制到的微卫星DNA片段进行PCR扩增, 25 μL反应体 系 包 括 10×Buffer(含 Mg2+) 4 μL, dNTP (10mmol/L)0.4 μL, Taq聚 合 酶(5U/μL), 0.2 μL,MseI-A引物1.4 μL (10 μmol/L), 17 μL ddH2O, 精制产物2 μL。PCR反应条件为: 95℃预变性2 min, 20×(95℃ 30 s, 58℃ 30 s, 72℃ 60 s), 最后72℃保持10 min完成扩增片段的末端加尾A。用0.1%琼脂糖检测PCR扩增产物的片段大小。产物用GenClean PCR回收试剂盒(上海捷瑞)纯化以除去多余的dNTPs及接头等杂质。
1.8 微卫星DNA片段克隆、筛选和序列分析
将纯化的PCR产物用pMD19-T载体于16℃连接4h。用感受态大肠杆菌DH5α进行转化克隆, 得到细角螺微卫星文库。挑取单克隆于LB液体培养基中, 37℃振荡培养过夜。用M13F/R通用引物对单克隆菌进行PCR检测, 挑取片段大于等于500 bp的阳性克隆的菌液送至华大基因测序。
2 结果
2.1 克隆及测序结果
经PCR筛选, 在354个阳性克隆中有248个含有大小合适的插入片段, 片段大小为500~1000bp, 从中随机挑选220个克隆送出测序。图1为部分筛选结果的电泳图。

图1 单克隆检测结果 Fig. 1 Result of the monoclonal detection
依据Linda等[9]对微卫星的划定标准: 单核苷酸重复大于15次, 二核苷酸重复大于7次, 三核苷酸大于5次, 四核苷酸及五核苷酸大于4次。序列比对后用SSR hunter软件进行微卫星序列的查找, 结果表明在成隆功测序的220个阳性克隆中, 除5个不含有微卫星序列外, 在剩余的215个克中共得到278个重复次数在5以上的微卫星序列。依据Weber制定的标准[10], 在本研究筛选到的278个细角螺微卫星序列中, 完美型171个, 占61.5 %, 非完美型80个, 占29.1%, 复合型26个, 占9.4%(表1)。除探针使用的GT和CT的重复序列外, 还筛选到三碱基GTT、TGG、GAA、CAA; 四碱基TCTA、ACAG、TAGA、GTGA、GTCT、GATA及五碱基TTTTG的重复序列, 具体统计见表2。
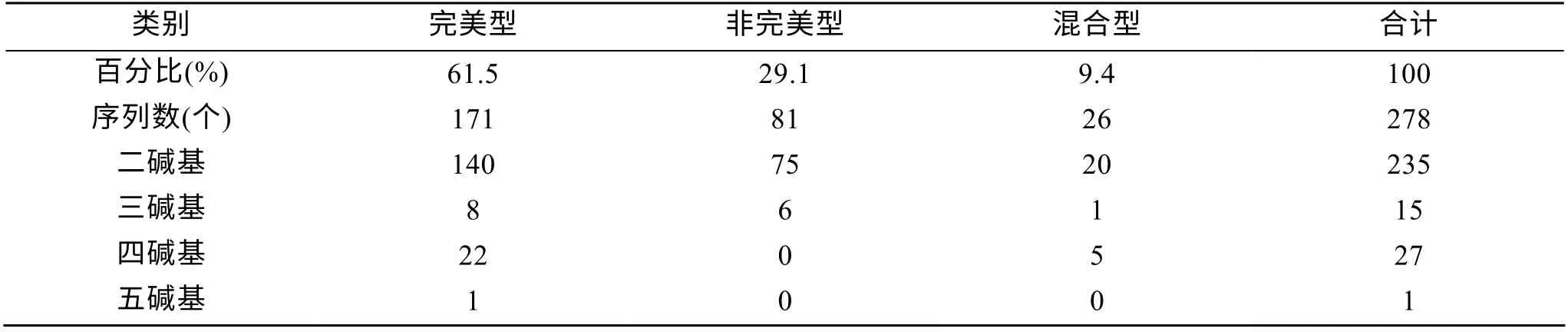
表1 细角螺基因组微卫星序列的分类情况 Tab.1 Classification of microsatellites for H. ternatanus
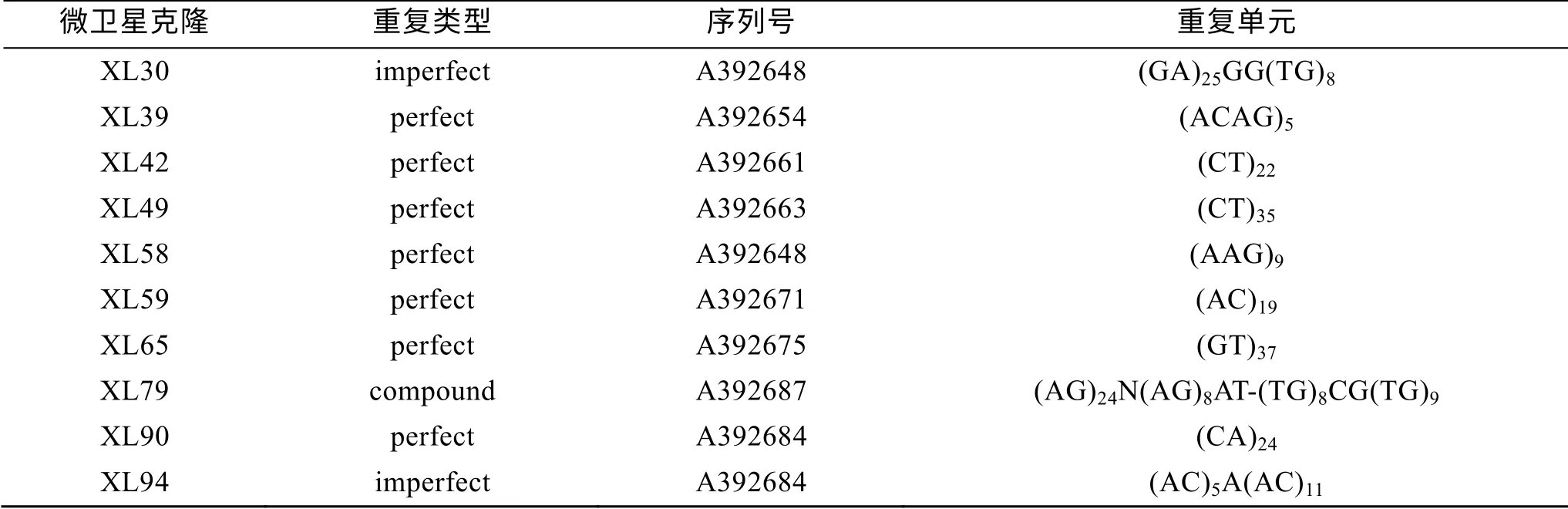
表2 部分微卫星序列的重复单元、重复类型及序列号情况 Tab.2 The repeat motifs, category and genebank accession number of part of microsatellite sequences
2.2 克隆测序峰图
在测序的220个阳性克隆中, 有156个单向测序不能测通, 占所有克隆的70%, 需再用载体的反向引物来测序,才能测序成功。并且这部分的测序结果显示, 在单个克隆中有多处微卫星重复序列, 有的甚至高达6处, 并且出现复杂的碱基重复。这可能是导致测序过程出现信号衰减, 测序峰图紊乱的原因。图2为单向测序测通的某克隆部分峰图。

图2 XL46位点部分微卫星测序峰图 Fig. 2 Part of the microsatellite sequencing peak figure in XL46 loci
3 讨论
被用于微卫星分子标记的生物素探针有多种, 可根据研究对象的特点来选择相应的探针。在所有动物基因组中(CA)n微卫星的含量最为丰富, 其次是(CT)n微卫星[11], 故本实验在用(GT)15探针的基础上又采用了(CT)15探针, 以期获得更多的标记位点。在真核生物基因组中, 双核苷酸重复的微卫星DNA最为丰富, 三核苷酸重复的微卫星DNA比双核苷酸重复微卫星DNA的含量低10倍, 四核苷酸重复的微卫星DNA与之相比, 含量更少[12]。本研究的结果显示细角螺基因组DNA中二碱基重复的微卫星序列(84.53%)含量最高, 这与研究报告的结论相一致, 而四碱基重复的微卫星序列(9.71%)比三碱基重复的微卫星序列(5.4%)所占的比例更高, 与报告过的研究结果相反。原因可能是杂交时采用(GT)15、(CT)15混合探针, 导致在总的278个微卫星片段中出现多达21个GTCT以及与之相互补的CAGA重复序列。本研究获得的细角螺微卫星中, 完美型所占比例为61.7%, 与虾夷扇贝(Patinopecten yessoensis) 50.18%[13], 企鹅珍珠贝(Pteria penguin)65.5%[14], 合浦珠母贝(Pinctada martensii)77.3%[15], 大珠母贝(Pinctada maxima)63%[7]及西施舌(Coelomactra antiquate)68.4%[16]等贝类基本相似, 都是完美型所占的比例更高, 而混合型所占的比列(9.4%)与李琪等[17]研究的长牡蛎(Crassostrea gigas)的混合型比例(24.2%)相差很大。这些不同的结果与研究方法和筛选富集得到的不同微卫星区域有关, 同时也在一定程度上反映了贝类基因组微卫星的特性。
微卫星核心序列突变率相对较高, 造成了微卫星核心序列重复次数的变化, 这是微卫星多态性的基础[18]。Valdes[19]认为重复次数低于5 的微卫星几乎检测不出多态性, 一般微卫星重复次数程度与多态性程度成正相关[20]。本实验所得序列重复次数多集中在7~30, 占62%, 最高达到64次。理论上讲, 本研究所得的细角螺微卫星序列中,应该有约60%的序列可以设计的引物。但是由于一条克隆中出现多处微卫星序列, 最高高达6处, 并且出现复杂的碱基重复, 导致测序峰图紊乱以及引物设计困难, 在278条序列中仅有82条可以设计引物, 并且质量较高的仅有50条。原因可能是在富集过程中多次洗涤磁珠, 使得一个克隆里有多个重复序列片段, 保守序列碱基数少, 导致引物设计和筛选困难。另一个原因可能是细角螺基因组中的微卫星本来就比较复杂, 与多数贝类遗传背景一样, 在进化过程中发生染色体重排[21],导致基因组结构复杂和存在多个位点,从而对引物的设计及筛选产生影响。
目前国内对细角螺的研究主要集中在幼苗繁育[1-2]及生理生化上[22], 国外主要集中在幼螺生长的生态习性[23]、神经鞘脂类的组成和结构[24-27]、体内的砷分布[28], 对其遗传多样性的研究较少, 仅在核型特征[29]方面有所报道。本研究构建细角螺进行微卫星富集文库及分析其序列组成, 为进一步筛选大量的微卫星引物奠定基础, 以期为保护细角螺野生群体、开展苗种繁育, 种苗放流及良种培育等工作提供理论参考的目的。
[1] 洪国莲.细角螺人工育苗技术[J].福建水产, 2010, 2: 30-32.
[2] 许章程, 金亮, 宋普庆, 等.温度和盐度与细角螺幼体生存、生长及发育的关系[J]. 台湾海峡, 2009, 28(2): 266-271.
[3] 许章程, 王初升, 张玉生. 细角螺的繁殖生态条件及繁殖习性[J]. 水产学报, 2006, 30(6):848-851.
[4] Wang Z, Weber J L, Zhong G, et al. Survey of plant short tandem DNA repeats[J]. Theor Appl enet, 1994, 88(1): 1-6.
[5] Tautz D. Hypervariabflity of simple sequences as a general source for polymorphic DNA markers [J]. Nucleic AcidsResearch, 1989,17(16):6463-6467.
[6] 王家祺, 郭 丰, 丁少雄, 等. 斜带石斑鱼不同地理群体遗传变异的微卫星分[J]. 海洋科学, 2009, 33(11): 60-64.
[7] 谷龙春, 黄桂菊, 何毛贤, 等. 大珠母贝两个野生群体遗传多样性的微卫星分析[J]. 渔业科学进展, 2009, 33(4): 96-101.
[8] 王文琪, 张 毅, 刘梦侠, 等. 鲉许氏平 4 个野生群体遗传多样性微卫星分[J]. 海洋科学, 2012, 36(1): 10-16.
[9] Linda C, Luke R, Dan M, et al. Computational and experimental characterization of physically clustered simple sequence repeats in plants [J]. Genetics, 2000, 156(2): 847-854.
[10] Weber J L. Informativeness of human (dC-dA)n (dG-Dt)n ploymorphisms[J]. Genomics, 1990, 7(3): 524-530.
[11] Brenner S, Elgar G, Sandford T, et a1.Characterization of thepufferfish(Fugu) genome as a compact model vertebrate genome[J].Nature, 1993, 366: 265-268.
[12] Ma Z Q, Rder M, Sorrells M E. Frequencies and sequence characteristics of di-, tri-, and tetra-nucleotide microsatellites inwheat[J]. Genome, 1996, 39(1): 123-130.
[13] 赵莹莹, 朱晓琛, 孙效文, 等 . 磁珠富集法筛选虾夷扇贝微卫星序列[J]. 中国水产科学, 2006, 13(5): 749-755.
[14] 许友卿, 肖群平, 李咏梅, 等. 企鹅珍珠贝微卫星分子标记的筛选[J]. 水生态学杂志, 2011, 32(3): 88-93.
[15] 曲妮妮, 龚世园, 黄桂菊, 等. 基于FIASCO 技术的合浦珠母贝微卫星标记分离与筛选研究[J]. 热带海洋学报, 2010, 29(3): 47-54.
[16] 朱立静, 孙中响, 沈颂东, 等. 磁珠富集法筛选西施舌微卫星序列[J]. 南方水产科学, 2011, 7(4): 10-15.
[17] 李琪, 木岛明博. 长牡蛎(Crassostrea. gigas)微卫星克隆快速分离及特性分析[J]. 海洋与湖沼, 2004, 35(4): 364-370.
[18] 刘志毅, 相建海. 微卫星DNA 分子标记在海洋动物遗传分子中的应用[J]. 海洋科学, 2001, 25(6): 11-13.
[19] Valdes A M. Allele frequencies at microsatellite loci:the stepwise mutation model revisited[J]. Genetics, 1993, 133(3): 737-749.
[20] Rico C, Ibrahim K M, Rico I, et al. Stock composition North Atlantic populations ofwhitingusing microsatellite markers [J]. Journal of Fish Biology, 1997, 51: 462-475.
[21] Wang Y, Guo X. Chromosomal rearrangement in Pectinidae revealed by rRNA loci and implications for bivalveevolution[J]. Biological Bulletin, 2004, 207(3): 247-256.
[22] 梁海鹰, 曹伏君, 张亮珠, 等. 细角螺几种组织的同工酶分析[J]. 吉首大学学报(自然科学版), 2007, 28(5): 87-91.
[23] Kishineh, Hayashi A, Morita M. Sphingolipids ofHemifusus ternatanusⅣ[J]. Nihon Yuka Gakkai NenkaiKoen Yoshishu,2001, 40(1): 69.
[24] Mishima Y, Kishima H, Hayashi A. Sphingolipids ofHemifusus ternatanusCeramide Monohexosides and Ceramide Aminoethylphosphonate of the Viscera in Japanese [J]. Yukagaku, 1997, 46(1): 39-49.
[25] Hamada S. Growth and feeding ofHemifususternatanus in early crawling stage[J]. Japanese Journal of Malacology, 1974, 33(2): 75-79.
[26] Hayashia D, Kishine H. Comparative biochemical studies on lipids of gastropods(mollusca): Structures and compositions of long-chain bases of sphingolipids[J]. The Japanese Journal of Malacology, 1997, 56(2): 157-167.
[27] Kishine H, Mishima Y, Hayashi A. Structural determination of long-chain bases of ceramide aminoethylphosphonate,Hemifusus ternatanus[J].Yukagaku, 1995, 44(11): 977-984.
[28] Phillips D J, Epledgem H D. Dstribution of inorganic and total arsenicin tissues of the marine gastropodHemifusus ternatanus[J]. Marine Ecology Progress Series, 1986, 34: 261-266.
[29] 曹伏君, 李长玲, 罗杰, 等. 管角螺、细角螺的核型研究[J]. 广东海洋大学学报, 2008, 28(1): 15-18.

