miR-219基因及其靶基因CAMK2G多态性与精神分裂症的关联研究☆
张燕霞 汪栋祥 李华芳 傅迎美 禹顺英
目前的研究表明,N-甲基-D-天冬氨酸受体功能障碍与精神分裂症密切相关。近几年研究发现微小核糖核苷酸-219(microRNA-219,miR-219)可在受体(N-甲基-D-天冬氨酸受体,N-methyl-D-aspartate recepter,NMDAR)[1]和第二信使(钙离子/钙调蛋白依赖性蛋白激酶IIγ,Ca2+/calmodulin-dependent protein kinase II gamma,CAMK2G)[2]水平抑制NMDAR介导的谷氨酸信号(简称NMDAR信号),因此miR-219可能参与该信号的传导。另外,有研究发现微小核糖核苷酸(microRNA,miRNA)编码基因及其靶基因mRNA 3’端非编码区(3’untranslated regions,3’UTR)的多态性变化能够影响miRNA的转录加工过程及miRNA/mRNA之间的相互作用[3-4],本项研究假设位于miR-219编码基因序列及其靶基因CAMK2G 3’UTR区上单核苷酸多态性(single nucleotide polymorphism,SNP)可能会影响精神分裂症发生风险,故通过病例对照的关联研究来探讨上述基因多态与精神分裂症的关系。
1 对象与方法
1.1 研究对象 主要来自2006年1月至2011年12月于上海市精神卫生中心门诊和住院的精神分裂症患者,入选标准:①符合《美国精神障碍诊断统计手册第4版》(Diagnostic and Statistical Manual of Mental Disorders,Fourth Edition,DSM-IV)精神分裂症诊断标准;②性别不限、年龄≤65岁;③汉族;④签署知情同意书。排除标准:①符合除了精神分裂症外的DSM-IV轴I诊断;②有严重躯体疾病者。本研究共纳入精神分裂症患者1041例,男531例,女510例,年龄12~65岁,平均(33.65±9.14)岁;健康对照组来自上海市精神卫生中心的健康职工以及健康志愿者,无精神疾病史,无重大躯体疾病,共953例,男512例,女441例,年龄19~64岁,平均 (34.47±10.49)岁。
1.2 研究方法
1.2.1 基因组DNA提取 采集患者及健康对照者静脉血各2 mL,EDTA抗凝,采用基因组DNA试剂盒(北京天根生物有限公司生产)提取基因组DNA。
1.2.2 SNP的选择 首先通过HapMap或NCBI db-SNP数据库在汉族人群中查找miR-219基因的前体转录产物(precusor microRNA-219,pre-miR-219)及其侧翼序列+/-500bp区域内的最小等位基因频率(Minor Allele Frequency,MAF)≥0.1的SNPs,结果发现一个SNP rs107822(T>C),其位于pre-miR-219-1编码序列-37bp,MAF=0.433。其次利用 Patrocles数据库(http://www.patrocles.org/)查找CAMK2G基因3’UTR内位于预测的miRNAs靶向位点上的SNPs,并且同样要求MAF≥0.1,结果发现了一个可能具有功能意义的SNP,即 rs2306327,MAF=0.102。
1.2.3 基因分型 采用TaqMan探针SNP基因分型技术,对rs107822、rs2306327进行分型。引物和探针由美国应用生物系统公司定制,PCR反应试剂TaqMan UniversalPCR MasterMix由ABI公司提供。PCR反应体系为5µL,包括模板10 ng,2×Taq-Man PCR MasterMix 2.5µL,20×SNP Assay(包括引物和FAM/VIC探针)0.25µL。反应条件为95℃10 min;(92℃15 s,60℃1 min)×40个循环。PCR反应和荧光信号读取均在ABI7900HT Sequence Detector(384孔板)上完成,以SDS 2.0图像分析软件Allelic Discrimination程序进行终点分析,通过检查不同等位基因所标的FAM和VIC荧光强度,判断待测样本为野生纯合子、突变纯合子或杂合子。
为了进行质量控制,分型结束后从整个样本中随机抽取了5%的样本进行了重复检测,结果一致性达到了98%。
1.3 统计学方法 首先,通过SHEsis软件(http://analysis.bio-x.cn/SHEsisMain.htm)进 行 Hardy—Weinberg(HW)吻合度检验以及等位基因频率的统计,通过χ2检验检测等位基因在病例组和对照组中的分布差异[5]。然后,利用SNPstats在线软件(http://bioinfo.iconcologia.net/snpstats/start.htm)的多因素Logistic回归模型评估在共显性、显性、隐性和加性4种遗传模式中rs107822及rs2306327基因型与精神分裂发生风险的相关程度,并且利用似然比检验(likelihood ratio test,LRT)进行两模型间的拟合优度检验,即通过与一个简单的模型(共显模型)比较,评估另一个相对复杂的模型(如显性、隐性、加性遗传模型)是否更适合当前数据分析,同时根据阿凯克信息准则(Akaike information criteria,AIC)进行最佳模型的选择,即AIC值越小说明模型越精确。
1.4 二级结构预测利用CentroidFold软件[6](http://www.ncrna.org/centroidfold)预测rs107822位点对miR-219的初级转录产物(primary microRNA-219,pri-miR-219)二级结构的影响。
2 结果
2.1 Hardy-Weinberg平衡检验 通过HW平衡检验发现位点rs107822和rs2306327均符合HW平衡(P值分别为0.746和0.995),因此样本具有一定的群体代表性。
2.2 miR-219及CAMK2G基因多态性rs107822和rs2306327在患者组与对照组中的等位基因分布比较 miR-219基因多态位点rs107822等位基因分布在病例组和对照组之间差异存在统计学意义,C等位基因在对照组中的分布频率高于患者组,分别为0.399 和 0.357(χ2=7.33,P=0.007)。CAMK2G基因多态位点rs2306327等位基因分布频率在患者组与对照组分别为0.108和0.109,但两组间差异无统计学意义(χ2=0.00,P=0.978),见表1。
2.3 miR-219及CAMK2G基因多态性rs107822和rs2306327在4种遗传模式(共显性、显性、隐性、加性)下的非条件性Logistic回归分析 利用在线的SNPstats对两组的基因型进行非条件Logistic回归模型分析,表2给出了4种遗传模式(共显性、显性、隐性、加性)的Logistic回归分析结果,以及OR值,95%CI,AIC值。AIC值越小说明模型越精确。miR-219编码基因多态位点rs107822的基因型分布符合共显性(P=0.027,AIC=2703.7)、显性(P=0.015,AIC=2703.0)及加性(P=0.0072,AIC=2701.7)遗传模式。根据AIC信息准则,加性遗传模式为最佳模型(AIC值较小),随着等位基因C拷贝数的增加可降低疾病发生风险(OR=0.84,95%CI=0.74~0.95)。然而Logistic回归分析结果未发现CAMK2G基因的多态位点rs2306327与精神分裂症之间的相关性,见表2。
2.4 miR-219基因多态性rs107822对pri-miR-219二级结构的影响 图1是CentroidFold在线软件对pri-miR-219二级结构的预测示意图,软件预测发现当miR-219基因多态性rs107822为T等位基因(即U等位)时,其相应的二级结构较之C等位具有更小的自由能,说明前者较后者更稳定,并且当T等位(即U等位)变成C等位后,该位点附近的二级结构发生了改变,如图1所示,在位点上游增加了一个小的环状结构,而下游的两个小环状结构合成了一个大环,非环状结构变成了环状结构。因此rs107822可能会影响pri-miR-219二级结构的稳定性。

表1 miR-219及CAMK2G基因多态性rs107822和rs2306327的等位基因在患者组与对照组中的分布
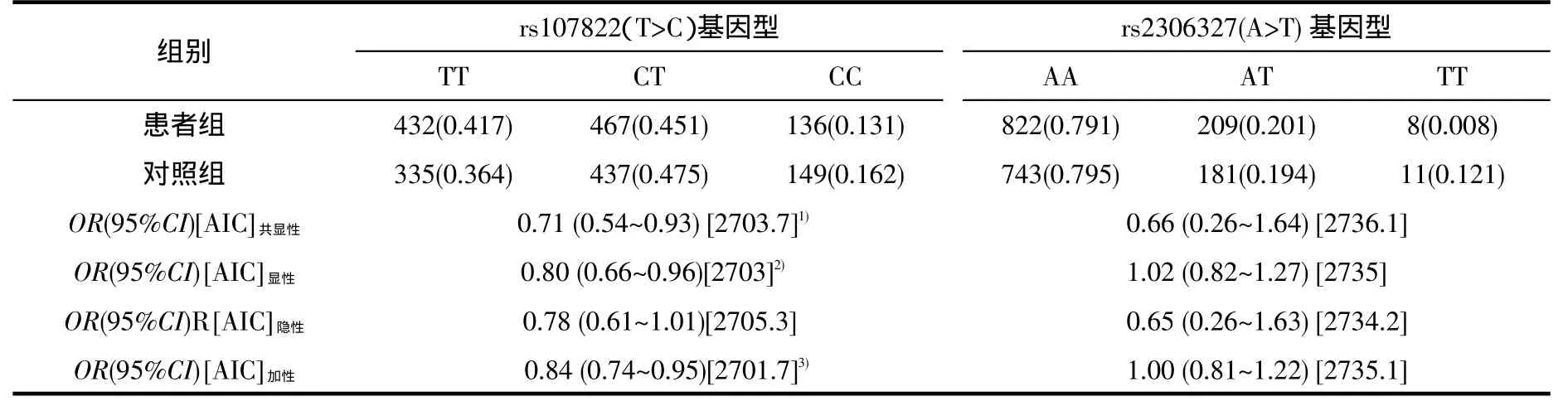
表2 miR-219基因rs107822和CAMK2G基因rs2306327在4种不同遗传模式下的的非条件性Logistic回归分析结果
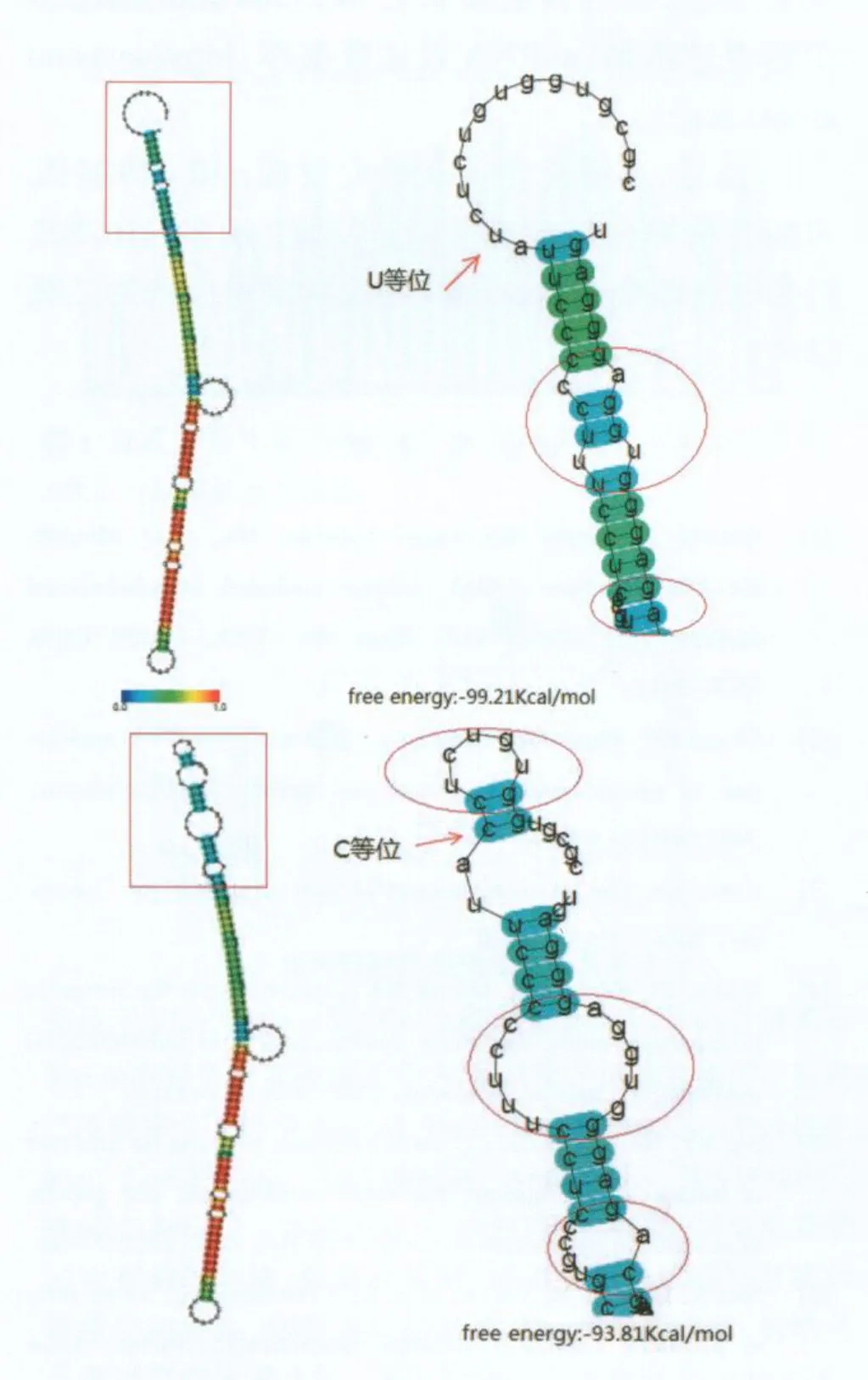
图1 rs107822等位基因T(即U等位)(A)及C(B)对pri-miR-219二级结构的影响。红色箭头指示的是rs107822所在的位置,红圈表示两结构之间的不同之处。
3 讨论
本研究通过关联分析发现miR-219基因多态性rs107822(T>C)可能与精神分裂症有关,其等位基因分布在病例组和对照组之间差异存在统计学意义,在加性遗传模式下,等位基因C能以加性(additive)形式降低疾病的发生风险,也就是说含有双拷贝的C等位基因的纯合子基因型CC降低疾病的发生风险是含有单拷贝C等位基因的杂合子基因型TC的两倍,故而携带rs107822 C等位基因能降低疾病发生风险(OR=0.84,95%CI=0.74~0.95)。
miR-219基因位于精神分裂症的易感染色体区域6p21区[7],并且在大脑内有着丰富表达[8]。据Beveridge等[9]的尸脑研究报道,miR-219在精神分裂症患者背外侧前额叶区是显著上调的。动物研究发现抑制NMDAR功能能显著下调小鼠前额叶miR-219水平;当抑制正常小鼠脑内miR-219表达时,则会上调其靶基因CAMK2G表达,该基因编码蛋白是NMDAR介导的一个下游重要信号蛋白,其异常表达可导致NMDAR信号的障碍,引起正常小鼠相关行为的改变,如出现刻板行为,运动明显增多等[1];另外,当小鼠预先给予氟哌啶醇或氯丙嗪处理后再次注射NMDAR拮抗剂地佐环平则不会引起miR-219水平的改变,说明miR-219可能在NMDAR信号通路中起着重要作用[1]。此外,有文献报道miR-219亦能降低NMDAR诱导的神经元的去极化状态,降低神经元的兴奋性[2]。因此,上述研究结果提示miR-219可在受体和第二信使水平抑制NMDAR介导的信号传导,这一假设与精神分裂症NMDAR功能低下的假说相一致。本关联研究结果也进一步证明了miR-219可能与精神分裂症有关。
另外,miR-219似乎也能从遗传学角度解释精神分裂症和双相情感障碍在多方面存在的共性问题。有研究发现生物钟基因CLOCK/BMAL1能够调控pre-miR-219昼夜节律性表达,抑制视交叉上核miR-219表达则会延长昼夜间期[2]。既往关联研究发现CLOCK基因可能是双相障碍的易感基因[10],该基因敲除后能使小鼠出现类似躁狂的行为表现[11]。因此miR-219可能是精神分裂症以及情绪障碍相关风险通路的共同交叉点,它既可通过影响NMDAR下游信号分子CAMK2G表达导致NMDAR功能低下,继而产生精神病性症状,又可通过抑制CLOCK–BMAL1功能产生情绪症状。故而miR-219可能也与双相障碍有关,可通过关联研究进行初步的验证。
MiRNA是一类不编码蛋白质的小分子RNA,成熟miRNA的生成需要pri-miRNA经过两次的序列切割而成,首先切割形成pre-miRNA,后剪切成成熟miRNA。Han等[11]通过计算机分析,认为一个典型的多细胞动物pri-miRNA二级结构通常由一个含33bp的茎(stem)构成,其末端有一个环(loop)和侧翼片段,茎环状的pre-miRNA结构则包埋于其中。有研究认为在pri-miRNA由Drosha-DGCR8复合物切割其成为pre-miRNA过程中,侧翼片段则对此加工过程至关重要。其长度或二级结构变化很可能会影响pri-miRNA的正常切割过程[12]。因此能够引起该侧翼序列结构变化的突变可能会影响miRNA的表达或稳定性。rs107822位于pre-miR-219-1上游37bp,通过CentroidFold软件预测发现rs107822能够影响pri-miR-219的二级结构。结合本次关联研究结果以及既往miR-219的尸脑表达谱研究[9],推测是否rs107822可通过改变pri-miR-219的二级结构影响成熟miR-219的表达?因此下一步我们将通过体外实验检测rs1027822对miR-219基因表达水平的影响。
目前的研究结果显示CaMKII可通过多种形式与NMDAR的不同亚基相互作用,进而影响NMDAR的功能[13]。因此该激酶对于NMDAR功能的维持是非常重要的。CAMK2G是激酶CAMKII的γ链的编码基因,然而通过SZGene数据库(http://www.szgene.org/)搜索发现,CAMK2G基因并非是精神分裂症相关基因,本关联研究也未发现CAMK2G基因多态位点rs2306327与精神分裂症存在关联。利用Patrocles数据库(http://www.patrocles.org/)发现,rs2306327 恰好位于 miR-384在CAMK2G的3’UTR区上的预测靶结合序列上。近年来研究认为miRNA主要通过与靶基因3’UTR以完全或不完全互补配对的方式在转录后水平调控基因的表达[3]。基因3’UTR区内部相应的能与成熟miRNA相互结合的靶序列区上的多态性变化能破坏miRNA与靶基因的相互作用,继而影响miRNA对靶基因的靶向调控作用。因此rs2306327很可能会破坏miR-384对CAMK2G基因表达的抑制作用,然而,虽然已有数据显示miR-384在大鼠及小鼠的海马、中脑及脑皮层有着丰富表达,但是目前尚缺乏miR-384在人类组织中的表达数据(miRNA表达数据库:http//www.mi crorna.org)。
总之,本研究经关联研究发现miR-219基因可能与精神分裂症有关,该基因上的多rs107822经预测可能会影响pri-miR-219转录本序列的二级结构。
[1]Kocerha J,Faghihi MA,Lopez-Toledano MA,et al.MicroR⁃NA-219 modulates NMDA receptor-mediated neurobehavioral dysfunction[J].Proc NatlAcad SciUSA,2009,106(9):3507-3512.
[2]Cheng HY,Papp JW,Varlamova O,et al.microRNA modula⁃tion of circadian-clock period and entrainment[J].Neuron,2007,54(5):813-829.
[3]Kosik KS.The neuronal microRNA system[J].Nat Rev Neuro⁃sci,2006,7(12):911-920.
[4]Mishra PJ,Bertino JR.MicroRNA polymorphisms:the future of pharmacogenomics,molecular epidemiology and individualized medicine[J].Pharmacogenomics,2009,10(3):399-416.
[5]Shi YY,He L.SHEsis,a powerful software platform for analyses of linkage disequilibrium,haplotype construction,and genetic association at polymorphism loci[J].Cell Res,2005,15(2):97-98.
[6]Sato K,Hamada M,Asai K,et al.CENTROIDFOLD:a web serv⁃er for RNA secondary structure prediction[J].Nucleic Acids Res,2009,37(Web Server issue):W277-280.
[7]闫景星,周玉萍,楚平华精神分裂症的分子遗传学研究进展[J].中国神经精神疾病杂志,2009,35(1):60-64.
[8]Wienholds E,Kloosterman WP,Miska E,et al.MicroRNA ex⁃pression in zebrafish embryonic development[J].Science,2005,309(5732):310-311.
[9]Beveridge NJ,Gardiner E,Carroll AP,et al.Schizophrenia is as⁃sociated with an increase in cortical microRNA biogenesis[J].Mol Psychiatry 2010,15(12):1176-1189.
[10]Benedetti F,Serretti A,Colombo C,et al.Influence of CLOCK gene polymorphism on circadian mood fluctuation and illness re⁃currence in bipolar depression[J].Am J Med Genet B Neuropsy⁃chiatr Genet 2003,123B(1):23-26.
[11]Roybal K,Theobold D,Graham A,et al.Mania-like behavior in⁃duced by disruption of CLOCK[J].Proc Natl Acad Sci USA,2007,104(15):6406-6411.
[12]Han J,Lee Y,Yeom KH,et al.Molecular basis for the recogni⁃tion of primary microRNAs by the Drosha-DGCR8 complex[J].Cell,2006,125(5):887-901.
[13]Colbran RJ.Targeting of calcium/calmodulin-dependent protein kinase II[J].Biochem J,2004,378(Pt1):1-16.

