冰葡萄在自然发酵过程中酵母的动态变化*
赵新节,史涛涛,张汉波,秦邵智,3,张家荣
1(山东轻工业学院 山东省微生物工程重点实验室,山东 济南,250353)
2(云南大学生命科学学院,云南 昆明,650091) 3(德钦梅里酒业,云南 德钦,674500)
葡萄酒发酵是一个复杂的微生物反应过程,包含了不同酵母群体和数量的演替。有研究表明,酵母菌群的差异和组成会明显地影响葡萄酒的感官特征[1-3]。同时,文献中提到,在自然发酵的初级阶段,葡萄表面某些细尖的非酿酒酵母为优势菌种[4-5],随着发酵的进行,乙醇浓度的升高,酿酒酵母主导整个发酵过程[6]。
很多学者采用不同地区以及不同品种的酿酒葡萄进行野生酵母的研究。其中包括Sipiczki 等人分析了匈牙利Tokaj 酒厂环境中自然发酵条件下分离到的酵母类型[7],Sipiczki 等人对匈牙利Aglianico 葡萄自然发酵过程中的野生酿酒酵母进行遗传分离的研究[8],Antonella 等人对意大利的Aglianico,Fiano,
Trebbiano,Malvasiaand,Grechetto,Malvasiadelle Lipari,Inzolia 葡萄自然发酵过程中出现的野生酵母资源进行了相关的鉴定[9-12],MillaAlves 等人对巴西葡萄园中与Isabel,Bordeaux 葡萄相关的野生酵母进行分离和分子鉴定[13],Barbara Brežná 等人评估了捷克斯洛伐克与酿酒有关的酵母菌群的多样性[14],Demuyter 等人发现葡萄汁酵母连续3 年为法国阿尔萨斯酒厂的优势菌种[15],Van Keulen 等人研究了美国伊利湖畔的Chardonnay,Pinot Gris 和Riesling 品种在自然发酵过程中存在的酵母类型[16],澳大利亚的学者Yap 等人报道了致死性非酿酒酵母存在的概率[17],Combina 等人对来自阿根廷3 个不同地区的Malbec 葡萄品种在自然发酵过程中酵母菌株的消长进行了研究[18]。但是,尚未见关于冰葡萄自然发酵过程中酵母动态变化的报道。
本文采用形态学与分子生物技术相结合的方法对冰葡萄自然发酵过程中的酵母进行鉴定,探明了德钦产区与冰酒相关的野生酵母的种群多样性,并为利用本土优良酵母资源提供科学依据。
1 材料与方法
1.1 材料
葡萄品种:赤霞珠;果实样品:冰葡萄;采摘地点:云南省德钦县澜沧江河谷海拔2500 m 左右的冰葡萄基地。
1.2 样品处理
将一定质量的冰葡萄果粒在无菌袋中破碎,加入SO2(30 mg/L),进行自然发酵。葡萄破碎后立即取样(A 取样点),48 h 后第2 次取样(B 取样点)。待发酵启动,根据发酵液中残糖量的变化量取样(具体取样点如表1 所示),再进行梯度稀释。
1.3 菌株的形态划分
取梯度稀释后的样品200 μL 涂布于YEPD 固体培养基平板(加氯霉素100 mg/L)上,28℃富集培养2 ~3 d,挑取单个菌落划线接种到WL 培养基上。根据该培养基上菌落的颜色形态分型,然后用显微镜观察同种类型不同菌株间形态是否存在差异,并计算每种类型菌的数量。

表1 冰葡萄自然发酵过程中还原糖和酒精度变化情况Table 1 The change of sugar and alcohol during spontaneous fermentation associated with ice grape
1.4 菌株ITS,18S rDNA 与26SrDNA 的D1/D2 区序列测定
根据WL 培养基聚类分析结果,随机挑选每种类型的菌株2 株,用试剂盒法(生工生物)提取酵母组DNA。使用引物ITS4 和ITS5 对其ITS 基因进行扩增[19]。对个别ITS 区域差异性较小的菌株进行18S rDNA[20]与26S rDNA 的D1/D2 区域扩增[21]。扩增结束后,以1% 的琼脂糖凝胶进行水平电泳,电压150V,完毕后紫外灯下拍照扩增结果,扩增产物于-20℃保存备用。
1.5 构建系统发育树
测试结果通过DNAstar 和Chromas 软件校对,然后使用NCBI 搜索,找出与之相似度最高的序列,相关序列用MEGA 4 软件经CLUSTALW 比对,采用Jukes-Cantor 模型构建N-J 树。
2 结果与分析
冰葡萄人为破碎后,在室温下进行自然发酵(室温大约为16℃),在发酵过程中选取了10 个样点,共分离到511 株酵母菌株。
2.1 冰葡萄自然发酵过程中酵母菌株的形态学鉴定
WL 琼脂培养基可以用来监测饮料发酵过程中的微生物类群。Cavazza 等[22]研究表明,在葡萄酒自然发酵过程中出现的大多数典型的酵母菌种都可以用WL 培养基进行区分。根据在WL 培养基上酵母的菌落形态,本实验筛选的511 株酵母菌株可分为7类(表2)。通过显微镜观察可知,同种类型不同菌株间形态并不存在差异。通过查询相关文献可知,1,6,7 类酵母分别为Cryptococcus flavescens,Hanseniasporauvarum,Saccharomyces cerevisiae。2,3,4,5 类酵母的菌落形态未见报道。

表2 酵母菌落形态划分结果Table 2 Division results of colony morphologies of yeasts
2.2 冰葡萄自然发酵过程中酵母菌株的分子学鉴定
根据WL 分型结果,从每一类型中选取2 株菌株进行DNA 提取,并对其ITS 区域进行PCR 扩增,具体扩增结果见图1,然后进行分子鉴定。
从图1 中可以看出A2 -1,A2 -2,A3 -1,A3 -2,A4 -1,A4 -2,A7 -1,A7 -2 的PCR 产物长度大约为550bp,A5 - 1,A5 - 2 的PCR 产物长度接近500bp,D1 -1,D1 -2,D 2 -1,D2 -2 的PCR 产物长度大约为750bp。

图1 酵母菌ITS 区基因的PCR 扩增结果Fig.1 Nucleotide fragments length of the 5.8S-ITS profile in the study
通过对酵母菌株PCR 产物测序,DNAstar 和Chromas 软件校对,NCBI 搜索,建立系统发育树,如图2 所示。根据系统发育树及其同源性高低可知A2-1,A2 -2,A4 -1,A4 -2,A7 -1,A7 -2;A5 -1,A5-2;D1 -1,D1 -2;D2 -1,D2 -2 分别为Cryptococcus flavescens,Hyphopichiapseudoburtonii,Hanseniasporauvarum,Saccharomyces cerevisiae。但A3 -1 与A3 -2 在ITS 区域与Cryptococcus amylolentus和Tsuchiyaeawingfieldii的碱基序列完全相同,所以对这2 株菌进行多基因序列分析,即对A3 - 1 与A3 - 2 的18S(SSU),26S(D1/D2)区域同时进行分析,最终得出A3 -1 与A3 -2 为Cryptococcus amylolentus。

图2 5.8SrDNA-ITS 基因构建的系统发育树Fig.2 Phylogenetic tree of 5.8S-ITS gene
2.3 冰葡萄自然发酵过程中酵母菌株的动态变化
对冰葡萄自然发酵过程中的不同阶段进行取样,各个菌种的消长关系如图3 所示。
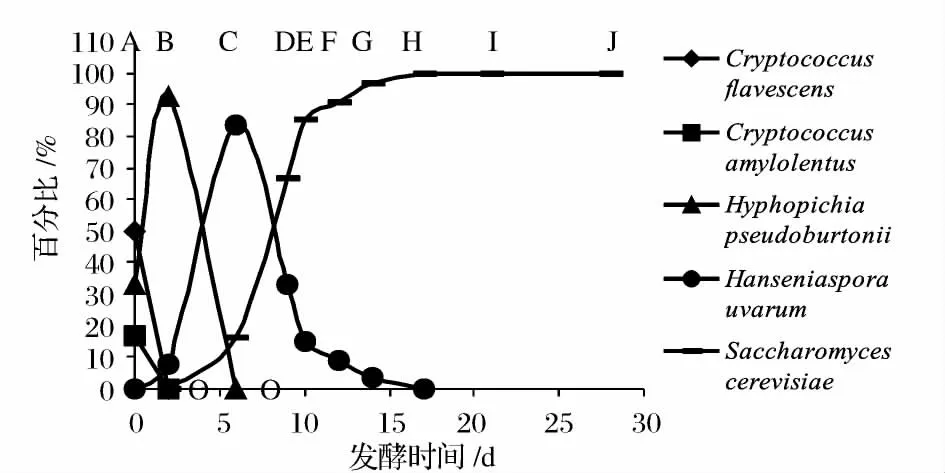
图3 冰葡萄自然发酵过程中酵母菌株的动态变化Fig.3 Thedynamic change of the yeasts during spontaneous fermentation associated with ice grape
在A 取样点,即冰葡萄刚破碎阶段,Cryptococcus flavescens占总酵母数的50%,Cryptococcus amylolentus和Hyphopichiapseudoburtonii各占16.7% 和33.3%。48 h 后,Cryptococcus flavescens,Cryptococcus amylolentus迅速减少甚至消失,Hyphopichiapseudoburtonii成为优势菌种,同时发现Hanseniasporauvarum。当还原糖降为336 g/L,乙醇体积分数为1.9%时,发酵液中只存在Hanseniasporauvarum与Saccharomyces cerevisiae这2 种酵母。随着发酵的进行,乙醇含量升高,Hanseniasporauvarum所占的比例呈动态减少趋势。当还原糖含量为172 g/L,乙醇体积分数上升为11.6%时,发酵液中只分离到Saccharomyces cerevisiae,直至发酵结束。
3 讨论
对于葡萄酒酵母的研究,大量文献探讨的都是正常条件下与成熟浆果有关的酵母菌群[23],未见关于冰葡萄相关酵母的研究。因此,本实验首次对冰葡萄自然发酵过程中酵母的动态变化进行分析。
实验中发现了2 种未在相关报道中出现的酵母——Cryptococcus amylolentus,Hyphopichiapseudoburtonii。其中,Cryptococcus amylolentus最初是由Walt等人从南非的甲虫上分离到[24],之后未见其他分离源。关于这2 种菌的生理特性有待进一步探索。研究表明,在葡萄表皮以及发酵初期存在大量的非酿酒酵母,非酿酒酵母可以产生水解酶,例如D-葡萄糖苷酶,这有利于香气成分的产生和增强葡萄酒的综合特性[25-26]。因此,非酿酒酵母的差异可能是冰葡萄酒有着更为复杂的香气结构和独特口感的原因之一。此外,在整个发酵过程中,Hanseniasporauvarum是非酿酒酵母中在发酵液里存活时间最长的菌种。这可能与Hanseniasporauvarum对酒精有较强的耐性有关。一些学者通过研究发现Hanseniasporauvarum可以通过本身的几种酶来分泌酯和甘油,进而对葡萄酒的香气产生积极的影响[27]。
尽管酿酒酵母在葡萄酒发酵过程中起着重要的作用,但在冰葡萄发酵初期并没有分离到该菌种,然而从发酵中期到结束,酿酒酵母均为优势菌种,这与之前的研究是相符合的[28]。
在成熟葡萄浆果果皮上,附着着天然的酵母菌,其种类、构成比例及菌密度受产地、土壤、气候、年份、葡萄品种、成熟期、葡萄园的管理状况等影响。到目前为止,从葡萄表面已经分离到52 种酵母,它们属于22 个属:Aureobasidium,Auriculibuller,Brettanomyces,
Bulleromyces,Candida,Cryptococcus,Debaryomyces,Hanseniaspora,Issatchenka,Kluyveromyces,Lipomyces,Metschnikowia,Pichia,Rhodosporidium,Rhodotorula,Saccharomyces,Sporidiobolus,Sporobolomyces,Torulaspora,Yarrowia,Zygoascus,和Zygosaccharomyces[29]。在本实验中,我们从冰葡萄发酵过程中只分离到4 种非酿酒酵母:Cryptococcus flavescens,Cryptococcus amylolentus,Hyphopichiapseudoburtonii,Hanseniasporauvarum,这可能是与该产区自然条件以及产区历史有关[30]。
了解葡萄浆果发酵过程中各酵母的动态变化对葡萄酒的酿造具有重要的意义。本文首次对赤霞珠冰葡萄相关酵母资源以及发酵过程中不同菌种的消长关系进行了研究,这对德钦产区冰酒发酵工艺的控制以及优良酵母的筛选和优化具有一定的意义,也为该地区酵母资源的开发利用奠定了基础。
[1] Soles R M,Ough C S,Kunkee R E.Ester concentration differences in wine fermented by various species and strains of yeasts[J]. Am J Enol Vitic,1982,33(2):94 -98.
[2] Lema C,Garcia-Jares C,Orriols I,et al.Contributionof Saccharomyces and non-Saccharomyces populations to theproduction of some components of Albarino wine aroma[J]. Am J Enol Vitic,1996,47(2):206 -216.
[3] Patrizia Romano.Metabolic characteristics of wine strains during spontaneous and inoculated fermentation[J]. Food Technology and Biotechnology,1997,35(4):255 -260.
[4] Masoud W,Cesar L B,Jespersen L,et al. Yeast involved in fermentation ofCoffeaarabica in East Africa determined by genotyping and by direct denaturating gradient gel electrophoresis[J]. Yeast,2004,21(7):549 -556.
[5] Hierro N,González A,Mas A,et al. Diversity and evolution of non-Saccharomyces yeast populations during wine fermentation:effect of grape ripeness and cold maceration[J]. FEMS Yeast Res.,2006,6(1):102 -111.
[6] Fleet G H .Yeast interactions and wine flavor[J]. Int J Food Microbiol,2003,86(1/2):11 -22.
[7] Sipiczki M,Romano P,Lipani G,et al.Analysis of yeasts derived from natural fermentation in a Tokaj winery[J].Antonie van Leeuwenhoek,2001,79(1):97 -105.
[8] Sipiczki M,Romano P,Capece A,et al. Genetic segregation of naturalSaccharomyces cerevisiaestrainsderived from spontaneous fermentation of Aglianico wine[J]. J Appl.Microbiol,2004,96(5):1 169 -1 175.
[9] Calabretti A,Volpe M G,Sorrentino A,et al.Aglianico and Fiano wines obtained with an autochthonousnon-Saccharomyces yeast[J]. Ann Microbiol,2011,61(1):131 -136.
[10] Domizio P,Lencioni L,Ciani M,et al. Spontaneous and inoculated yeast populations dynamics and their effect on organoleptic characters of Vinsanto wine under different process conditions[J]. Int J Food Microbiol,2007,115(3):281 -289.
[11] Agnolucci M,Scarano S,Santoro S,et al.Genetic and phenotypic diversity of autochthonous Saccharomyces spp.strains associated to naturalfermentation of‘Malvasiadelle Lipari’[J]. Lett Appl Microbiol,2007,45(6):657 -662.
[12] CapeceA,Pietrafesa R,Romano P.Experimental approach for target selection of wild wine yeastsfrom spontaneous fermentation of“Inzolia”grapes[J]. World Journal ofMicrobiol Biotechnology,2011,27(12):2 775 -2 783.
[13] MillaAlves Baffi,Carolina dos Santos Bezerra,MaríaArévalo-Villena,et al.Isolation and molecular identification of wine yeasts from aBrazilian vineyard[J].Ann Microbiol,2011,61(1):75 -78.
[14] Barbara Brežná,Katarínaženišová,Katarína Chovanová,et al.Evaluation of fungal and yeast diversity in Slovakianwine-related microbial communities[J]. Antonie van Leeuwenhoek,2010,98(4):519 -529.
[15] Demuyter C,Lollier M,Legras J L,et al. Predominance ofSaccharomycesuvarumduring spontane-ous alcoholic fermentation,for three consecutive years,in an Alsatian winery[J]. J Appl Microbiol,2004,97(6):1 140 -1 148.
[16] Keulen H van,Lindmark D G,Zeman K E,et al. Yeasts present during spontaneous fermentation of Lake Erie Chardonnay,Pinot Gris and Riesling[J]. Antonie van Leeuwenhoek ,2003,83(2):149 -154.
[17] Yap N A,De Barros Lopes M,Langridge P,et al. The incidence of killer activity of non-Saccharomyces yeasts towards indigenous yeast species of grape must:potential application in wine fermentation[J]. J Appl Microbiol,2000,89(3):381 -389.
[18] Combina M,Elia A,Mercado L,et al.Dynamics of indigenous yeast populations during spontaneous fermentation of wines from Mendoza,Argentina[J]. Int J Food Microbiol,2005,99(3):237 -243.
[19] White T J,Bruns T,Lee S,et al. PCR protocols,a guide to methods and applicitions[M]. New York:Academic Press,1990:315 -322.
[20] Spatafora J W,Blackwell M. The phylogenetic origins ofophiostomatoidfungi[J]. Mycological Research,1994,98(1):1 -9.
[21] 谢荣. 甘肃野生酵母菌遗传多样性研究[D]. 兰州:甘肃农业大学,2007:20 -21.
[22] Cavazza A,Grando M S,Zini-Vignevini C. RilevazionedeIlaflora microbicadi mostievini[J].Vignevini,1992,9:17 -20.
[23] Shuangshi Li,Chao Cheng,Zheng Li,et al.Yeast species associated with wine grapes in China[J]. Int J Food Microbiol,2010,138(1/2):85 -90.
[24] J P Van Der Walt,D B Scott,Wilhelmina C Van Der Klift.Six new candida species from south African insect soueces[J]. Mycopatholigia et Mycologia Applicata,1972,47(3):221 -236.
[25] Ubeda J,Briones A. Characterization of differences in theformation of volatiles during fermentation within synthetic andgrape must by wild Saccharomycesstrain[J].Lebensm.-Wiss.u.-Technol.,2000,33(6):408 -414.
[26] ArévaloVillena M,UbedaIranzo J F,Briones Pérez A I.β-Glucosidase activity in wine yeasts:application in enology[J]. Enzyme and Microbial Technology,2007,40(3):420 -425.
[27] Jolly N P,Augustyn O P H,PretoriusI S.The role and use of non-Saccharomyces yeasts in wine production[J].S Afr J Enol Vitic,2006,27(1):15 -39.
[28] Huihui Sun,Huiqin Ma,Meiling Hao,et al.Identification of yeast population dynamics of spontaneous fermentation in Beijing wine region,China[J]. Annals of Microbiology,2009,59(1):69 -76.
[29] Renouf V,Claisse O,Lonvaud-Funel A. Inventory and monitoring of wine microbial consortia[J]. ApplMicrobiolBiotechnol,2007,75(1):149 -164.
[30] Constanti M,Poblet M,Arola L,et al. Analysis of yeast populations during alcoholic fermentation in a newly established winery[J]. Am J Enol Vitic,1997,48(3):339 -344.

