利用COI基因序列鉴别赛加羚羊角与其代用品△
陈静,蒋志刚 *
(1.中国科学院动物研究所 动物生态与保护生物学重点实验室,北京 100101;2.中国科学院大学,北京 100049)
利用COI基因序列鉴别赛加羚羊角与其代用品△
陈静1,2,蒋志刚1,2 *
(1.中国科学院动物研究所 动物生态与保护生物学重点实验室,北京 100101;2.中国科学院大学,北京 100049)
目的:用COI序列作为分子标记鉴定赛加羚羊角与其代用品山羊Caprahircus、绵羊Ovisaries、藏原羚Procaprapicticaudata、蒙原羚P.gutturosa、鹅喉羚Gazellasubgutturosa和藏羚Pantholopshodgsonii的角,为监管羚羊角市场贸易和控制赛加羚羊角走私提供技术支持。方法:从3种不同类型的赛加羚羊角以及藏原羚、蒙原羚、鹅喉羚样品中提取基因组DNA,设计巢式PCR体系,扩增349 bp COI片段,结合从GenBank中下载的序列构建赛加羚羊及其代用品的系统发育树。结果:保存期1~2年的新鲜赛加羚羊角中提取出的基因组DNA浓度比陈旧羚羊角、羚羊角片的高,而陈旧羚羊角不同位置提取出的基因组DNA浓度差别较小。NJ树上,7个物种分别单独聚群,形成独立的分枝,支持率都达到了99%以上。结论:DNA分子鉴定技术结合针对低浓度基因组DNA模板的巢式PCR反应体系客观准确地区分了赛加羚羊角和其易混代用品。本研究不仅为赛加羚羊角的分子鉴定技术建立了操作方法,而且提供了参考序列,简化了实际应用中的鉴定流程,对赛加羚羊贸易监管以及走私监控具有实践参考意义。
羚羊角;贸易;代用品;基因组DNA;COI;分子鉴定
羚羊角是牛科动物赛加羚羊Saigatatarica的角(以下简称羚羊角),因具有清热解毒等功效而被广泛使用。19世纪60年代,分布于我国新疆地区的赛加羚种群因过度捕猎而灭绝[1]。前苏联解体后,分布于其他中亚国家的种群也由于盗猎、疾病等原因在短短几年内下降了90%[2]。1989年,赛加羚羊被列为国家一级重点保护野生动物,1995年,赛加羚羊被列为《濒危野生动植物种国际贸易公约》(CITES)附录Ⅱ物种。在我国民间,山羊Caprahircus、绵羊Ovisaries、藏原羚Procaprapicticaudata、蒙原羚Procapragutturosa、鹅喉羚Gazellasubgutturosa和藏羚Pantholopshodgsonii等的角可当作赛加羚羊角的代用品使用[3]。中药材市场上,羚羊角被制成粉末、丝条和片块等形状销售,加上部分商贩有意造假,羚羊角种类难以识别,给羚羊角贸易监管带来困难,也不利于监控赛加羚羊等濒危物种的盗猎和走私活动。目前已有的羚羊角鉴别方法包括形态比较、显微结构比较[4-5]、蛋白电泳法[6]、薄层色谱法[7]等。实践中使用较多的是依赖经验判断鉴定的形态比较法,相比之下,DNA分子鉴定技术具有成熟的、可重复的实验和统计方法,更为准确、实用。本研究采用COI(cytochrome oxidase subunit 1)基因片段作为分子标记,探索并建立了赛加羚羊角及其易混代用品的分子鉴定方法,以期为羚羊角的市场监管和相似羚羊类物种的保护提供可靠的种类鉴定技术。
1 材料与方法
1.1 材料
中国北京同仁堂(集团)有限责任公司提供了1份赛加羚羊角片以及1个储存了30年以上的陈旧整角;1个1~2年的赛加羚羊新鲜整角由河北省林业厅提供;3个藏原羚样品采集自青海省;2个蒙原羚标本由内蒙古自治区阿日哈沙特口岸提供;3个鹅喉羚样品由新疆维吾尔自治区卡拉麦里自然保护区提供。其他COI基因序列来源于GenBank数据库,见表1。
注:*保护等级:国家重点保护野生动物名录中的保护等级,非名录中物种为空白;**GenBank登录号:从GenBank中下载的序列的登录号,不包括本研究提交的登录号。
1.2 基因组DNA提取
赛加羚羊角以及藏原羚、蒙原羚、鹅喉羚组织样品基因组DNA均采用Universal Genomic DNA Extraction Kit Ver.3.0 (TaKaRa)提取。为验证羚羊角DNA提取效果是否因取样位置而异,分别从陈旧整角角尖、中部、根部以及骨塞处采集样品提取DNA。使用NanoDrop 2000分光光度计 (Thermo Scientific)测定基因组DNA浓度。
1.3 巢式PCR引物设计、PCR扩增和测序
为从低浓度模板DNA中稳定扩增COI序列,本研究根据赛加羚羊线粒体基因组DNA全序列(GenBank登录号JN632700)设计了一套巢式PCR引物。两对引物分别是:
外引物5COI(Forward: 5′-TGAGCCGGCATAG TAGGAAC-3′; Reverse: 5′-CCTGAGTAGTAGGTGAC AATGTG-3′);
内引物SaigaCOI(Forward:5′-GTAGTCGTAA CCGCACAT-3′;Reverse:5′-GTAGGAGGACAGCCGT AAT-3′)。
引物PCR反应体系如下:1×PCR buffer,MgCl22.0 mmol·L-1,4种dNTP各0.2 mmol·L-1,引物各0.5 μmol·L-1,Ex Taq DNA 聚合酶(TaKaRa)0.025 units·μL-1以及2~20 ng·μL-1DNA模板。第一轮PCR反应在20 μL体系下进行,反应条件是:94 ℃ 7 min预变性,94 ℃ 30 s,52 ℃45 s,72 ℃ 1 min,20个循环,最后72 ℃延伸10 min。第二轮PCR反应中,取1 μL第一轮PCR产物作为模板,40 μL反应体系,退火温度为54 ℃,35个循环,其他条件与第一轮PCR相同。扩增产物送到北京诺赛基因组研究中心测序,所获得序列登录到GenBank中,登录号为:KC679005、KC679006、KC679007、KC679011、KC679012、KC679014、KC679019、KC679020、KC679021、KC679028、KC679029。
1.4 数据分析
测序得到的COI序列用BioEdit(Ibis Bioscience)编辑,剪切掉两端的模糊片段。用ClustalX 1.8.1软件进行比对[8]。使用MEGA 4[10]构建基于Kimura 2 parameter[9]模型的Neighbour-Joining 树,系统树各分支的置信度用自举检验法(bootstrap)检验,共进行1 000次循环,以家牛Bostaurus(GenBank登录号AF492351)为外群。
2 结果
2.1 基因组DNA提取
从陈旧整角的不同位置以及经过处理的赛加羚羊角片提取出的DNA浓度相当,并且都远低于1~2年的新鲜赛加羚羊整角,见表2。

表2 赛加羚羊角中提取出的基因组DNA浓度
2.2 COI基因序列比较
研究运用巢式PCR技术从赛加羚羊角以及其易混代用品样品中成功扩增了11条400 bp的COI序列,并从GenBank中获得另外13条序列,经剪切后序列长度为349 bp。全部24条序列进行比对后,其中保守位点有243个,多态性位点有106个,平均核苷酸频率为A(26.2%)、T(31.0%)、C(28.0%),G(14.9%)。
2.3 种间系统发育树
在邻接法构建的系统发育树中,赛加羚羊的4个样品单独聚为一支,支持率为100%。其他6种动物的COI序列也分别单独聚群,形成独立的分枝,支持率都达到了99%以上。在其他6种动物中,藏原羚、蒙原羚、鹅喉羚与赛加羚羊同属于羚羊亚科,藏羚、绵羊、山羊属于羊亚科,亲缘关系较远。COI序列构建的NJ树明显地区分了赛加羚羊与其易混品,并且无论亲缘关系远近都有很好的鉴别效果。见图1。
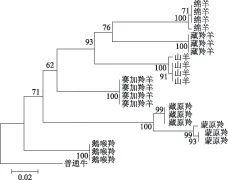
图1 依据349 bp COI序列构建的赛加羚及其易混代用品系统发育NJ树
3 讨论
羚羊角是高度角质化的真皮组织,质地坚韧,细胞含量少,从其中提取基因组DNA比较困难,而市场上存放多年的陈旧羚羊角以及经过加工处理后的羚羊角片、羚羊角粉中的基因组DNA降解严重。因市场上的羚羊角大多是20年以上的库存,本研究分析了陈旧整角不同部位以及新鲜羚羊角的基因组DNA提取效果的差异,以期对羚羊角的市场监管有实际的指导意义。结果发现,取自新鲜赛加羚羊角根部的样品提取的基因组DNA浓度很高,完全可以满足PCR反应需要,而对于陈旧样品,无论样品取自整角的角尖、中部、根部还是骨塞,基因组DNA浓度都很低,无法用常规PCR反应进行扩增。因此,研究中设计了针对于低浓度模板DNA的巢式PCR扩增体系,通过一对外引物进行扩增以富集目的片段,再用第二对内引物进行PCR反应,成功地从低浓度基因组DNA中扩增了349 bp COI序列,并且构建了赛加羚、藏原羚、蒙原羚、鹅喉羚、藏羚、绵羊、山羊的系统发育树,直观、正确地区分了赛加羚羊与其代用品,为利用DNA分子鉴定技术鉴别赛加羚及其代用品构建了实验操作方法,提供了参考序列。
目前国内的赛加羚羊角原料绝大部分依靠库存原料,合法原料供应越来越少,为了获得高额利润,赛加羚羊角走私案件时有发生[11],研究建立的赛加羚羊角DNA分子鉴定技术可以用于赛加羚羊角走私案件的法医鉴定,为打击赛加羚羊偷猎和走私提供了一个有效的技术手段。另外,由于赛加羚羊角与其代用品整角外形相似,刨片或者磨粉之后更加难以区分,部分药材商以次充好、以假掺真[12],严重损害了消费者的利益,本研究建立的利用COI序列鉴定赛加羚羊与其易混代用品的方法也可以为羚羊角市场贸易监测与管理提供可靠的技术支持。CITES公约缔约国大会已将只分布于蒙古的、原来为赛加羚羊一个亚种的北方赛加羚羊亚种S.t.mongolica作为单独的物种,北方赛加羚羊S.borealis列入CITES附录,北方赛加羚与赛加羚这两个物种的区分方法尚需进一步研究。
致谢:中华人民共和国濒危物种科学委员会办公室孟智斌先生、曾岩博士、平晓鸽博士对文章提出宝贵意见并协助采集样品,新疆阿勒泰地区林业局初红军博士提供鹅喉羚样品,河北省林业厅武明录先生以及中国北京同仁堂(集团)有限责任公司提供赛加羚羊角样品,东北师范大学刘炳万博士提供蒙原羚样品,在此谨致谢意!
[1] Jiang Z G, Gao Z X, Sun Q Y. Current status of antelopes in China[J].J Northeast For Univ,1996,7(1): 58-62.
[2] Milner-Gulland E J,Kholodova M V,Bekenov A,et al. Dramatic declines in saiga antelope populations[J].Oryx,2001,35(4): 340-345.
[3] 徐必达,张华林.羚羊角及其代用品的研究进展[J].中药材,2003,26(12):910-914.
[4] 何志刚,郑涛.羚羊角与其人工伪制品的鉴别[J].成都中医药大学学报,2002, 25(4):50-51.
[5] 翟延君,王荣祥,纪俊元.羚羊角及其伪品的生药鉴定研究[J].中国中药杂志,2000,25(6):334-337.
[6] 赵华英,许欣荣,陈永林,等.羚羊角及其伪品的蛋白电泳鉴别[J].中国中药杂志,1994,19(9):524.
[7] 马新.羚羊角与绵羊角,山羊角,牛角及鹿角的薄层色谱鉴别[J].中药新药与临床药理,1994,5(1):47-48.
[8] Thompson J D,Gibson T J,Plewniak F,et al.The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res,1997,25(24): 4876-4882.
[9] Kimura M.A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. J Mol Evol,1980,16(2): 111-120.
[10] Tamura K,Dudley J,Nei M,et al.MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol,2007,24(8): 1596-1599.
[11] Von Meibom S,Vaisman A,Neo Liang S H,et al.Saiga Antelope Trade: Global Trends with a Focus on South-east Asia[R]. TRAFFIC project report to the CITES Secretariat,2010: 56.
[12] Coghlan M L,Haile J,Houston J,et al.Deep Sequencing of Plant and Animal DNA Contained within Traditional Chinese Medicines Reveals Legality Issues and Health Safety Concerns[J]. PLoS genetics,2012,8(4): e1002657.
DiscriminationofSaigaAntelopeHornfromSubstitutesin“Lingyangjiao”MarketsbyGeneticIdentificationTechnology
CHEN Jing1,2,JIANG Zhi-gang1,2*
(1.KeyLaboratoryofAnimalEcologyandConservationBiology,InstituteofZoology,ChineseAcademyofSciences,Beijing100101,China;2.UniversityofChineseAcademyofSciences,Beijing100049,China)
Objective: Employ genetic species identification technology to distinguish Saiga antelope horns from the substitutes in Traditional Chinese Medicine (TCM) markets,including horns of goat (Caprahircus),sheep (Ovisaries),Tibetan gazelle (Procaprapicticaudata),Mongolia gazelle (Procapragutturosa),Goitere d gazelle (Gazellasubgutturosa) and Tibetan antelope (Pantholopshodgsonii). Those substitutes were morphologically similar to Saiga horn,especially when they were processed,for instance,when the horns were grinded into powder,chopped into slides and segments. The final aim of this study was to facilitate the management of TCM markets of “Lingyangjiao” and control Saiga antelope or other endangered Chinese gazelles smuggling.Methods: Genomic DNA was extracted from three Saiga horn samples and samples of Goitered gazelle,Tibetan gazelle and Mongolia gazelle. Two newly designed primer sets were used to amplify COI fragments of 349 bp with nest-PCR systems. Phylogenetic NJ tree of saiga antelope and six substituted species was constructed using the sequences of this study and those retrieved from GenBank.Results: Genomic DNA extracted from different parts of the timeworn Saiga horn,such as the tip,middle part and the base,were low,as well as the processed horn block,whereas concentration of DNA from the new Saiga antelope horn was much higher. In NJ tree,all the seven “Lingyangjiao” species formed independent clades with >99% support values.Conclusion: Combining with nest-PCR systems,molecular diagnosis technology using COI gene as markers proved to be a powerful tool to discriminate Saiga antelope horns from the other “Lingyangjiao” species,especially when dealing with degraded samples from processed or timeworn horns. With the diagnostic protocol and reference sequences provided in this study,identification of biological origins of “Lingyangjiao” sold in TCM could be simplified. The technology might contribute to monitoring the trade of Saiga antelope and controlling Saiga antelope and the other endangered Chinese gazelle smuggling.
Lingyangjiao; TCM; Molecular Diagnosis; COI; Substitutes
2013-01-10)
△
中国科学院知识创新工程重要方向项目——重要CITES附录物种资源评估及致危机理(KSCX2-EW-Z-4);自然科学基金面上基金(31070469);科技基础性工作专项重大项目(2013FY110300)
*
蒋志刚,Tel:(010)64807268,E-mail:jiangzg@ioz.ac.cn

