2,3-丁二醇代谢途径关键酶基因敲除对克雷伯氏菌发酵产1,3-丙二醇的影响
郭欣坤,方慧英 ,诸葛斌,宗红,宋健,诸葛健
1 江南大学 工业生物技术教育部重点实验室,江苏 无锡 214122
2 江南大学 生物工程学院工业微生物研究室,江苏 无锡 214122
3 江南大学 化学与材料工程学院,江苏 无锡 214122
1,3-丙二醇是一种三碳平台化合物,是合成对苯二甲酸丙二醇酯、聚亚氨酯、聚醚、聚胺酯和芳香聚酯的重要单体[1-4]。自然界中能够以甘油为底物生产1,3-丙二醇的菌主要有克雷伯氏菌Klebsialla、弗氏柠檬酸菌Citrobacter、乳杆菌Lactobacilli、梭菌Clostridia[5]等,其中克雷伯氏菌是生产强度和转化率较高的菌种之一,其甘油代谢途径分为还原途径和氧化途径:还原途径生产1,3-丙二醇;氧化途径提供生物生长所需的能量 (ATP)、生物量、还原当量 (NADH),并形成2,3-丁二醇、乳酸、琥珀酸、乙酸、乙醇等副产物。
2,3-丁二醇与 1,3-丙二醇都含有两个羟基,具有高沸点和与水互溶的特点,在发酵液精馏分离过程中,随着2,3-丁二醇在液相中含量的降低,其在汽相中的摩尔分率越来越小,需要采用更大的回流比才能分离提纯1,3-丙二醇,从而造成分离成本增高[6]。此外,2,3-丁二醇的合成需要消耗大量碳源和还原力NADH[7],致使依赖NADH合成的1,3-丙二醇的转化率大大降低,所以降低发酵过程中2,3-丁二醇的产量成为一个需要解决的问题[8-9]。
利用基因敲除技术消除发酵过程中副产物产生是一种提高目的产物产量的有效策略[10-14]。2,3-丁二醇合成途径中有 3种酶:α-乙酰乳酸合成酶 (α-acetolactate synthetase,ALS)、α-乙酰乳酸 脱 羧 酶 (α-acetolactate decarboxylase ,α-ALDC) 和 2,3-丁二醇脱氢酶 (2,3-BD dehydrogenase,BDH)[15-16]。这三个酶的基因位于budABC操纵子上,其中budB编码α-乙酰乳酸合成酶,budA编码α-乙酰乳酸脱羧酶,budC编码2,3-丁二醇脱氢酶。在2,3-丁二醇代谢途径中,α-乙酰乳酸合成酶可以催化丙酮酸形成 α-乙酰乳酸,α-乙酰乳酸在 α-乙酰乳酸脱羧酶作用下形成3-羟基-2-丁酮,2,3-丁二醇脱氢酶催化3-羟基-2-丁酮形成 2,3-丁二醇 (图 1)。本研究利用 Red同源重组技术[17]分别构建了budC和budA基因缺陷株,并通过发酵实验考察这两个基因的缺失对克雷伯氏菌发酵产 1,3-丙二醇的影响。
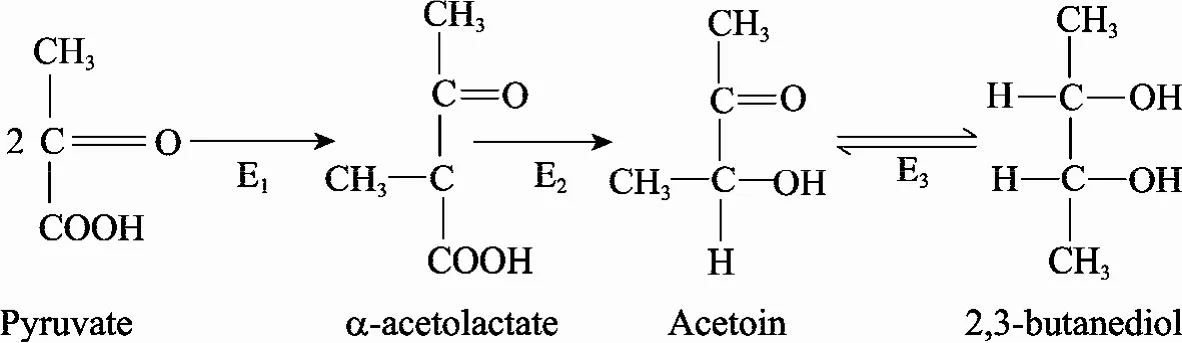
图1 2,3-丁二醇的代谢途径及相关酶[15]Fig. 1 Metabolism pathway of 2,3-butanediol and related enzymes[15]. E1: α-acetolactate synthase; E2: α-acetolactate decarboxylase; E3: 2,3-butanediol dehydrogenase.
1 材料与方法
1.1 菌株与质粒
克雷伯氏菌Klebsiella pneumoniaeZG25由本研究中心筛选并保藏。用于基因敲除所需要的工具质粒pKD13、pKD46和pCP20均购自美国耶鲁大学大肠杆菌菌株库。本研究中使用的引物见表1。
1.2 工具酶及试剂
质粒小量提取试剂盒、细菌基因组DNA抽提试剂盒、DNA凝胶回收试剂盒均购自北京博大泰克生物基因技术有限责任公司;TaqDNA聚合酶、PrimeSTAR HS DNA聚合酶购自宝生物工程 (大连) 有限公司;氨苄青霉素 (Amp)、卡那霉素 (Kan)、氯霉素 (Cm) 购自上海朝瑞生物技术有限公司,2-乙酰氧基-2-甲基-乙酰乙酸乙酯购于Sigma公司;L-阿拉伯糖、monohydrate 2-(N-吗啉代) 乙烷磺酸一水、Brij-35聚环氧乙烯月桂酰醚购自生工生物工程 (上海) 有限公司;其他试剂为国产分析纯。
1.3 培养基与培养方法
种子培养基 (g/L):酵母提取物 5,胰蛋白胨10,NaCl 10。葡萄糖蛋白胨水培养基 (g/L):胰蛋白胨5,葡萄糖5,K2HPO42。发酵培养基(g/L):甘油 40,葡萄糖 5,酵母膏 5,KH2PO47.5,MgSO42,(NH4)2SO42,FeSO4·7H2O 0.005,VB120.015,微量元素溶液10 mL/L,KOH调pH值至8.5。微量元素溶液 (g/L):ZnCl20.07,MnCl2·4H2O 0.1,H3BO30.06,CoCl2·6H2O 0.2,CuCl20.02,NiCl2·6H2O 0.025,Na2MoO4·2H2O 0.035。需要时加入 Amp (100 μg/mL)、Kan(50 μg/mL)、Cm (34 μg/mL)。
种子培养:1%接种量 (V/V)、37 ℃、180 r/min培养6~7 h。发酵培养:250 mL三角瓶、50 mL装液量,4%接种量 (V/V),37 ℃、100 r/min旋转式摇床振荡培养,实验重复3次,取平均值;5 L发酵罐 (韩国BIOTRO公司,型号:BIOG-M),装液量2.5 L,37 ℃,初始甘油浓度20 g/L,转速200~250 r/min,通气量0.5 L/min。从 8 h起流加甘油使其保持在 20 g/L左右,直至 22 h,用10 mol/L的KOH控制pH保持在7.5。

表1 本研究使用的引物Table 1 Primers used in this study
1.4 budC和budA基因的敲除
提取K. pneumoniaeZG25全基因组,根据Klebsiella pneumoniae342 (GenBank Accession No. NC011283) 分别设计budC和budA基因引物CJ1、CJ2和AJ1、AJ2,扩增budC和budA基因并连接 pMD18-T测序。根据测序结果设计引物C1、C2和 A1、A2 (表1),其中下划线部分为目标基因上下游约50 bp的同源臂序列,以pKD13的 DNA为模板,分别扩增目标基因打靶 DNA片段geneD50-Kan-geneD50。将pKD46质粒电转入K. pneumoniaeZG25,接种至含有 Amp(1.4 mg/mL)的 LB液体培养基中,37 ℃培养至8~10 h,然后转移至含有Amp (2.0 mg/mL) 的LB固体平板上12~15 h,筛选出含有pKD46质粒的菌体。将目标基因打靶 DNA片段电转导入K. pneumoniaeZG25 (pKD46),在加入 Kan的 LB固体平板上筛选转化子,升温至42 ℃培养12 h以去除pKD46质粒,将pCP20质粒电转导入菌体细胞,30 ℃培养2 h,利用pCP20消除转化子中Kan抗性基因,然后升温至42 ℃培养12~16 h去除pCP20质粒[18-19]。
1.5 转化子的验证
将打靶DNA片段电转后在Kan抗性平板上长出的转化子用鉴定引物K1、K2;AJ1、AJ2;CJ1、CJ2;KZ1、KZ1’和 KZ2、KZ2’进行 PCR验证。K1和 K2分别是 Kan抗性基因的上游引物和下游引物,CJ1、CJ2和AJ1、AJ2分别是基因bud C和bud A基因上游和下游片段。KZ1、KZ1’和 KZ2、KZ2’分别是 Kan抗性基因中间某段基因序列及其反向互补序列。
1.6 测定方法
1.6.1 生物量及代谢产物测定
发酵液中的菌体密度以分光光度计吸光值OD650表示。细胞干重根据经验公式 1OD=0.25 g/L。发酵液中甘油、1,3-丙二醇、2,3-丁二醇、乙酸、乙醇、乳酸、琥珀酸的测定参考文献[10]。
1.6.2 酶活测定
α-乙酰乳酸脱羧酶的测定参考文献[15],酶活单位定义为37 ℃下每分钟由α-乙酰乳酸脱羧酶脱羧 α-乙酰乳酸产生 1 μmol 3-羟基-2-丁酮的酶量。
2,3-丁二醇脱氢酶的测定参考文献[20],酶活力定义为37 ℃下每分钟转化1 μmol NADH成为NAD+的酶量。
1.6.3 蛋白质含量测定
粗酶液蛋白质含量采用Bradford法[21]测定,以BSA为标准蛋白。
2 结果
2.1 budC和budA单基因缺失菌株的获得
以质粒pKD13 NDA为模板,扩增出目标基因打靶 DNA 片段budCD50-Kan-budCD50和budAD50-Kan-budAD50,转化至含 pKD46质粒的K. pneumoniaeZG25感受态中,同源重组后在Kan平板上初步筛选budC和budA基因缺失突变株。
将含有Kan抗性基因的budC缺失菌株进行菌落 PCR验证,结果如图 2所示。分别利用 4对引物 K1、K2;CJ1、CJ2;CJ1、KZ2’和 KZ2、CJ2对budC缺失菌进行菌落 PCR鉴定,budC基因缺失菌株可以得到 1 303 bp、1 394 bp、627 bp和790 bp大小片段,出发菌株仅CJ1、CJ2引物可扩增得到862 bp片段。含Kan抗性基因的budA缺失菌株的菌落PCR验证如图3所示。分别利用 4对引物 K1、K2;AJ1、AJ2;AJ1、KZ1’和AJ2、KZ1进行菌落PCR鉴定,budA基因缺失菌株可以得到1 303 bp、1 600 bp、752 bp和742 bp大小片段,出发菌株仅AJ1、AJ2可得到1 077 bp片段。DNA凝胶电泳的结果均与理论值相符,表明budC和budA基因敲除成功,将质粒pCP20转化至budC和budA基因缺失菌中,将Kan抗性基因消除,获得的budC基因缺失菌命名为K. pneumoniaeZG38 (budC),获得的budA基因缺失菌命名为K. pneumoniaeZG38(budA)。

图2 budC基因缺失菌的菌落PCR鉴定Fig. 2 Identification of the budC knockout by colony PCR. M: DL2 000 DNA marker; 1, 3, 5, 7: mutant(budC) containing Kan resistance gene; 2, 4, 6, 8:parent strain.
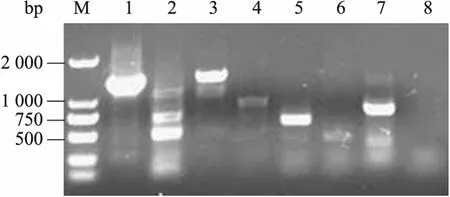
图3 budA基因缺失菌的菌落PCR鉴定Fig. 3 Identification of the budA knockout by colony PCR. M: DL2 000 DNA marker; 1, 3, 5, 7: mutant(budA) containing Kan resistance gene; 2, 4, 6, 8:parent strain.
两株基因突变株的2,3-丁二醇脱氢酶酶活如图4A所示,出发菌株在整个发酵阶段都有酶活性。K. pneumoniaeZG38 (budC)没有检测出2,3-丁二醇脱氢酶酶活,说明K. pneumoniaeZG38(budC)的budC基因已经被成功敲除,同时发现发酵过程中 3-羟基-2-丁酮大量积累 (数据未列出),在发酵5~15 h时发酵液中3-羟基-2-丁酮浓度约为出发菌株的120%,进一步说明因为budC基因的缺失,造成了3-羟基-2-丁酮在发酵液中大量积累。K. pneumoniaeZG3 (budA) 没有检测出 2,3-丁二醇脱氢酶活性,为budA基因的缺失提供了佐证。
两株基因突变株的 α-乙酰乳酸脱羧酶酶活如图4B所示,出发菌株在整个过程中都保持α-乙酰乳酸脱羧酶的活性。K. pneumoniaeZG38(budC) 的budC基因缺失造成3-羟基-2-丁酮积累的同时,α-乙酰乳酸脱羧酶的活性相比出发菌株提高了约20% (6~24 h),K. pneumoniaeZG38(budA) 没有检测出α-乙酰乳酸脱羧酶活性,说明K. pneumoniaeZG38 (budA) 的budA基因已经被敲除。

图 4 K. pneumoniae ZG25、K. pneumoniae ZG38(budC)、K. pneumoniae ZG38 (budA) 2,3-丁二醇脱氢酶 (A) 与 α-乙酰乳酸脱羧酶 (B) 比酶活曲线的测定Fig. 4 Time course of specific activity of BDH (A) and α-ALDC (B) in K. pneumoniae ZG25, K. pneumoniae ZG38 (budC ) and K. pneumoniae ZG38 (budA).
2.2 突变株的分批发酵性能
为了初步考察基因敲除对菌体生长和代谢产物的影响,本研究对出发菌株K. pneumoniaeZG25及构建的基因缺失菌K. pneumoniaeZG38(budC) 和K. pneumoniaeZG38 (budA)进行了摇瓶发酵实验,结果表明,K. pneumoniaeZG38(budC) 和K. pneumoniaeZG38 (budA)的平均比生长速率 (2.07/h,0.583/h)和生长量 (图 5) 均低于出发菌株 (2.22/h),说明budC和budA基因的缺失抑制了菌体的生长,相比之下,budA基因的缺失对菌体的影响更加严重,在整个发酵过程中,K. pneumoniaeZG38 (budA) 的菌体量只有出发菌株的25%~35%。K. pneumoniaeZG38(budA) 的发酵结果显示,菌体不产生 1,3-丙二醇和2,3-丁二醇,而其他副产物的产量明显增加,乳酸、琥珀酸、乙醇和乙酸的产量分别为出发菌株的6.6、2.7、2.1和1.8倍。
甘油测定结果表明 (图 6),K. pneumoniaeZG38 (budC) 利用甘油的能力稍弱于出发菌株,出发菌株 12 h时已经消耗大部分甘油,而K. pneumoniaeZG38 (budC) 到24 h才消耗大部分甘油,至发酵结束时残留甘油含量与出发菌株残留甘油含量都保持在相同的水平 (0.5~1 g/L),而K. pneumoniaeZG38 (budA) 菌株利用甘油的能力降低,直至发酵结束时,发酵液中仍然残留约30 g/L的甘油。

图 5 K. pneumoniae ZG25、K. pneumoniae ZG38(budC) 和 K. pneumoniae ZG38 (budA) 的生长曲线Fig. 5 Time course of cell growth of K. pneumoniae ZG25, K. pneumoniae ZG38 (budC) and K. pneumoniae ZG38 (budA).

图 6 K. pneumoniae ZG25 (A)、K. pneumoniae ZG38(budC) (B )和 K. pneumoniae ZG38 (budA) (C) 的摇瓶发酵Fig. 6 Shake-flask fermentations of K. pneumoniae ZG25 (A), K. pneumoniae ZG38 (budC) (B) and K.pneumoniae ZG38 (budA) (C). 1,3-PDO:1,3-propanediol; 2,3-BD: 2,3-butanediol.
发酵液代谢产物的分析结果如图 6所示,K. pneumoniaeZG38 (budC) 的 1,3-丙二醇和2,3-丁二醇产量分别达到21.7 g/L和3.4 g/L,为出发菌株的111%和65.3%。与出发菌株相比,2,3-丁二醇产量明显下降,此外,K. pneumoniaeZG38 (budC) 单位菌体的 1,3-丙二醇产率、摩尔转化率达到 0.603 g/(L·h)和 0.682 mol/mol,分别为出发菌株的109%和112%。由此可知,budC基因敲除造成的 2,3-丁二醇脱氢酶缺失降低了2,3-丁二醇产量,并提高了1,3-丙二醇产量。
2.3 突变株的分批补料发酵
在摇瓶发酵实验的基础上进一步考察K. pneumoniaeZG38 (budC) 发酵生产 1,3-丙二醇的能力,在 5 L发酵罐中对出发菌株及K.pneumoniaeZG38 (budC) 进行了分批补料发酵实验,发酵结果如表2所示。
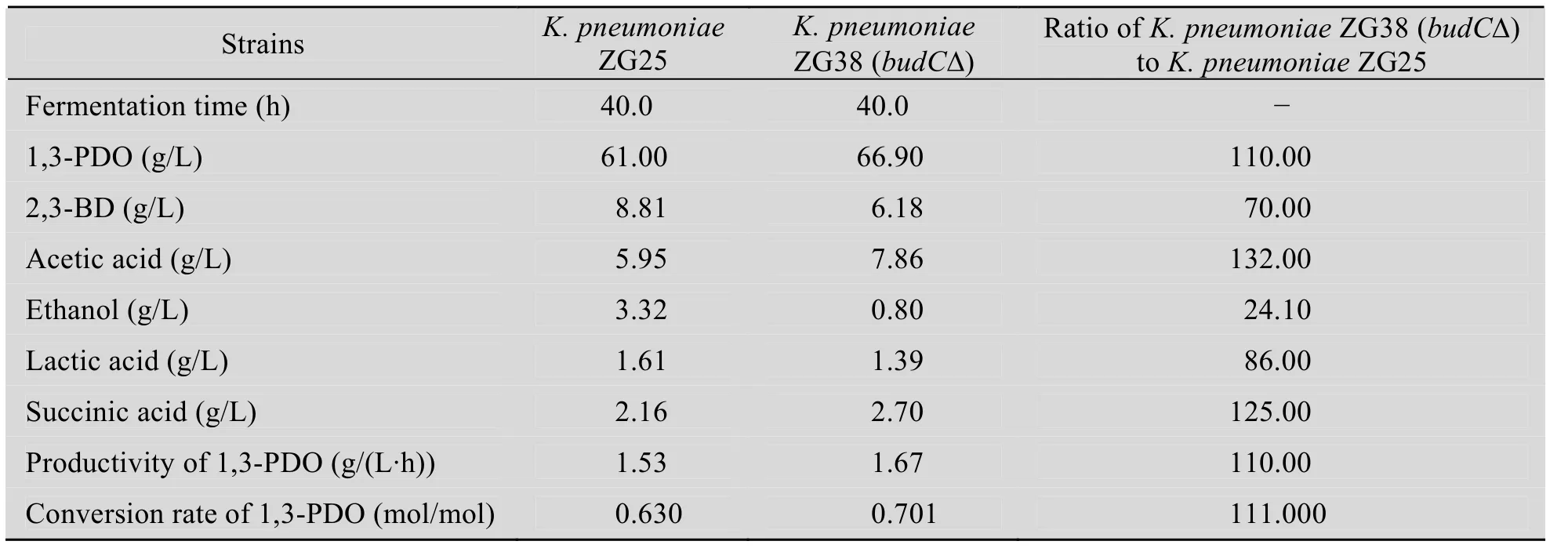
表2 K. pneumoniae ZG25和K. pneumoniae ZG38 (budC) 的分批补料发酵结果Table 2 Fermentation results of fed-batch fermentation of K. pneumoniae ZG25 and K. pneumoniae ZG38(budC)
由表 2可以看出,K. pneumoniaeZG38(budC) 的 1,3-丙二醇浓度为 66.9 g/L,相比于出发菌株提高了10%,1,3-丙二醇产率、对甘油的摩尔转化率也明显增加。正如所预测的,2,3-丁二醇脱氢酶失活导致了2,3-丁二醇和乙醇产量的降低,分别降低了30.0%和75.9%。然而budC基因缺失菌却产生了更多的乙酸、琥珀酸,表明budC基因的缺失可以导致流向 2,3-丁二醇途径的碳流转向乙酸和琥珀酸途径。
3 讨论
菌体代谢旁路途径基因敲除往往会在一定程度上影响菌体的生长,这在许多关于克雷伯氏菌基因敲除研究中已经得到证实[10-11]。本研究中对budC基因和budA基因的敲除也得到了相同的结果。就发酵目的产物来说,budC基因缺失菌株2,3-丁二醇的产量只是比出发菌株降低了 30%,并没有完全按照预测消除2,3-丁二醇的产生,表明克雷伯氏菌可能存在另一条2,3-丁二醇途径,即2,3-丁二醇循环,这条途径已经在枯草芽胞杆菌、蜡状芽胞杆菌和尿素微球菌[22-23]中被发现,并且2,3-丁二醇循环的生理机制还没有阐明,至今尚无关于K. pneumoniae2,3-丁二醇回补途径的研究。
本研究利用基因敲除技术对K. pneumoniae2,3-丁二醇回补途径进行了探索,如图 7所示,当2,3-丁二醇途径因budC基因缺失而被阻断时,通向 2,3-丁二醇途径的碳流通过节点 3-羟基-2-丁酮转向 2,3-丁二醇的回补途径生成 2,3-丁二醇,在这个过程中,乙酸作为回补途径的中间代谢产物被大量形成,理论上1 mol 2,3-丁二醇形成的同时也会形成2 mol乙酸,在分批补料发酵过程中,乙酸的产量约是2,3-丁二醇产量的1.88倍 (表2),与理论预测值基本吻合,从而有力的证实了克雷伯氏菌中可能存在2,3-丁二醇回补途径,并且当主要合成途径被阻断后会通过此回补途径合成2,3-丁二醇。
budA基因的缺失对菌体的生长和生理产生了明显影响,2,3-丁二醇代谢途径在微生物胞内有着重要的生理功能,它维持着细胞内NAD+/NADH平衡,3-羟基-2-丁酮和2,3-丁二醇的转化是一个可逆的生化反应,故2,3-丁二醇代谢途径是细胞的一个还原力库,它的存在可以自动调节胞内的氧化还原平衡,而budA基因的缺失则使其丧失了这一功能,使变异菌株在生长上受到抑制,代谢紊乱,甘油的还原途径被抑制,氧化途径中产生的 NADH消耗在糖酵解到三羧酸循环途径的产物形成过程中,从而导致发酵过程中乳酸、琥珀酸、乙醇和乙酸的产量成倍增加。这与Zhang等[24]报道的产酸克雷伯氏菌budA基因缺失菌株发酵结果相似,Zhang等将budA基因敲除后虽然菌体生长受到抑制,但却提高了1,3-丙二醇的产量,说明 2,3-丁二醇合成途径对1,3-丙二醇产量的影响在不同克雷伯氏菌中的表现是不同的。
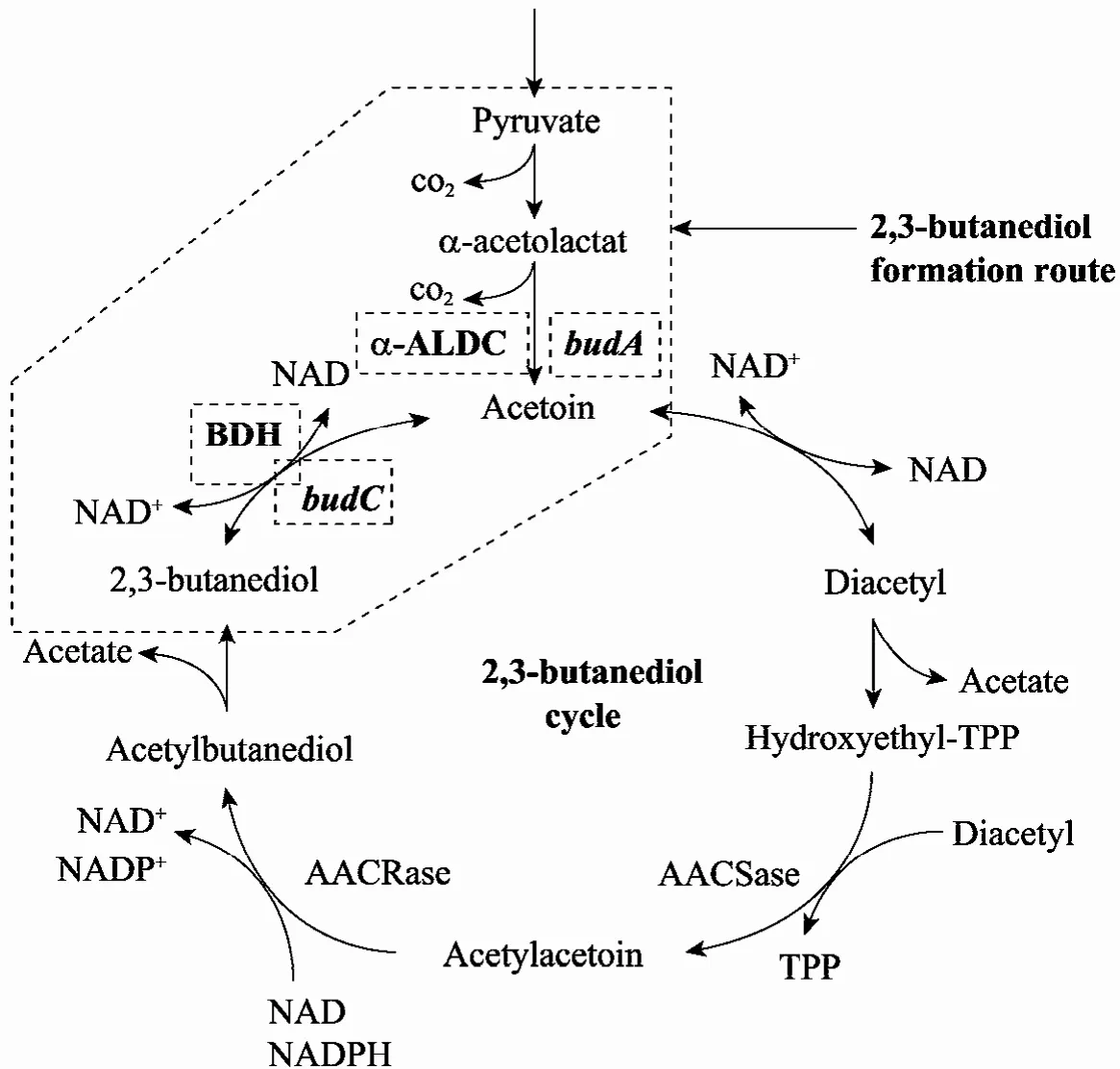
图7 2,3-丁二醇代谢途径和2,3-丁二醇循环[22]Fig. 7 Metabolic pathway of 2,3-BD and the 2,3-BD cycle[22].
根据枯草芽胞杆菌Bacillus subtilis、蜡状芽胞杆菌Bacillus cereus和尿素微球菌Micrococcus urea公布的2,3-丁二醇循环途径[22]推测,若budA基因被敲除则使得α-乙酰乳酸脱羧酶缺失,从而使3-羟基-2-丁酮完全无法合成,导致2,3-丁二醇两条代谢途径都被阻断,最终无2,3-丁二醇产生,这与本研究结果完全一致。基于本研究budC和budA基因敲除和代谢产物分析的结果,可以推断克雷伯氏菌中可能存在2,3-丁二醇回补途径,且2,3-丁二醇循环是以3-羟基-2-丁酮为起点。有关2,3-丁二醇循环的研究结果为以后更加深入的研究提供了基础。在以后的研究中,应该重点研究克雷伯氏菌的2,3-丁二醇循环途径,找出该途径的关键酶基因并尝试敲除关键酶基因,以进一步降低2,3-丁二醇和增加1,3-丙二醇的产量。
[1]Liu HJ, Xu YZ, Zheng ZM, et al. 1,3-propanediol and its copolyments: research, development and industrialization. Biotechnol J, 2010, 5(11):1137−1148.
[2]Kurian JV. A new polymer platform for the future-Sorona from corn derived 1,3-propanediol. J Polym Environ, 2005, 13(2): 159−167.
[3]Kaur G, Srivastava AK, Chand S. Advances in biotechnological production of 1,3-propanediol.Biochem Eng J, 2012, 64: 106−118.
[4]Saxena RK, Anand P, Saran S, et al. Microbial production of 1,3-propanediol: recent developments and emerging opportunities. Biotechnol Adv, 2009,27(6): 895−913.
[5]Biebl H, Menzel K, Zeng AP, et al. Microbial production of 1,3-propanediol. Appl Microbiol Biotechnol, 1999, 52(3): 289−297.
[6]Gao SL, Fang YJ, Qi YW. Prediction of vapor-liquid equilibrium by ASPEN PLUS software for water-2,3-butanediol. Zhejiang Chem Ind, 2007, 138: 25−28 (in Chinese).
高山林, 方云进, 戚一文. 用aspen plus软件预测水-2,3-丁二醇汽液相平衡数据. 浙江化工, 2007,138: 25−28.
[7]Converti A, Perego P, Del Borghi M. Effect of specific oxygen uptake rate on Enterobacter aerogenes energetics: carbon and reduction degree balances in batch cultivations. Biotechnol Bioeng,2003, 82(3): 370−377.
[8]Xiu ZL, Zeng AP. Present state and perspective of downstream processing of biologically produced 1,3-propanediol and 2,3-butanediol. Appl Microbiol Biotechnol,2008, 78(6): 917−926.
[9]Hao J, Xu F, Liu H, et al. Downstream processing of 1,3-propanediol fermentation broth. J Chem Technol Biotechnol, 2006, 81(1): 102−108.
[10]Zhang YP, Li Y, Du CY, et al. Inactivation of aldehyde dehydrogenase: A key factor for engineering 1,3-propanediol production byKlebsiella pneumoniae.Metab Eng, 2006, 8(6):578−586.
[11]Yang G, Tian JS, Li JL. Fermentation of 1,3-propanediol by a lactate deficient mutant ofKlebsiella oxytocaunder microaerobic conditions.Appl Microbiol Biotechnol,2007, 73(5):1017−1024.
[12]Xu YZ, Guo NN, Zheng ZM, et al. Metabolism in 1,3-propanediol fed-batch fermentation by a D-lactate deficient mutant ofKlebsiella pneumoniae.Biotechnol Bioeng, 2009, 104(5):965−972.
[13]Seo MY, Seo JW, Heo SY, et al. Elimination of by-product formation during production of 1,3-propanediol inKlebsiella pneumoniaeby inactivation of glycerol oxidative pathway. Appl Microbiol Biotechnol,2009, 84(3): 527−534.
[14]Guo NN, Zheng Z M, Mai YL, et al. Consequences of cps mutation ofKlebsiella pneumoniaeon 1,3-propanediol fermentation. Appl Microbiol Biotechnol, 2010, 86(2): 701−707.
[15]Blomqvist K, Nikkola M, Lehtovaara P, et al.Characterization of the genes of the 2,3-butanediol operons fromKlebsiellaterrigenaandEnterobacter aerogenes. J Bacteriol, 1993, 175(5):1392−1404.
[16]Mayer D, Schlensog V, Bock A. Identification of the transcriptional activator controlling the butanediol fermentation pathway inKlebsiella terrigena. J Bacteriol, 1995, 177(18): 5261−5269.
[17]Wei D, Wang M, Shi JP, et al. Red recombinase assisted gene replacement inKlebsiella pneumoniae. J Ind Microbiol Biotechnol, 2012,39(8): 1219−1226.
[18]Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes inEscherichia coliK-12 using PCR products. PNAS, 2000, 97(12): 6640−6645.
[19]Baba T, Ara T, Hasegawa M, et al. Construction ofEscherichia coliK-12 in-frame, single-gene knockout mutants: the Keio collection. Mol Syst Biol, 2006, 2(1): 1−11.
[20]Wardwell SA, Yang YT, Chang HY, et al.Expression of theKlebsiella pneumoniaeCG21 acetoin reductase gene inClostridium acetobutylicumATCC 824. J Ind Microbiol Biot,2001, 27(4): 220−227.
[21]Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Ana Biochem, 1976, 72(1): 248−254.
[22]Juni E, Heym GA. A cyclic pathway for the bacterial dissimilation of 2,3-butanediol,acetylmethylcarbinol, and diacetyl I. General aspects of the 2,3-butanediol cycle. J Bacteriol,1956, 71(4): 425−432.
[23]Ui S, Hosaka T, Mizutani K, et al. Acetylacetoin Synthase as a Marker Enzyme for Detecting the 2,3-Butanediol Cycle. J Biosci Bioeng, 2002,93(2): 248−251.
[24]Zhang G, Yang G, Wang X, et al. Influence of blocking of 2,3-butanediol pathway on glycerol metabolism for 1,3-propanediol production byKlebsiella oxytoca. Appl Biochem Biotechnol,2012, 168(1): 116−128.

