80株中国大陆新生隐球菌环境株表型和分子特征的初步研究
林丽娜,谈 燕,朱定衡,赵 瑾,陈江汉
在中国大陆引起人类疾病的隐球菌主要是新生隐球菌,它有2 个血清型,5 个基因型。在自然界中绝大部分新生隐球菌存在于鸽粪中,主要侵犯中枢神经系统,因此鸟粪和人体中的隐球菌之间的关系一直为人们所关注。对此,国外已有关于这方面的研究[1,2],国内的研究大都集中于临床株。本研究拟对中国大陆新生隐球菌环境株的基因型和分子特征进行初步分析,以期进一步了解环境株与临床株之间的内在关系。
1 材料与方法
1.1 标本
所有标本采集于2011 年2 月~2012 年2 月黑龙江省、北京市、上海市、江苏省、浙江省、四川省、广西壮族自治区等地区私人和公共养鸽场所(如人寓所、公棚及广场等),不限季节,共计1 372 份。主要利用各地信鸽协会集鸽时采集鸽粪,以痰瓶封闭保存并标记,送至实验室分批分离;鸽粪外观以不干不湿为佳,每次每份约采集2 g。
1.2 方法
1.2.1 鸽粪中新生隐球菌的分离 培养基采用咖啡酸玉米琼脂培养基(caffeic acid cornmeal agar,CACA),试验证实此方法可以较好地分离出新生隐球菌环境株[3]。其制备方法如下:取40 g 玉米粉加入到1 L 蒸馏水中,80 ℃水浴20 min 后用尼龙布过滤,补充蒸馏水至1 L 后加入300 mg 咖啡酸,125 mg 氯霉素和20 g 琼脂。100 kPa 高压灭菌15 min 后制成CACA 平板(直径100 mm)。将采集的标本分置于无菌瓶中,取1.0 g 加入10 ml 无菌生理盐水,充分震荡混匀,溶解后静止15 min,使之沉淀。吸取0.5 ml 上清液接种于CACA 培养基上,30 ℃孵育2 周,每3 d 观察1 次。若发现培养基上有棕褐色菌落生长,即将其挑取接种于YEPD 培养基(1%酵母抽提物,2%蛋白胨,2%葡萄糖)上进行分离纯化。印度墨汁涂片染色,在显微镜下见厚壁、圆形带有清晰荚膜的孢子,即可初步确定为隐球菌。
1.2.2 尿素酶试验 尿素琼脂培养基的配制:①将蛋白胨 1 g,葡萄糖 1 g,NaCl 5 g,KH2PO42 g,尿素 20 g 加入到100 ml 蒸馏水中,彻底混合溶解,再加入酚红 0.012 g,在室温25 ℃下调整pH 6.0±0.1。②将上述配制好的溶液过滤消毒。③溶解15 g 琼脂到900 ml 蒸馏水中煮沸及高压灭菌消毒(121 ℃,15 min)。④等待冷却至50℃~55 ℃,无菌操作加入②中液体。⑤彻底混合后分装于平皿以备用。以标准菌株新生隐球菌H99 作为阳性对照,白念珠菌ATCC76615∶09 作为阴性对照,将分离纯化后的待检菌接种于尿素琼脂培养基,于30 ℃培养1 d,观察培养基颜色变化,培养基呈粉红或桃红色为阳性,说明该菌能产生尿素酶,否则为阴性。
1.2.3 37 ℃生长试验 以标准菌株新生隐球菌H99作为阳性对照,白念珠菌ATCC76615∶09 作为阴性对照,将分离纯化后的待检菌接种于YEPD 培养基,于37 ℃培养3 d,观察菌落生长情况。培养基见菌落生长,则为阳性,否则为阴性。
1.2.4 M13-PCR 将上述环境菌株进行PCR 指纹分析,结合核糖体基因内转录间隔区( internal transcribedspacer, ITS)序列及系统进化分析。图1 中的3 株菌[Hljf4(黑龙江)、Sh2(上海)、SC6(四川)]分别代表中国大陆不同地区的环境株,并与有地域代表性的临床株JMY(辽宁)、YGY(浙江)、WSS(江苏)和5 株标准株VN Ⅰ(WM148) 、VN Ⅱ(WM626)、VN Ⅲ(WM628) 、VN Ⅳ(WM629) 、VNB (BT63)作对照。①隐球菌基因组DNA 的抽提按照Wang 等[5]氯化苄法操作。②M13-PCR 指纹分型按照Meyer 等[6]反应体系及反应条件操作。③ITS 基因扩增及测序按照Katsu 等[7]凝胶电泳检查分析系统比较临床菌株与标准株扩增条带的异同,不比较相应条带亮度的强弱,仅比较条带的有无,进行分型归类。
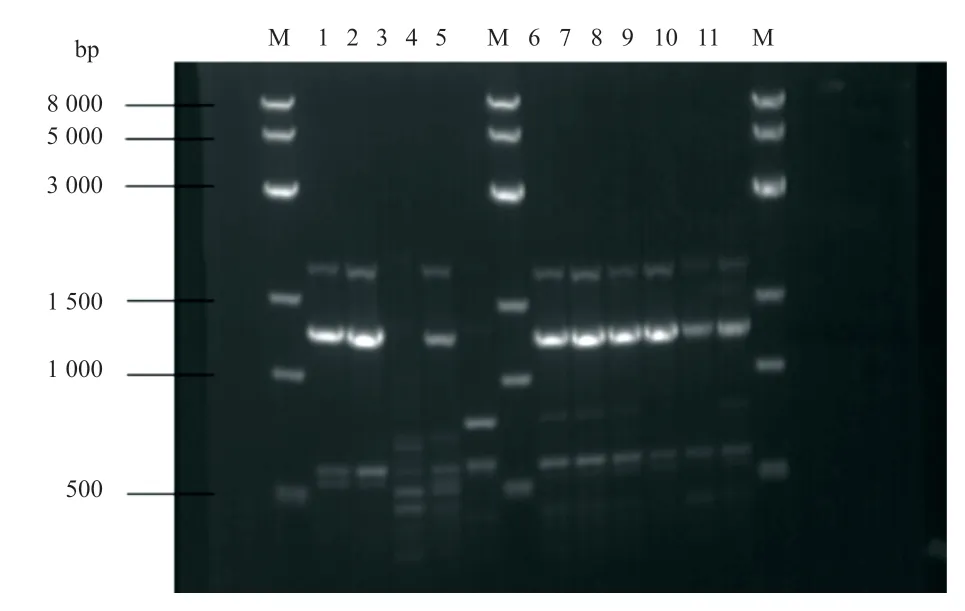
图1 部分新生隐球菌环境株及临床株、标准株M13-PCR指纹图谱凝胶电泳图
1.2.5 动物毒力试验 以10 只9 周大的BALB/c 雌性小鼠,选取环境株Hljf4、Sh2、SC6,临床株JMY、YGY、WSS,标准株H99、VN Ⅰ(WM148)、VN Ⅱ(WM626),由尾静脉注射1×108菌数/株[4],共观察2 个月,1 周1 次,观察动物的存活率。
2 结果
2.1 新生隐球菌分离结果
集鸽鸽粪分离新生隐球菌结果见表1,从分离结果可见集鸽鸽粪分离率大于公共广场。
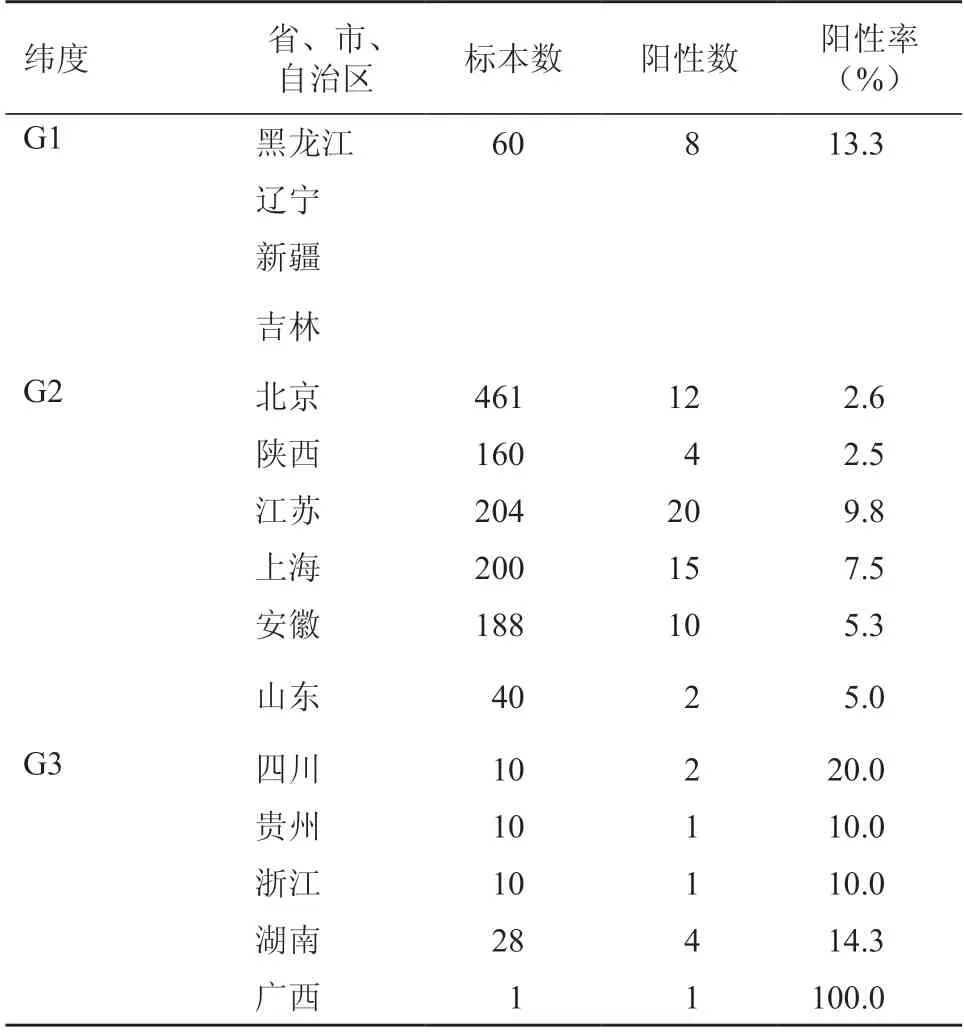
表1 不同地域集鸽鸽粪分离新生隐球菌菌株比较(株)
2.2 尿素酶试验
所有受试菌与阳性对照菌在尿素培养基上培养40 min ~1 d,培养平皿均出现桃红色色素,阴性对照管始终未见变色,未发现尿素酶阴性菌株。
2.3 37 ℃生长试验
所有受试菌与阳性对照菌在YEPD 培养基上培养3 d 后均出现菌落生长,阴性对照组未见生长,未发现37 ℃生长阴性菌株。
2.4 M13-PCR
环境株与3 株临床株以及标准株的M13-PCR 指纹图谱显示,它们均为VNⅠc 基因型,显示了与临床株的高度一致性。ITS 序列分析表明,它们均属于血清型A 。标准株及临床株、环境株M13-PCR 指纹图谱见图1。ITS 序列分析结果已上传至美国国家生物技术信息中心(NCBI)Genebank(JMY NCBI Genbank 序列号:GQ850150)。
2.5 动物毒力试验
动物毒力试验见图2。3 株感染新生隐球菌环境株Hljf4、Sh2、SC6 的小鼠分别与感染新生隐球菌临床株JMY、YGY、WSS 的小鼠出现相近的存活率,并与感染VNⅠ、VNⅡ的存活率相近,且均未死亡,而感染H99 的小鼠在第20 d 死亡。提示新生隐球菌环境株及临床株的毒力相近,且毒力均小于H99。

图2 动物毒力试验
3 讨论
研究新生隐球菌环境株与临床株关系的意义在于揭示其感染人体的自然过程,为临床上有效预防和治疗该病提供可能的方法和途径。中国大陆的临床株为独特的VNⅠc 型,且表现出明显的一致性[8],并与周边国家如韩国、越南等相似。这些均提示它们可能有共同的来源,但未得到证实。M13-PCR 显示,中国大陆不同区域的新生隐球菌环境株也都是VNⅠc 型,且一致性明显。因此证实,我国大陆地区的临床株可能均来自于自然界中的鸽粪。由于新生隐球菌较易发生于免疫缺陷患者,因此对这些易感人群的防控将会大大降低疾病的发生率。
本次调查显示鸽粪中新生隐球菌的阳性分离率平均约为5.5%,低于国内外文献报道[9-14],这可能与采集方法、标本新鲜程度、阳光、温度、气候和分离方法等有关。
调查结果显示,来源于集鸽鸽粪的分离率高于公共广场,可能的原因是,利用各地集鸽时间采集的鸽粪基本来自每户私人养鸽场所,其通风不良,鸽棚地面和笼子中堆积的鸽粪也较厚,这些条件可能为新生隐球菌在鸽粪中生长繁殖提供了更为适宜的条件。另外,分离率还与地域相关,按照北纬20oN ~50oN划分的3 个区域,鸽群的标本分离率以20oN ~30oN最高(G3,15.25%),其次为40oN ~50oN (G1,5.45%),30oN ~40oN (G2,5.1%)最低。分析其原因,可能除了与饲养条件、鸽子的活动习性、运动迁徙等有关外,还与气候条件紧密相关,提示相应的自然环境(温带)更适合该真菌的生存。这些原因与致病力及环境pH值的关系有待进一步研究。
[1] Hwang SM. Molecular characterization of clinical and environmental strains of Cryptococcus neoformans isolated from Busan, Korea [J]. J Bacteriol Virol, 2010, 40(2):91-98.
[2] Mseddi F, Sellami A, Jarboui MA, et al. First environmental isolations of Cryptococcus neoformans and Cryptococcus gattii in Tunisia and review of published studies on environmental isolations in Africa [J]. Mycopathologia, 2011, 171(5):355-360.
[3] 李安生, 吕桂霞, 沈永年, 等. 对两种新生隐球菌选择性培养基的比较研究 [J]. 微生物学通报, 2001, 28(5):69-73.
[4] Varma A, Wu S, Guo N, et al. Identification of a novel gene, URE2, that functionally complements a urease-negative clinical strain of Cryptococcus neoformans [J]. Microbiology, 2006, 152(Pt 12):3723-3731.
[5] Wang S, Miao X, Zhao W, et al. Geneticdiversity and population structure among strains of the entomopathogenic fungus, Beauveria bassiana, as revealedbyinter-simplesequencerepeats (ISSR) [J]. Mycol Res, 2005, 109(Pt 12):1364-1372.
[6] Meyer W, Marszewska K, Amirmostofian M, et al. Molecular typing of global isolates of Cryptococcus neoformans var. neoformans by polymerase chain reaction fingerprinting and randomly amplified polymorphic DNA-a pilot study to standardize techniques on which to base a detailed epidemiological survey [J]. Electrophoresis, 1999, 0(8):1790-1799.
[7] Katsu M, Kidd S, Ando A, et al. The internal transcribed spacersand5.8SrRNA gene show extensive diversityamongisolates of the Cryptococcus neoformans speciescomplex [J]. FEMS Yeast Res, 2004, 4(4-5):377-388.
[8] Chen J, Varma A, Diaz MR, et al. Cryptococcus neoformans strains and infection in apparently immunocompetent patients, China [J]. Emerg Infect Dis, 2008, 14(5):755-762.
[10] Castanón-Olivares LR, López-Martínez R. Isolation of Cryptococcus neoformans from pigeon(Columbalivia)droppings in Mexico City [J]. Mycoses, 1994, 37(9-10):325-327.
[11] Criseo G, Gallo M. Serotyping of Cryptococcus neoformans isolation from environmental and clinical sources in extreme southern Italy [J]. Mycoses, 1997,40(3-4):95-100.
[12] Refai M, Taha M, Selim SA, et al. Isolation of Cryptococcus neoformans, Candida albicans and other yeasts from pigeon droppings in Egypt [J]. Sabourandia, 1983, 21(2):163-165.
[13] Bottone EJ, Kirschner PA, Salkin IF. Isolation of highly encapsulated Cryptococcus neoformans serotype B From a patient in New York city [J]. J Clin Microbiol, 1986, 23(1):186-188.
[14] Romeo O, Scordino F, Chillemi V, et al. Cryptococcus neoformans/Cryptococcus gattii species complex in southern Italy: an overview on the environmental diffusion of serotypes, genotypes and mating-types [J]. Mycopathologia, 2012, 174(4):283-291.

