药用植物长春花WRKY转录因子的鉴定及表达谱分析
杨致荣,王兴春,薛金爱,孟令芝,李润植
1 山西农业大学分子农业与生物能源研究所,山西太谷 030801
2 山西农业大学文理学院,山西太谷 030801
3 山西农业大学生命科学学院,山西太谷 030801
WRKY 是植物特有的一个转录因子超家族,它们都含有1个或2个独特的WRKY DNA 结合域。WRKY 域是一段由60个左右氨基酸残基组成的多肽,因其N 端有高度保守的WRKYGQK核心序列而得名[1]。WRKY 域的C 末端是一个典型的锌指结构,即C2H2(氨基酸的组成模式为C-X4-5-C-X22-23-H-X1-H)或C2HC (氨基酸的组成模式为C-X7-C-X23-H-X1-C)[2]。WRKY 域能特异地与靶基因启动子 W-box 序列[2],或 SURE(Sugar-responsive cis-element)[3]结合,从而调控靶基因的表达。根据其结构域的数目及锌指结构的类型可将WRKY 转录因子分为3大类群:第1类含有2个WRKY 结构域,锌指结构类型为C2H2型;第2和第3大类群WRKY 转录因子只含有1个WRKY 结构域,锌指结构分别为C2H2和C2HC型。其中第2大类群WRKY 可进一步分为a、b、c、d 和e 等5个亚类[1]。
自从第1个WRKY基因SPF1从白薯Ipomoea batatas 中克隆后[4],许多物种的WRKY 基因相继被克隆和鉴定,包括拟南芥 Arabidopsis thaliana[5-6]、水稻Oryza sativa[7]、大豆Glycine max[8-9]、松树Pinus monticola[10]、苜蓿Medicago truncatula[11]等20多种高等植物,和绿藻Pinus monticola[12-13]及两种非植物的真核生物肠兰伯式鞭毛虫 Giardia lamblia 和盘基网柄菌Dictyostelium discoideum[13]。长春花Cantharanthus roseus 既是一种优良的园艺观赏花卉,又是一种重要的药用植物。因其植株体内含有抗癌活性的长春碱(Vinblastine)等70多种萜类吲哚生物碱(Terpenoid indole alkaloids,TIAs)而被广泛应用于抗癌药物的提取和开发,是目前研究TIAs 次生代谢的主要模式植物[14]。我们对长春花TIAs次生代谢物合成途径的一些主要酶蛋白基因的启动子分析发现,这些基因的启动子序列含有W-box 顺式元件,暗示长春花CrWRKY 转录因子可能参与TIAs 合成途径的调控(杨致荣等,未发表数据)。然而,有关CrWRKY 转录因子的种类、数量、结构和功能还未见详尽报道。为此,我们从长春花26009个蛋白中筛选和鉴定出47个CrWRKY 转录因子,并用数字基因表达谱和实时荧光定量PCR (Real time quantitative PCR,qPCR)数据对CrWRKY 转录因子的分类和表达谱进行了系统研究,深化了对CrWRKY 转录因子家族的成员、蛋白结构、基因序列及其进化的认识。本研究为进一步全面解析长春花WRKY的功能和萜类吲哚生物碱的合成调控网络奠定了基础,有利于建立优化的代谢工程策略以实现抗癌长春碱等目标次生代谢物大规模生物合成和利用。
1 材料与方法
1.1 序列的收集与整理
拟南芥和长春花的蛋白序列分别从拟南芥生物资源中心(The Arabidopsis Information Center,ABRC,http://www.arabidopsis.org/)及药用基因组资源数据库(Medicinal Plant Genomics Resource,MPGR,http://medicinalplant genomics.msu.edu/)下载。首先,将下载的序列拷贝到Notepad++软件中进行编辑,若某一蛋白有不同序列,选择最长的氨基酸序列;然后,将这些编辑好的蛋白序列拷贝到Excel 文件中,建成用于后续分析的蛋白序列数据库。
1.2 长春花CrWRKY 转录因子的筛选和鉴定
从上述编辑好的单拷贝蛋白序列数据库中检索含有WRKY 的序列,并统计WRKYGQK 和WRKYGKK 以及这两个基序变异序列WRKYGEK 和WRKYGSK 的数目,从而初步确定 WRKY 转录因子的数目。为保证获得的WRKY 转录因子信息的准确性,将已鉴定含有WRKY 基序的蛋白序列在NCBI 保守域数据库(NCBI Conserved Domain Database,http://www.ncbi.nlm.nih.gov/cdd/)中进行保守域分析,进一步鉴定具有WRKY 功能域的蛋白序列。最后,再利用 PlantTFcat (http://plantgrn.noble.org/PlantTFcat/)转录因子在线分析工具分析这些具有WRKY 功能域的蛋白序列,进一步验证其是否属于转录因子。
1.3 长春花及拟南芥WRKY 序列的聚类分析及比对
参照Eulgem 等[2]定义的WRKY 结构域,以上述鉴定的长春花及拟南芥WRKY 核心序列N端第10个氨基酸残基开始的65个氨基酸残基为靶标序列,运用MEGA 5.0软件[15]采用邻位相连法(Neighbor-joining)进行聚类分析并构建系统进化树。靴值(Bootstrap)设定为3000个重复,进化距离采用p 距离法及每个位点不同氨基酸的数目进行计算。
运用CLUSTAL W2.1软件[16],将长春花CrWRKY 序列的WRKY 域分别与拟南芥相应类群WRKY 结构域进行比较分析,进而确定长春花CrWRKY 的分类。将包括有N 末端WRKY结构域(N-terminal WRKY domains,NTWD)和C 末端 WRKY 结构域(C-terminal WRKY domains,CTWD)的WRKY 蛋白分别标注N 末端及C 末端结构域进行分析,以鉴定N 和C 末端WRKY 结构域的特点。
1.4 长春花CrWRKY 基序的组成分析
WRKY 转录因子的WRKY 核心序列及不同类锌指结构基序用MEME 4.8.0在线基序分析程序(http://meme.nbcr.net/meme/intro.html)分析其保守性。具体参数选择如下:选择基序位点数量最小2次以上,最大不超过50;选择单一基序重复次数为任何;每个基序的宽度为6~100个氨基酸残基。
1.5 CrWRKY 的表达分析
长春花基于转录组测序的表达数据从MPGR 数据库下载。对47个已鉴定的WRKY 不同组织、幼苗、原生质体(Protoplast)、毛状根和色氨酸脱羧酶干扰(Trptophan decarboxylase interference,TDCi)及色胺卤化酶(RebH/F)转基因根系利用MeJA 或YE 处理的表达数据进行系统聚类及其表达谱分析。分析选用GenePattern(http://genepattern.broadinstitute.org/gp/pages/inde x.jsf)在线分析工具,参数设置为选用皮尔森相关系数(Pearson correlation coefficient)及类平均(Average-linkage)法,无需定心(Centering)、标准化(Normalization)及对数转换(log transformation)处理,蓝色、红色和黑色分别表示WRKY 基因表达下调、上调和不受调节,颜色的深浅表示相对表达量的高低。
1.6 长春花毛状根和原生质体培养以及MeJA处理
长春花毛状根的培养按照杨致荣等[17]描述的方法进行。选用生长良好、重量相同的无性系毛状根为样品用100μmol/L MeJA 处理,处理方法采用Suttipanta 等的方法[18]。长春花原生质体分离和悬浮培养依据Pattanaik 等[19]描述的方法进行。毛状根分别在处理后0、12和24 h 取样,原生质体分别在处理后0、6、12和24 h 取样,随即置于液氮中,以备后续提取RNA 之用。
1.7 总RNA 的提取和qPCR
100 mg 上述材料在液氮中研磨成粉末,用于后续RNA 提取。用DNase I 处理RNA 样品以除去可能的DNA 残余。cDNA 合成采用Invitrogen公司SuperScriptⅡ反转录体系。实时定量PCR应用SYBR GreenⅠ试剂盒。每个96孔PCR 板上同一RNA 样品重复3次,每处理进行3次生物学重复。20µL 反应体系含9µL cDNA 模板,10µL SYBR Green Supermix 和基因特异引物各0.5µL (终浓度230 nmol/L)。PCR 反应程序为:95℃30 s;95℃5 s ,60℃20 s,40个循环。选用长春花40S 核糖体蛋白S9基因(RPS9基因)作为内参对照。以2-DDCt 方法计算各基因的相对表达量。实验所用各CrWRKY 引物以及内参基因引物见表1。

表1 定量PCR 所用引物Table 1 Primers for qPCR
2 结果与分析
2.1 长春花CrWRKY 转录因子的筛选和鉴定
为鉴定长春花CrWRKY 转录因子,我们首先从MPGR 长春花蛋白数据库中检索到26009个单拷贝蛋白序列,再从中筛选出62个含有WRKY 基序的蛋白。进一步的分析表明,这62个蛋白中有45个含有WRKYGQK 核心序列,1个含有 WRKYGKK 核心序列,1个含有WRKYGRK 核心序列(表2)。这47个可认定为候选WRKY 家族转录因子,其余15个含有WRKY 基序的蛋白C 末端序列严重缺失,不具有典型WRKY 转录因子的特征。随后的NCBI CDD 和PlantTFcat 分析进一步证实这47个蛋白均包含WRKY 结构域,属于WRKY 转录因子家族蛋白(杨致荣等,未发表数据)。
为验证所获得的47个长春花CrWRKY 序列是否完整,将其全长氨基酸序列与拟南芥72个WRKY 转录因子进行比对。结果表明47个CrWRKY 中序列最长的为731个氨基酸残基(Cra_locus_9152),最短的只有105个氨基酸残基(Cra_locus_6519)。40个CrWRKY 序列含有1个WRKY 域,其余7个包含2个WRKY 域(表2)。4个序列因C 末端序列不全,缺失C 末端的锌指结构域(Cra_locus_2271,Cra_locus_56567、Cra_locus_19330和Cra_locus_1702),1个序列仅含不完全的锌指结构域(Cra_locus_6519)(图1)。
2.2 长春花CrWRKY 转录因子的序列比对和分类

图1 CrWRKY 和代表性AtWRKY 结构域比对分析Fig.1 Alignment of multiple CrWRKY and selected AtWRKY domain sequences.Alignment was performed with Clustal W.The suffix “N” or “C” indicates the N- or C-terminal WRKY domains of a specific WRKY protein.The amino acids forming the zinc-finger motif are highlighted in yellow.The conserved WRKY amino acid signature is highlighted in grey,and gaps are marked with dashes.For Cra_locus_1702 and 18989,upper lines show WRKY motifs and partial zinc-finger motifs using 65 amino acid sequences of the WRKY domains for alignment.Whereas,lower lines show WRKY motifs and typical zinc fingers using the first 25 AA sequences of the 65 AA sequences and the downstream 39 AA sequence beginning at the 53rd AA of the 65 AA sequence in Cra_locus_1702 and at the 65th AA in Cra_locus_18989,respectively,for alignment.
以WRKY 域65个氨基酸残基为靶序列,将47个CrWRKY 转录因子和拟南芥已知的72个AtWRKY 转录因子进行聚类分析。含有2个WRKY 域的蛋白序列分别标注 CTWD 及NTWD,并且分别作为独立的WRKY 蛋白来运算。结果表明,已鉴定的47个CrWRKY 转录因子可分为3大类(图2)。第1类群(Group 1,G1)包括11个CrWRKY 蛋白。其中7个蛋白含有2个WRKY 域,它们的NTWD 和CTWD 分别与拟南芥第1类群的N 末端群(Group 1-N,G1-N)和C 末端群(Group1-C,G1-C)聚类在一起。其余4个含一个WRKY 域的蛋白,分别与拟南芥的G1-C 类群(Cra_locus_10348,65433和43671)和G1-N 类群(Cra_locus_2271)聚类在一起。第2类群(Group 2,G2)包括31个CrWRKY 蛋白,分别与拟南芥的G2-a、G2-b、G2-c、G2-d 和G2-e 5个亚类群聚类在一起。第3类群(Group 3,G3)包括5个CrWRKY 蛋白,全部与拟南芥的G3-a 亚类聚类在一起。值得注意的是,与拟南芥WRKY 不同,目前已鉴定的长春花WRKY 蛋白中还未发现G-3b 亚类成员(图2)。
为验证上述CrWRKY 蛋白聚类分析的准确性,从上述各类群中随机选取一个拟南芥AtWRKY 蛋白和相应类群的CrWRKY 蛋白核心域的65个氨基酸残基进行序列比对。结果表明同一类群中的CrWRKY 除了WRKY 基序及锌指结构相同外,整个WRKY 结构域中的氨基酸残基序列存在高度相似性,而且与相应群体的AtWRKY 具有高度同源性(图1)。这说明上述CrWRKY 的分类结果是正确的。
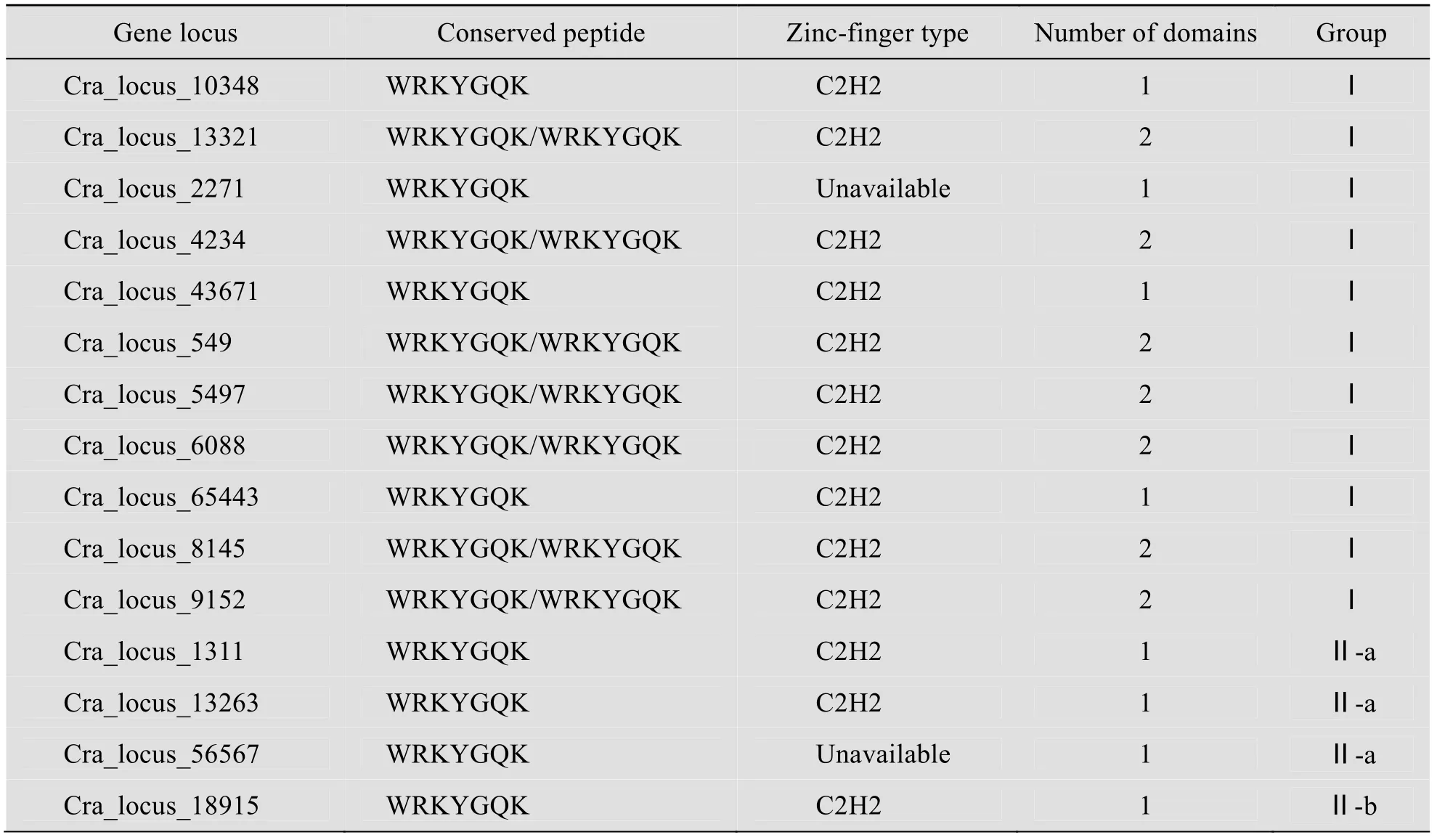
表2 长春花中WRKY 转录因子家族Table 2 WRKY transcription factor families in C.roseus
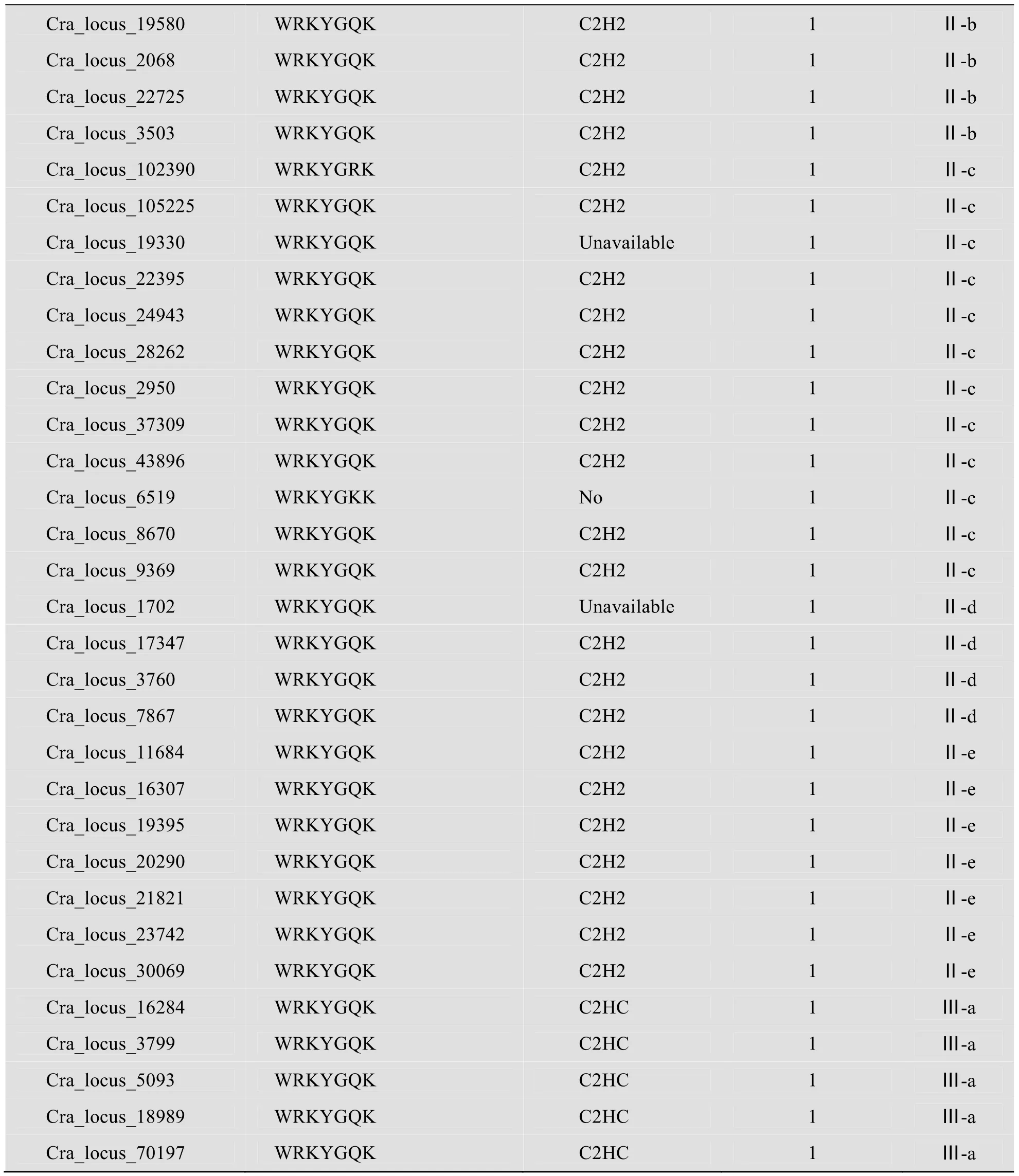
续表2
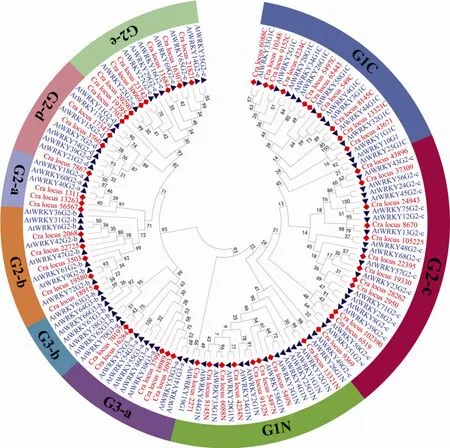
图2 长春花和拟南芥WRKY 结构域进化关系系统树Fig.2 Phylogenetic tree representing relationship among WRKY domains of C.roeus and Arabidopsis thaliana.The amino acid sequences of the WRKY domain of all CrWRKY and AtWRKY proteins were aligned with ClustalW and the phylogenetic tree was constructed using the neighbor-joining method in MEGA 5.0.Group 1 proteins with the suffix“N” or “C” indicates the N-terminal WRKY domains or the C-terminal WRKY domains.The leaf labels of WRKY members from C.roeus (Cra_locus_Nos) and Arabidopsis (AtWRKYs) are denoted in red and blue respectively.The WRKYs with similar structure clustered together are indicated by filled circle.
2.3 CrWRKY 家族WRKY 基序和锌指结构的保守性与多样性
将CrWRKY 结构域中包括WRKY 基序的25个氨基酸残基及锌指结构的35个氨基酸残基分别运用MEME 4.8.0在线软件分析,以检测CrWRKY 转录因子家族中WRKY 基序和锌指结构的保守性与多样性。如图3所示,WRKYGQK基序在CrWRKY 不同成员中是高度保守的,仅在G2-c 中发现一些变异序列(WRKYGRK 和WRKYGKK)。与WRKY 基序高度保守性不同,锌指结构在不同类群CrWRKY 中差异较大。第1和第2类群成员的锌指结构为C2H2型,其中G1-N 锌指结构的序列为 C-X4-C-X22-H-X1-H(图4A),G1-C 和 G2-c 锌指结构序列为C-X4-C-X23-H-X1-H (图4B),而G2-a、G2-b、G2-d和G2-e 的序列均为C-X5-C-X23-H-X1-H (图4C)。第3类群成员的锌指结构为C2HC 型,序列为C-X7-C-X23-H-X1-C (表2,图4D)。需指出的是,Cra_locus_2271、56567、19330和17024个CrWRKY 蛋白现有序列缺少C 末端序列,因而未检出典型的锌指结构。需进一步对其DNA 和RNA 进行测序来分析这4个CrWRKY 蛋白的完整序列。Cra_locus_6519蛋白序列仅含有典型锌指结构的部分基序(缺少C 氨基酸残基)(图1)。

图347 个CrWRKY 蛋白的WRKY 基序Fig.3 Conserved WRKY motifs in 47 CrWRKY proteins.The CrWRKY sequence used for alignment was 25 AA in length containing WRKY motifs.The bit score indicates the frequency of the amino acids at each position in the sequences.
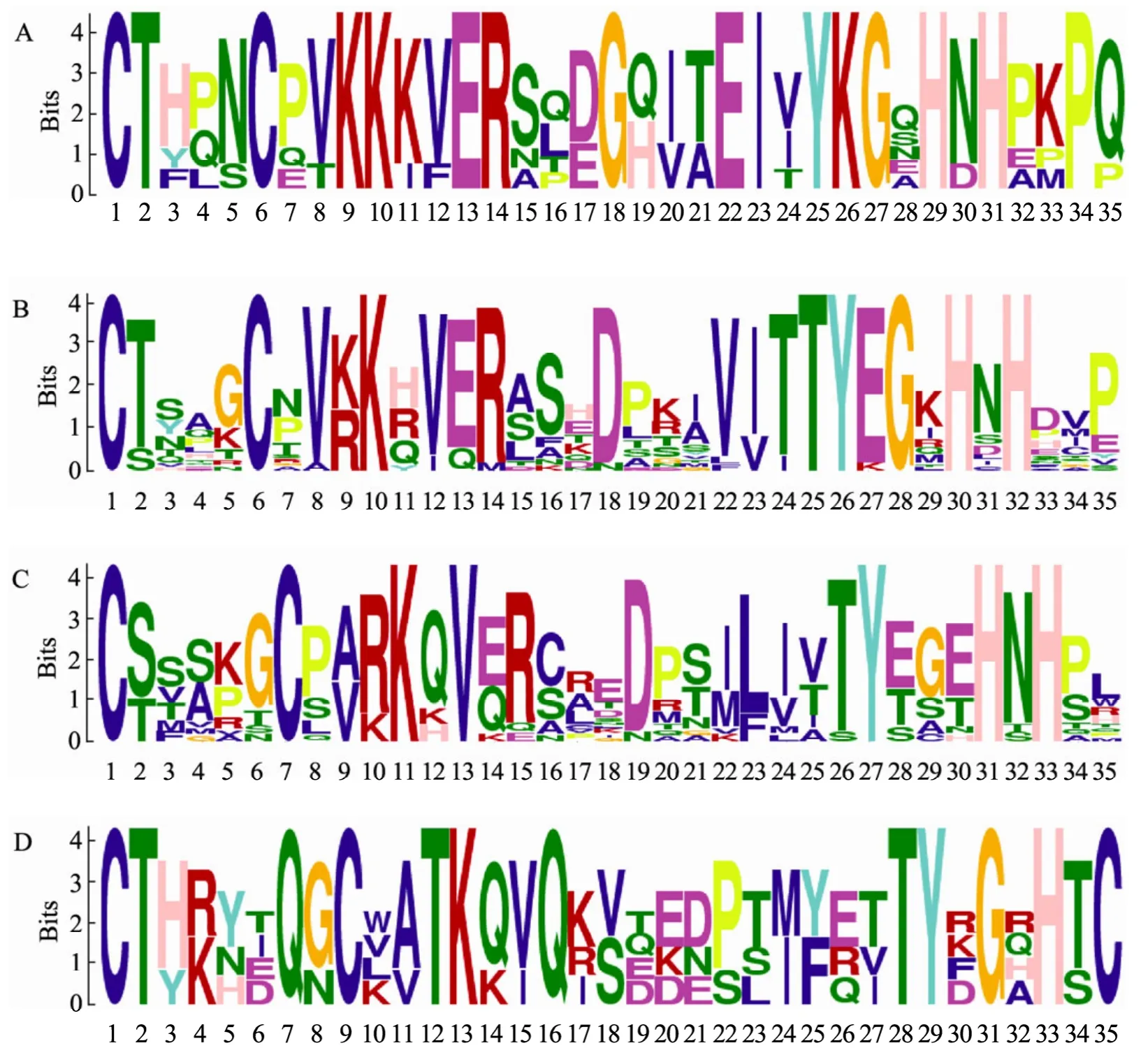
图447 个CrWRKY 蛋白锌指结构的保守性和多样性Fig.4 Conservation and diversity of the zinc-finger motifs in 47 CrWRKY proteins.The CrWRKY sequence used for alignment was 35 AA in length containing zinc-finger motifs.The bit score indicates the frequency of the amino acids at each position in the sequences.
2.4 CrWRKY 的表达谱
基因表达模式的分析是鉴定基因功能的重要手段。为深入了解各CrWRKY 转录因子的生物学功能,我们从MPGR 数据库下载了47个CrWRKY 基因已有的表达数据。这些数据包括CrWRKY 在长春花根、毛状根、茎、叶和花等不同器官,TDCi 及RebH/F 转基因毛状根系,以及MeJA 和YE 处理的幼苗、毛状根和原生质体中的表达数据。系统聚类和表达谱分析表明,CrWRKY 基因的表达具有器官特异性(图5)。在根中高表达的有9个,其中3个显著高表达(Cra_locus_6519、2271和3799)。20个CrWRKY在茎中高表达,其中10个(Cra_locus_6519、17347和4234等)显著高表达,4个(Cra_locus_1311、3760、20290和2950)为茎特异高表达。叶片中高表达的有7个,其中3个(Cra_locus_19359、70197和4234)为显著高表达,仅Cra_locus_70197为叶片特异高表达。花器官有4个显著高表达的 CrWRKY,其中Cra_locus_8145、549和18915为花特异表达。仅Cra_locus_4234在根、茎和叶片中均表达。
毛状根是长春花等一些药用植物合成积累萜类吲哚生物碱等次生代谢的重要器官。对CrWRKY 基因在长春花野生型和两种转基因毛状根(TDCi 和RebH)中表达数据分析表明,超过半数的 CrWRKY 成员在毛状根中高表达(图5)。如野生型毛状根中高表达的CrWRKY 有23个,TDCi 的转基因毛状根中26个CrWRKY高表达,RebH 转基因毛状根在MeJA 处理后高表达的CrWRKY 达29个。在这3种毛状根中均高表达的CrWRKY 有11个。MeJA 处理引起野生型毛状根中CrWRKY 上升表达有11个,减低表达的有12个。TDCi 毛状根经MeJA 处理后,上升表达的CrWRKY 为6个,下降表达的有11个。可见MeJA 处理即可上调也可下调CrWRKY在毛状根中的表达,而且下调表达的多于上调表达的。在原生质体中高表达的CrWRKY 有8个,MeJA 处理导致CrWRKY 上升表达有10个,下降表达的有3个。YE 处理原生质体则诱导11个CrWRKY 上升表达,仅2个下降表达。与MeJA处理毛状根对CrWRKY 表达影响不同,MeJA 和YE 处理原生质体诱导CrWRKY 上升表达的数目明显高于下降表达的。
表达谱系统聚类分析将47个CrWRKY 表达谱划分为3种不同表达模式(图5):第1组(Group 1) CrWRKY 主要在花和MeJA 或YE 处理的悬浮培养的原生质体中高表达,如Cra_locus_13321、22725和10348等;第2组(Group 2) CrWRKY 蛋白主要在茎、毛状根(野生型毛根、转基因TDCi 毛根和转基因RebH/F毛根)和受MeJA 处理的毛状根中高表达(如Cra_locus_1311、6088、11684、16307、21821和2950等),其中少数在原生质体中高表达且受MeJA 诱导(如Cra_locus_5497、19330和22395等);第3组(Group 3)主要在根、茎、叶、幼苗和毛状根及受MeJA 处理的毛状根中高表达,但在原生质体中低表达且不受MeJA 诱导(如Cra_locus_6519、17347和7867等)。
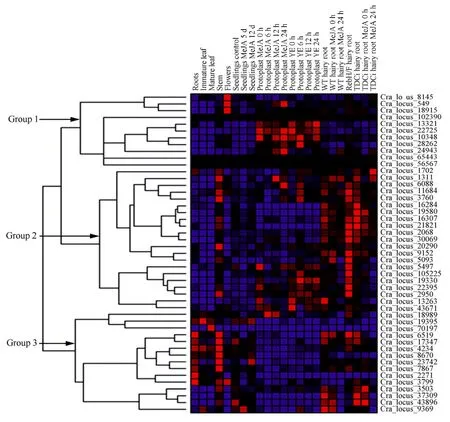
图5 长春花中的47个CrWRKY 基因在不同组织器官和不同处理条件下的表达谱聚类分析Fig.5 Hierarchical clustering and expression profiles of the 47 CrWRKYs in different organs/tissues and under various treatments in C.roseus.Blocks with colors indicate low/down expression (blue),high/up expression (red) or no expression/no change (black).WT,wild-type;RebH,flavin-dependent halogenase;TDCi:tryptophan decarboxylase gene silenced by RNAi.
进一步的分析表明,部分蛋白序列聚类在一起的CrWRKY 成员,其表达模式也类似,但不完全吻合。 G2-c 亚类 CrWRKY 的Cra_locus_105225、22395和19330的表达谱极相似,同划在第2组表达模式。G2-c 的Cra_locus_43896和37309表达谱相近,划在第3组表达模式。 G2-e 亚类 CrWRKY 的Cra_locus_21821和16307表达谱亦相似,划在第2组表达模式。表达谱相似的 G1大类CrWRKY 的Cra_locus_549和8145划在第1组表达模式。但同在G1大类群的Cra_locus_13321、9152和4234的表达模式分别划分在第1、2和3组。
2.5 CrWRKY 表达模式的验证
为验证上述基于数据库已有表达数据系统分析所揭示的CrWRKY 转录因子表达模式,我们先选取5个在不同器官表达差异较大的CrWRKY 转录因子,应用实时定量PCR 检测它们的表达情况。其中 Cra_locus_2271、Cra_locus_20290、 Cra_locus_70197和Cra_locus_8145分别在根、茎、叶和花器官显著高表达。定量 PCR 检测结果表明,这4个CrWRKY 在所检测器官中表达量高低与上文数字基因表达谱表达分析结果基本一致(图6A)。数字基因表达谱和定量 PCR 数据均显示Cra_locus_1702在根和茎中表达量相似,尽管表达量不是很高,但显著高于在叶片和花中的表达(图5和6A)。
上述数字基因表达谱分析还显示,许多CrWRKY 基因的表达受MeJA 和YE 的调控。为验证这些CrWRKY 转录因子表达模式,我们用MeJA 分别处理长春花原生质体和无性系毛状根。原生质体分别在处理后0、6、12和24 h 取样。无性系毛状根在处理后0、12和24 h 取样。提取总RNA,用qPCR 检测所选取的CrWRKY基因的表达情况。用qPCR 检测MeJA 处理原生质体样品中 Cra_locus_13321、24943、1311、13263、18989和8670等6个CrWRKY 基因表达的结果表明,Cra_locus_13321、24943、1311、13263、18989五个基因受MeJA 诱导,它们的表达模式基本与数字基因表达谱所揭示一致,其表达峰值和时态变化有较大差异。而数字基因表达谱数据分析表明Cra_locus_8670不受MeJA 的影响,qPCR 结果表明该基因受MeJA 诱导,呈上升表达,但上升幅度很小(图6B)。依据数字基因表达谱数据,从毛状根受MeJA 诱导表达发生变化的CrWRKY 基因中,分别选取3个上升表达(Cra_locus_24943、11684和30069)和减低表达(Cra_locus_9152、6519和19580) WRKY 基因作为检测靶标。如图6C 所示,与数字基因表达谱分析结果不同,MeJA 处理毛状根并未引起Cra_locus_30069上升表达和Cra_locus_19580下降表达,而是导致2个CrWRKY 呈现相反表达模式。其余4个CrWRKY 基因受MeJA 影响,在毛状根中表达量发生显著变化,表达模式与数字基因表达谱分析所揭示的基本一致。
3 讨论
WRKY 转录因子家族含有众多成员,其蛋白结构和生物学功能具有多样性。鉴定WRKY 转录因子种类及数量是深入研究其生物学功能的基础。本研究从长春花的26009个蛋白中筛选鉴定出47个CrWRKY 转录因子(表2,图2),约占蛋白总数的0.18%。而在拟南芥(72个)、水稻(109个)和玉米(136个)基因组中这一比例分别为0.26%、0.41%和0.42%。无论从WRKY的总数还是所占比例来说,长春花都远远少于上述3种植物。这可能是在进化过程中拟南芥、水稻和玉米基因组中WRKY 基因发生了更多的基因复制事件,导致WRKY 基因的快速扩张[12,20],而在长春花中CrWRKY 基因单拷贝居多。但由于长春花全基因组测序还未完成,因此不排除一些CrWRKY 基因还未被发现的可能。
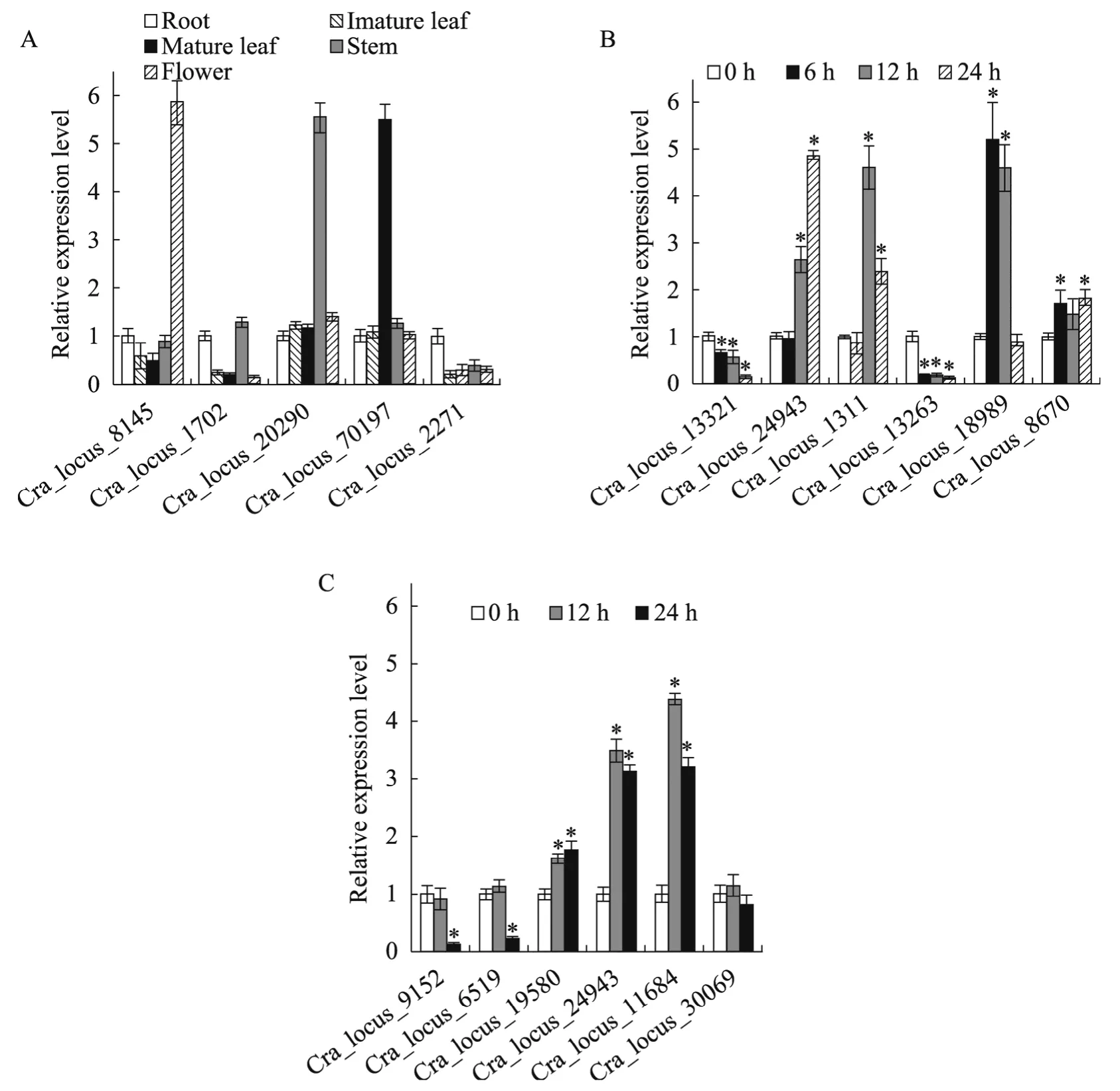
图6 代表性CrWRKY 基因表达模式的qPCR 验证Fig.6 Expression verification of the selected CrWRKY genes revealed by qPCR.(A) Expression patterns of five selected CrWRKY genes in various organs.(B) Expression patterns of six selected CrWRKY genes in MeJA-treated protoplasts.(C) Expression patterns of six selected CrWRKY genes in MeJA-treated hair roots.For real-time PCR,the relative expression of mRNA (y-axis) was calculated according to the description in materials and methods.The RPS9 gene was used as an internal control to normalize the data.Error bars are standard deviations of three biological replicates.Stars above the bars indicate statistically significant increases or decreases compared with the un-treated control (P<0.05,t test).
依据蛋白结构,高等植物WRKY 转录因子家族一般分为3个大类和8个亚类[1,11],本研究通过与拟南芥AtWRKY 蛋白序列比较,以及系统聚类和WRKY 域基序保守性等分析,将47个CrWRKY 也分为相似的类群,即G1、G2(G2-a、b、c、d 和e)和G3(G3-a),但无G3-b 成员(图1、图2及表2)。依其蛋白序列分析,推测Cra_locus_10348等3个CrWRKY 可能起源于具有2个WRKY 域的家族成员,在进化过程中丢失1个WRKY 域。G1类群成员WRKY 域丢失的现象也在拟南芥和玉米等基因组中发生。如拟南芥G1成员AtWRKY10只包括1个WRKY域[20],玉米G1类群中16个成员仅含有1个WRKY 域[21]。有关WRKY 家族进化研究认为,编码1个WRKY 域的基因是从编码2个WRKY域的基因进化而来,且C 末端WRKY 域相当保守,在决定DNA 结合过程中起主导作用[12]。换句话说,N 末端WRKY 域在进化过程中常发生丢失。这也可解释,已检测WRKY 基因的高等植物中,具有1个WRKY 域的WRKY 蛋白数量远大于具有2个WRKY 域蛋白数量。
拟南芥和水稻等植物的G3类群WRKY不仅数目多(拟南芥11个,占20%和水稻36个,占30%),而且基因扩增明显。例如,水稻G3-a 和G3-b 分别有10和26个OsWRKY 成员[12],且WRKY 域保守序列变异大于G1和G2类群的OsWRKY。G3类群的OsWRKY 扩增与基因复制相关。Zhang 等[13]报道,在单、双子叶植物发生歧化(约160 Mya 前)后,G3类群WRKY 开始独立复制。Ling 等[20]进一步证实,拟南芥、水稻和大豆的G3类群WRKY的扩增是进化上近期(24~40 Mya)基因组复制的结果。与之不同的是,长春花CrWRKY G3类群仅有5个成员,全聚类为G3-a 亚类。显然,长春花G3类群WRKY无扩增现象,这与黄瓜基因组中G3类群WRKY(6个成员)情形类似[20]。类似的,裸子植物松树Pinus monticola 中编码83个PmWRKY 蛋白,但亦未检出G3类群成员[10]。可见,G3类群WRKY 在植物进化过程中变异较大,暗示了在不同种属植物中该类群成员的功能有较高的歧化性。
此外,对47个CrWRKY 蛋白的WRKY 域保守基序的分析显示,WRKYGQK 是WRKY 基因最保守的基序,仅在G2-c 中发现一些变异序列(如WRKYGRK 和WRKYGKK)(图1)。然而,锌指结构基序在序列长短和结构上差异较大。与其他类群CrWRKY 蛋白C2H2型锌指结构不同,G3类群WRKY 锌指结构为C2HC 型。这反映了不同类型的WRKY 基因的所经历选择压力和进化模式是不同的。
WRKY 转录因子在植物抵御多种生物胁迫和逆境反应中起着重要的作用[22-24]。本研究也发现,约1/3 CrWRKY 基因的表达受MeJA 和YE调控。大量研究表明,YE 能诱导多种参与植物抵御病原菌侵袭的基因表达,而MeJA 直接参与植物对生物胁迫和非生物胁迫的防卫反应。众多CrWRKY 的表达受MeJA 和YE 调控,预示着这些CrWRKY 可能参与长春花植株抵御病原菌等生物胁迫以及非生物胁迫反应的调控网络。此外,这些CrWRKY 亦可能在长春花萜类吲哚生物碱(TIAs)的生物合成及代谢中起重要作用,因为长春花萜类吲哚碱的生物合成受YE 和MeJA等植物激素诱导。最近,Suttipanta 等[25]克隆和鉴定了一个长春花WRKY 转录因子CrWRKY1(即本文的Cra_locus_16284),该WRKY 在根中特异高表达,且受MeJA、赤霉素和乙烯的诱导,过量表达CrWRKY1使根中蛇根碱的含量提高了3倍多。这与本文该转录因子主要在毛状根中高表达和受MeJA 诱导的研究结果一致。
如上所述毛状根是长春花等一些药用植物合成积累萜类吲哚生物碱等次生代谢物的重要器官,而且长春花遗传转化体系目前多采用发根农杆菌介导的毛状根体系。因此,分析长春花毛状根中CrWRKY 表达谱有助于认识毛状根中萜类吲哚生物碱等次生代谢物合成调控机制。TDC是萜类吲哚生物碱生物合成的一个关键酶,催化色氨酸生成萜类吲哚生物碱生物合成的起始底物即色胺。RebH/F 是催化色胺生成卤代色胺(另一种供生物碱合成的前体物质)的关键酶。长春花TDCi 转基因毛状根是RNA 介导TDC 基因沉默的毛状根,该毛状根中色胺含量明显减少。长春花RebH/F 转基因毛状根能合成卤代色胺。显然这两种长春花转基因毛状根中用于生物碱生物合成的前体物质发生较大变化。我们先期对已知的一些参与萜类吲哚生物碱生物合成的关键酶基因启动子分析发现,几乎所有启动子都含WRKY 转录因子特异结合的元件W-box,其中TDC 启动子包含有4个W-box,这暗示长春花CrWRKY 转录因子可能参与TIAs 合成途径的调控。为此,本文比较分析野生型毛状根与这两种转基因毛状根中CrWRKY 基因表达谱,以期进一步解析CrWRKY 参与萜类吲哚生物碱生物合成的调控作用,为发现更多的参与调控萜类吲哚生物碱生物合成的WRKY 转录因子,特别是响应体内不同前体物质的WRKY 提供科学依据。表达谱分析结果(图5)显示,除了少数CrWRKY (如Cra_locus_6519和 Cra_locus_16307等)在这3种毛状根中均高表达外,多数CrWRKY (如 Cra_locus_1311、Cra_locus_9152和Cra_locus_13760等)在这3种毛状根间表达量发生显著变化,表明这些CrWRKY 可能参与对毛状根中生物碱生物合成的前体物质变化的响应以及下游某些生物碱合成积累的调控。
为进一步验证数字基因表达谱分析所揭示的CrWRKY 表达模式,我们选取16个CrWRKY为靶标,用qPCR 检测它们在不同器官,以及在MeJA 处理的原生质体和毛状根中的表达。结果显示,绝大多数所测CrWRKY 表达模式与数字基因表达谱分析结果基本吻合(图5、6)。只有2个CrWRKY 表达模式的qPCR 和数字基因表达谱分析结果不一致。这两种表达分析数据还显示,除CrWRKY1,其他一些CrWRKY 也可能参与TIA 生物合成途径的信号调控网络。深入研究这些CrWRKY 的生物学功能,将为全面解析长春花TIAs 生物合成的调控机制和提高长春花抗癌生物碱生物合成的代谢工程策略提供有益的信息。
一些研究认为,蛋白序列结构相近的WRKY转录因子,常有类似的表达模式,行使相似的生物学功能[26-27]。然而,我们的表达谱数据分析显示同一类群(或亚类)的CrWRKY 成员多数的表达模式不完全一致。这意味着结构相似的CrWRKY 成员,它们的表达模式和功能也可能是相异的。这正是WRKY 转录因子家族的典型特征,即WRKY 结构域高度保守和功能多样性。因此,未来需进行更多详尽研究,以全面认识本文所鉴定的47个CrWRKY 成员的具体生物学功能和调控靶基因或目标代谢途径表达的分子机理。
[1]Rushton PJ,Somssich IE,Ringler P,et al.WRKY transcription factors.Trends Plant Sci,2010,15(5):247−258.
[2]Eulgem T,Rushton PJ,Robatzek S,et al.The WRKY superfamily of plant transcription factors.Trends Plant Sci,2000,5(5):199−206.
[3]Sun C,Palmqvist S,Olsson H,et al.A novel WRKY transcription factor,SUSIBA2,participates in sugar signaling in barley by binding to the sugar-responsive elements of the iso1 promoter.Plant Cell,2003,15(9):2076−2092.
[4]Ishiguro S,Nakamura K.Characterization of a cDNA encoding a novel DNA-binding protein,SPF1,that recognizes SP8 sequences in the 5'upstream regions of genes coding for sporamin and beta-amylase from sweet potato.Mol Gen Genet,1994,244(6):563−571.
[5]Riechmann JL,Heard J,Martin G,et al.Arabidopsis transcription factors:genome-wide comparative analysis among eukaryotes.Science,2000,290(5499):2105−2110.
[6]Pérez-Rodríguez P,Riaño-Pachón DM,Corrêa LGG,et al.PlnTFDB:updated content and new features of the plant transcription factor database.Nucleic Acids Res,2010,38(suppl 1):D822−D827.
[7]Goff SA,Ricke D,Lan TH,et al.A draft sequence of the rice genome (Oryza sativa L.ssp.japonica).Science,2002,296(5565):92−100.
[8]Zhou QY,Tian AG,Zou HF,et al.Soybean WRKY-type transcription factor genes,GmWRKY13,GmWRKY21,and GmWRKY54,confer differential tolerance to abiotic stresses in transgenic Arabidopsis plants.Plant Biotechnol J,2008,6(5):486−503.
[9]Schmutz J,Cannon SB,Schlueter J,et al.Genome sequence of the palaeopolyploid soybean.Nature,2010,463(7278):178−183.
[10]Liu JJ,Ekramoddoullah AK.Identification and characterization of the WRKY transcription factor family in Pinus monticola.Genome,2009,52(1):77−88.
[11]Jiang T,Lin YX,Liu X,et al.Genome-wide analysis of the WRKY transcription factor family in Medicago truncatula.Acta Pratacul Sin,2011,20(3):211−218(in Chinese).江腾,林勇祥,刘雪,等.苜蓿全基因组WRKY转录因子基因的分析.草业学报,2011,20(3):211−218.
[12]Wu KL,Guo ZJ,Wang HH,et al.The WRKY family of transcription factors in rice and Arabidopsis and their origins.DNA Res,2005,12(1):9−26.
[13]Zhang Y,Wang L.The WRKY transcription factor superfamily:its origin in eukaryotes and expansion in plants.BMC Evol Biol,2005,5:1−12.
[14]Zhou ML,Shao JR,Tang YX.Production and metabolic engineering of terpenoid indole alkaloids in cell cultures of the medicinal plant Catharanthus roseus (L.) G.Don (Madagascar periwinkle).Biotechnol Appl Biochem,2009,52(Pt 4):313−323.
[15]Tamura K,Peterson D,Peterson N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods.Mol Biol Evol,2011,28(20):2731−2739.
[16]Larkin MA,Blackshields G,Brown NP,et al.Clustal W and Clustal X version 2.0.Bioinformatics,2007,23(21):2947−2948.
[17]Yang ZR,Wang XC,Xue JA,et al.An efficient transgenic system of Catharanthus roseus mediated by Agrobacterium rhizogens.Plant Physio J,2012,48(10):997−1004(in Chinese).杨致荣,王兴春,薛金爱,等.发根农杆菌介导的长春花高效转基因体系的建立.植物生理学报.2012,48(10):997−1004.
[18]Suttipanta N,Pattanaik S,Gunjan S,et al.Promoter analysis of the Catharanthus roseus geraniol 10-hydroxylase gene involved in terpenoid indole alkaloid biosynthesis.Biochim Biophys Acta,2007,1769(2):139−148.
[19]Pattanaik S,Kong Q,Zaitlin D,et al.Isolation and functional characterization of a floral tissue-specific R2R3 MYB regulator from tobacco.Planta,2010,231(5):1061−1076.
[20]Ling J,Jiang W,Zhang Y,et al.Genome-wide analysis of WRKY gene family in Cucumis sativus.BMC Genomics,2011,12:471−490.
[21]Wei KF,Chen J,Chen YF,et al.Molecular phylogenetic and expression analysis of the complete WRKY transcription factor family in maize.DNA Res,2012,19(2):153−164.
[22]Qiu YP,Yu DQ.Over-expression of the stress-induced OsWRKY45 enhances disease resistance and drought tolerance in Arabidopsis.Environ Exp Bot,2009,65(1):35−47.
[23]Jiang Y,Deyholos M.Functional characterization of Arabidopsis NaCl-inducible WRKY25 and WRKY33 transcription factors in abiotic stresses.Plant Mol Biol,2009,69(1-2):91−105.
[24]Ramamoorthy R,Jiang SY,Kumar N,et al.A Comprehensive transcriptional profiling of the WRKY gene family in rice under various abiotic and phytohormone treatments.Plant Cell Physiol,2008,49(6):865−879.
[25]Suttipanta N,Pattanaik S,Kulshrestha M,et al.The transcription factor CrWRKY1 positively regulates the terpenoid indole alkaloid biosynthesis in Catharanthus roseus.Plant Physiol,2011,157(4):2081−2093.
[26]Li S,Fu Q,Chen L,et al.Arabidopsis thaliana WRKY25,WRKY26,and WRKY33 coordinate induction of plant thermotolerance.Planta,2011,233(6):1237−1252.
[27]Lai Z,Vinod KM,Zheng Z,et al.Roles of Arabidopsis WRKY3 and WRKY4 transcription factors in plant responses to pathogens.BMC Plant Biol,2008,8(1):1−13.

