表面增强拉曼光谱快速鉴别无乳链球菌的研究
王毅谦,徐 丽,谢云飞,邵景东,*,姚卫蓉
(1.张家港出入境检疫检疫局,江苏张家港215633;2.江南大学食品科学与工程学院,江苏无锡214122)
链球菌属的病原微生物作为一类食源性致病菌,常见有无乳链球菌、牛链球菌、乳房链球菌和耐久链球菌等[1]。链球菌属的致病菌多见于牛乳中,其中无乳链球菌和乳房链球菌等的感染是引发奶牛乳房炎症进而污染乳品危害人畜的的主要原因之一[2]。因此,需要监控乳及乳制品中链球菌属的污染状况以保证食品安全。针对该类致病菌的检测方法主要有常规培养方法、全自动鉴定方法和PCR技术。全自动方法采用初步分离鉴定后结合VITEK32型全自动细菌鉴定仪鉴定,需要分离、镜检和纯化等步骤,虽然全自动鉴定仪检测细菌需要2~18h[3],可初步分离鉴定的时间约耗费36~48h[4];而PCR的操作要求高,需要提取基因组DNA,设计目的基因引物,凝胶电泳分离PCR扩增及目的基因克隆和分析,一般也要耗时2~3d[5]。目前由于表面增强拉曼光谱(SERS)具有良好的分辨能力,尤其是SERS基底对细菌的检测效果增强,实现了超灵敏和快速有效区分各类不同的微生物[6],如Erhan Temur等用金胶基底和大肠杆菌作用可以检测到浓度10~105cfu/mL[7]。因此,本文拟采用表面增强拉曼扫描技术对链球菌属进行检测,应用主成分分析和系统聚类分析方法将其和其他菌属区分,并分类属间细菌。同时,研究能快速鉴别的预培养和检测方式,采用人工接菌方式,考察能监测到无乳链球菌SERS特征峰所需的最短培养时间约为6h,为食品污染快速检测和防控提供基础数据,以期将SERS技术用于食品污染监测或食品安全预警领域。
1 材料与方法
1.1 材料与仪器
牛链球菌ATCC33317、乳房链球菌ATCC19436、无乳链球菌ATCC12386、耐久肠球菌ATCC35667、金黄色葡萄球ATCC12600、单增李斯特氏菌ATCC81861、蜡样芽孢杆菌ATCC11778 均购自美国ATCC;蛋白胨、牛肉膏、琼脂粉、酵母浸膏、氯化钠、磷酸氢二钠(Na2HPO4·12H2O)、磷酸二氢钾、柠檬酸三钠(AR)均购自国药集团化学试剂有限公司;蒸馏水,1mol/L盐酸溶液,1mol/L氢氧化钠溶液,超纯水,氯金酸钾(KAuCl4·2H2O,99%) 美国Alfa;全脂灭菌乳 市售;TSB液体培养基(1000mL) 酪蛋白17.0g,磷酸二氢钾2.5g,大豆蛋白胨3.0g,葡萄糖2.5g,氯化钠5g,pH(7.3±0.2);营养肉汤培养基(1000mL) 蛋白胨10.0g,氯化钠5.0g,牛肉膏3.0g,pH(7.4±0.2);LB培养基(1000mL) 胰蛋白胨10.0g,酵母浸膏5.0g,氯化钠10.0g,琼脂20.0g,pH(7.0±0.2);TSA培养基(1000mL)胰蛋白胨15.0g,大豆胨5.0g,氯化钠5.0g,琼脂13.0g;以上的培养基配制后均用蒸馏水加至1000mL,加热煮沸至完全溶解,121℃高压蒸汽灭菌15min备用。
便携式拉曼检测仪RamTracer®-200-HS OptoTrace Technologies Co.,Ltd.,恒温培养箱;DELTA-320型酸度计、AB104-N分析天平 梅特勒-托利多国际股份有限公司;超纯水制备仪 上海赛鸽电子科技有限公司;KH-500B超声波清洗仪 昆山禾创超声仪器有限公司;荧光定量PCR仪、无乳链球菌Sip Gene目的基因试剂盒 美国ABI 7500。
1.2 实验方法
1.2.1 细菌样品的制备 将甘油保藏的无乳链球菌菌种取出并常温融化,各取1mL菌液分别移至装有50mL TSB和50mL营养琼脂的活化培养基中,于37℃恒温有氧培养24h后,分别接种于LB固体培养基和TSA固体培养基上划线培养,37℃下培养24~48h,得到菌落计数约为100CFU/mL,用无菌接种环挑取分散的两份单一菌落,一份置于无菌蒸馏水中振荡,6500r/min离心5min,弃去上清液,重复三次,得到待测细菌样品A;一份直接置于已灭菌处理的离心管中作为待测细菌样品B。
1.2.2 SERS纳米溶胶的制备 胶制备采取传统的柠檬酸盐还原法,根据相关报道[8]进行条件优化:采用纳米金溶胶作为增强基底,制备优化条件为:精确称取KAuCl4·2H2O 30mg,用超纯水定容至50mL,在油浴中进行回流加热并搅拌至沸腾后,迅速加2mL 1%柠檬酸钠水溶液,继续搅拌35min。冷却到常温即可得到粒径50nm左右的金纳米粒子[9]。
1.2.3 拉曼光谱仪的条件设定 拉曼仪器参数设置为:激光光源波长:785nm;扫描功率:300mW;扫描积分时间:10s,最终谱图为扫描2次取平均值所得。
1.2.4 拉曼光谱检测细菌样品 将上述从TSA和LB固体培养基中得到的待测细菌样品A分别用1mL超纯水溶解,取10μL分别加入到装有200μL的金溶胶的96孔板中。静置5~10s,进行拉曼光谱扫描,得到不同浓度细菌溶液的SERS图谱。同样,将含有待测样品B的离心管中加入200μL的金溶胶混合均匀后进行拉曼扫描。
1.2.5 牛奶样品中无乳链球菌的SERS检测和荧光PCR验证 将100mL牛奶置于250mL的无菌三角瓶中,接种无乳链球菌,在接种后的12h内分别取不同时间的样品每次1mL,时间取点为0、1、2、3、4、6、8、10、12h,将试管放在摇床振荡37℃,150r/min培养,对应各个时间取出试管,放置-20℃保存。后将染菌不同时间的-20℃保存的牛奶样品自然解冻,每个样品取10μL直接与200μL的金胶混合,进行拉曼光谱扫描,得到SERS图谱,以标准菌株的无乳链球菌SERS作为对照。选取上述的人工接种无乳链球菌的牛奶样品,提取基因组DNA,根据祝长青等[10]研究结果方法合成无乳链球菌表面免疫相关蛋白Sip Gene的目的基因引物和探针,采用实时荧光PCR检测方法,验证SERS实验结果。
1.2.6 数据处理 主要采用Origin 8.0制图软件及SPSS数据分析软件处理进行主成分分析和聚类分析。具体是选用拉曼图谱中600~1800cm-1的数据,数据平滑后进行均一化,根据最大值和最小值分别为1和0,使数据分布在0~1之间。然后对数据进行二次求导。主成分分析是在Matlab 7.0软件中进行,系统聚类分析在SPSS 13.0中处理得到。
2 结果与讨论
2.1 无乳链球菌的活化培养基和预培养基的组合和菌落预处理的选择
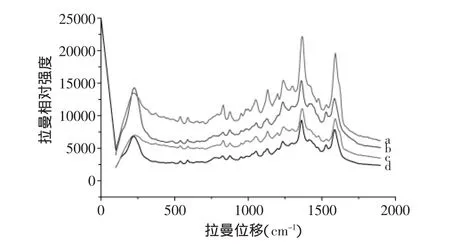
图1 活化和预培养组合对无乳链球菌SERS图谱的影响Fig.1 The effect of different medium on the SERS spectra of Streptococcus agalactiae
2.1.1 活化培养基和预培养基的组合选择 分别组合两种液体活化培养基和两种固体预培养基对无乳链球菌进行活化和预培养,挑取固体培养基上的单菌落与金胶混合,再测定SERS图谱。结果见图1,可以看出,不同组合得到的SERS图谱无论从峰的数目、主要峰的形状等基本一致,没有显著差别,因此,为了结果的一致性,在后续实验中都统一采用的是b的组合,即先用营养肉汤培养基对菌种进行活化,再用TSA固体培养基进行预培养。
2.1.2 菌落预处理与否的选择 以无乳链球菌为对象,经活化和预培养后,分别挑取固体培养基上的单菌落,即样品a,后与金胶混合,将菌落先用超纯水进行洗涤离心后悬浮于超纯水中制成菌悬液,即样品b,再与金胶混合,然后分别测定其SERS图谱(见图2)。结果发现,SERS光谱的峰的数目和峰形基本一致,说明菌落预处理与否对于无乳链球菌的SERS图谱并无明显影响。但样品a的SERS测定显示拉曼相对强度比样品b的大,同时考虑到操作的简便性,后续实验采用将单菌落与金胶直接混合再进行SERS测定的方法。
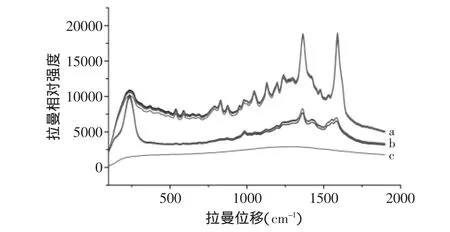
图2 菌落预处理与否对无乳链球菌SERS图谱的影响Fig.2 The effect of colonies pretreatment on the SERS spectra of Streptococcus agalactiae
2.2 链球菌属以及几个常见革兰氏阳性致病菌的SERS图谱和特征峰的归属指认

图3 几种常见革兰氏阳性菌的SERS图谱Fig.3 SERS spectra of several Gram-positive bacteria

表1 革兰氏阳性致病菌的SERS图谱峰的归属指认Table 1 Peaks assignments of SERS spectra of gram positive pathogenic bacteria
由图3和表1可以看出,根据链球菌和几种常见致病菌的SERS对比,可以看到金黄色葡萄球菌和单增李斯特氏菌在730cm-1处的腺嘌呤的峰强较大,同时金黄色葡萄球菌在1314cm-1腺嘌呤、鸟嘌呤,酪氨酸的伸展振动处[11],单增李斯特氏菌在1318cm-1处,蜡样芽孢杆菌在1352cm-1处和单增李斯特氏菌在1352cm-1(CH2基团变形振动和色氨酸振动)[12],链球菌科在1364cm-1(脂质中CH2基团的变形振动)[13]和1590cm-1(酪氨酸)[14]与其他的菌不同。表1中1112cm-1(L-丙氨酸)[15]在链球菌属中并没有出峰,而在其他革兰氏阳性菌中却有SERS信号;另外链球菌在1130cm-1[16]处,主要是糖类的CN和CC基团的伸缩振动,还有1236cm-1[17](CH2基团的伸缩振动)也是区别于其他的金黄色葡萄球菌、蜡样芽孢杆菌和单增李斯特氏菌的SERS特征峰。
2.3 用主成分分析(PCA)和系统聚类分析(HCA)分类链球菌属及几种常见革兰氏阳性致病菌
为了有效区分各菌,本论文采用主成分分析方法对几种菌的SERS谱图进行分类。从图4中可以看出单增李斯特氏菌、金黄色葡萄球菌、蜡样芽孢杆菌和链球菌科的细菌是在三维的PCA中可以分开,如蜡样芽孢杆菌和金黄色葡萄球菌的第一主成分上在-0.1~0的范围内,二者的区别主要是在第二和第三主成分上;单增李斯特氏菌和链球菌的第一主成分主要在0.2~0.3,它们的区别主要也是在第二、三主成分,前者的第三主成分主要是-0.1~0.2,后者的是在-0.2~-0.1。PCA结果分析,提取三个主成分,累积贡献率可以达到94.58%,前三个主成分分别为65.15%、23.33%和6.10%。但图4中的链球菌科的几种菌的分类不是很清楚,原因推测可能是和其他革兰氏阳性菌的差别较大,掩盖了科内细菌的差别,因此就将几种链球菌的致病菌的SERS光谱做同种的主成分分析处理,如图5所示。PCA结果分析显示,只需提取两个主成分就可以使累计贡献率达到97.43%,第一、二主成分的百分比分别为82.92%和14.51%。同样SERS图谱数据进行系统聚类分析(HCA)后得出的分类图如图6所示,和PCA分析图一致。HCA结果根据欧氏距离的Ward-linkage方法[18]处理得到,链球菌属的无乳链球菌、耐久链球菌、乳房链球菌和牛链球菌可以归类在一起,且区别于其他革兰氏阳性细菌。
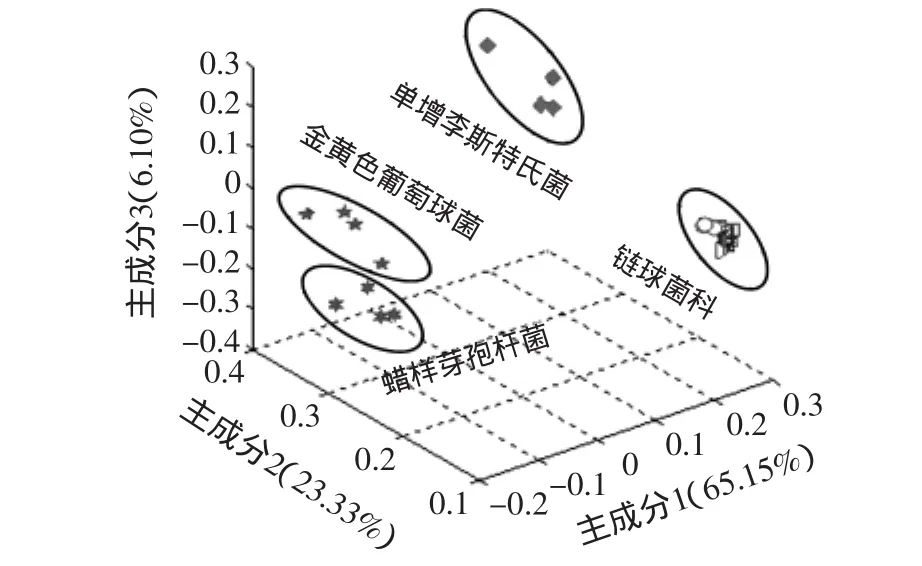
图4 革兰氏阳性致病菌的PCA分析图Fig.4 PCA plot of gram positive pathogenic bacteria
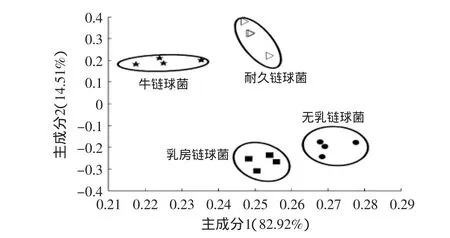
图5 链球菌的PCA分析图Fig.5 PCA plot of Streptococcus genus
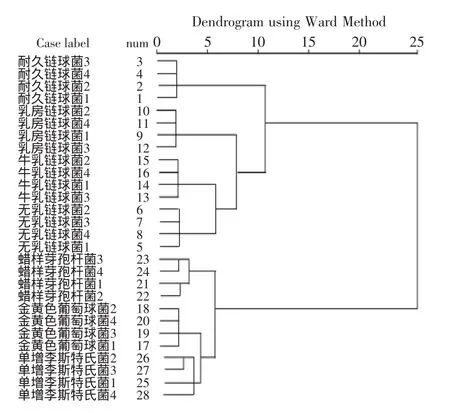
图6 链球菌属及常见食源性致病菌的系统聚类分析图Fig.6 HCA plot of Streptococcus genus and other common pathogenic bacteria
2.4 定性分析牛奶中的无乳链球菌的SERS图谱和荧光PCR验证

图7 牛奶体系中培养不同时间无乳链球菌的SERS图谱Fig.7 SERS spectra of S.agalactiae cultivated with different time in milk
经过不同时间培养的无乳链球菌和标准菌株的无乳链球菌的SERS图谱对比如图7所示,其中0和10分别对应未加菌株的牛奶和标准菌株按照上述样品b方法测定拉曼得到SERS曲线,1~9对应的是不同培养时间的图谱,分别是0、1、2、3、4、6、8、10、12h。通过与0号未添加无乳链球菌的牛奶样品(基本没有SERS信号)对比,从图7中可以看出在0~4h内接种培养后并没有很明显的SERS效果,6~12h的图谱有比较明显的SERS效果,而且在一些特征峰如999、1196、1362、1586cm-1有出峰,分别代表的是DNA骨架中C-O键或者苯丙氨酸的振动[14],芳香族蛋白质的振动,脂质中-CH的伸缩振动和酪氨酸的振动[19]。一些标准菌株特征峰在实际样品中约几个波数偏移,如1132、1238、1532cm-1,分别指代为糖类物质-CN和-CC的振动[16],-CH2变形振动[17]和反对称的ν(NO2-)[20]。结果显示人工染菌的牛奶在约6h的时候经过金胶基底增强就可以得到拉曼信号,实时荧光PCR结果显示,无乳链球菌在牛奶中各个时间采样点都可以检出(见图8),与SERS检测结果一致。
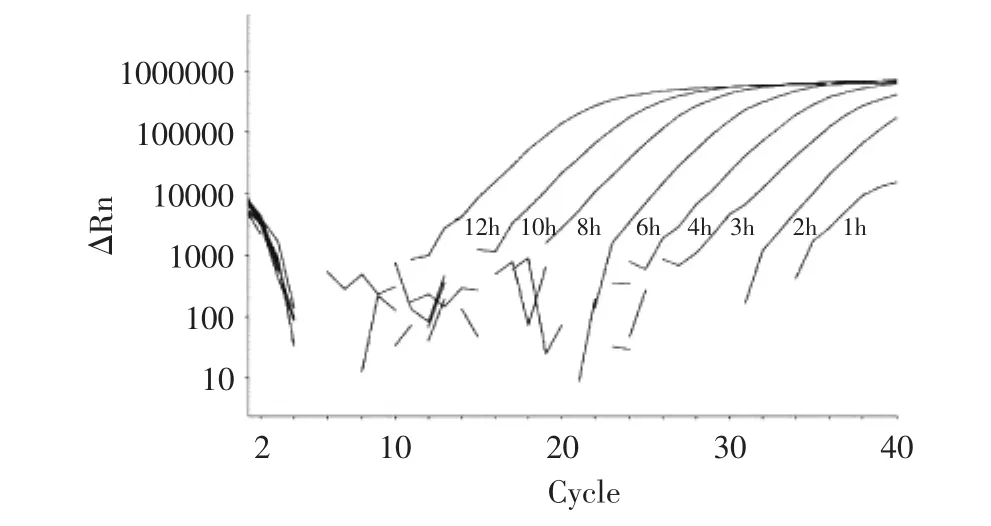
图8 牛奶体系中培养不同时间无乳链球菌的实时荧光PCR结果Fig.8 The amplification curves of Real-Time Fluorescence PCR of S.agalactiae cultivated with different time in milk
3 结果与讨论
本文采用优化后的组合培养基活化和预培养细菌方法,在挑取单菌落后直接混合金胶直接测定SERS图谱,可以快速有效的检测食源性致病菌,并依据SERS图谱数据将链球菌属和其他几种常见革兰氏阳性食源性致病菌的主成分分析和系统聚类分析分类,进行有效区分;采用SERS技术针对实际样品中的无乳链球菌的检测,检出起始时间约为6h即可得到较好的拉曼信号,检出的SERS图谱和对应标准菌株的图谱在999、1196、1362、1586cm-1等特征峰处有对应关系。采用荧光PCR技术进行验证,与SERS检测结果一致。
近些年来,随探测技术的改进和对被测样品要求的降低,SERS技术在物理、化学、医药、工业等各个领域得到了广泛的应用,越来越受研究者的重视。但是在微生物检测领域才刚刚起步,主要受制于其基底制备技术的运用。本文对SERS技术应用于快速筛查乳制品致病菌做了初步的探索和尝试,并获得较为可靠的数据,为进一步将SERS技术应用于快速筛查食品中食源性致病菌奠定基础,为今后在该领域的工作提供有力参考,希望以此促进纳米SERS基底在检测食品安全和质量控制领域的应用。
[1]李金钟.链球菌分类与鉴定新进展[J].临床检验杂志,2006,24(5):391-393.
[2]Schalm OW,Bovine Mastitis[M].USA:Lea & Febiger,1971:18-21.
[3]陶庆春.细菌自动鉴定及药敏系统的研究进展[J].临床检验及实验室设备,2005,7(2):37-39.
[4]邵雪花,陈创夫,乔军,等.奶样中无乳链球菌和乳房链球菌的分离与鉴定[J].黑龙江畜牧兽医,2009(9):86-88.
[5]王丽君,张利莉.奶牛乳房炎相关链球菌PCR检测方法的建立[J].畜牧与兽医,2010,42(10):7-9.
[6]汪朋,姚卫蓉.表面增强拉曼光谱检测食源性细菌的增强方式[J].食品工业科技,2012,33(4):427-433.
[7]Erhan Temur,smail Hakk Boyac,Ugˇur Tamer,et al.A highly sensitive detection platform based on surface-enhanced Raman scattering for Escherichia coli enumeration[J].Anal Bioanal Chem,2010,397(4):1595-1604.
[8]Yao W,Sun Y,Xie Y,et al.Development and evaluation of a surface-enhanced Raman scattering(SERS) method for the detection of the antioxidant butylated hydroxyanisole[J].European Food Research and Technology.2011,233(5):835-840.
[9]黄玉坤,王毅谦,汪朋,等.表面增强拉曼光谱快速鉴别食品中的金黄色葡萄球菌[J].中国卫生检验杂志,2011,21(12):2824-2827.
[10]祝长青,封莉,蒋鲁岩,等.牛奶中无乳链球菌DNA提取方法的比较[C].2011食品安全技术与标准国际研讨会暨AOAC中国区会议论文集,2011:235-240.
[11]Roger M,Jarvis,Nicholas Law,et al.Surface-enhanced Raman scattering from intracelluLar and extracelluLar bacterial locations[J].Anal Chem,2008,80(17):6741-6746.
[12]John Clarkson,Caroline Sudworth,Sergiu I Masca,et al.ΜLtraviolet resonance Raman study of the avidin biotin complex[J].Raman Spectrosc,2000,31(5):373-375.
[13]YuLing Wang,Kyuwan Lee,Joseph Irudayaraj.Silver nanosphere SERS probes for sensitive identification of pathogens[J].J Phys Chem C,2010,114(39):16122-16128.
[14]Naumann D.FT-infrared and FT-Raman spectroscopy in biomedical research[J].Appl Spectrosco Reviews,2001,36(2):239-298.
[15]Joke De Gelder,Kris De Gussem,et al.Reference database of Raman spectra of biological molecules[J].Journal of Raman Spectroscopy,2007,38(9):1133-1147.
[16]K C Schuster,E Urlaub,J R Gapes.Single-cell analysis of bacteria by Raman microscopy:spectral information on the chemical composition of cells and on the heterogeneity in a culture[J].Journal of Microbiological Methods,2000,42:29-38.
[17]Cui Fan,Zhiqiang Hu,Azlin Mustapha,et al.Rapid detection of food-and waterborne bacteria using surface-enhanced Raman spectroscopy coupled with silver nanosubstrates[J].Appl Microbiol Biotechnol,2011,92:1053-1061.
[18]A K Kniggendorf,T W Gaul,M Meinhardt-Wollweber.Hierarchical Cluster Analysis(HCA) of Microorganisms:An Assessment of Algorithms for Resonance Raman Spectra[J].Applied Spectroscopy,2011,65:165-173.
[19]Cui Fan,Zhiqiang Hu,Lela K Riley,et al.Detecting Food and Waterborne Viruses by Surface-Enhanced Raman Spectroscopy[J].Journal of Food Science,2010,75(5):303-307.
[20]朱自莹,顾仁敖,陆天虹.拉曼光谱在化学中的应用[M].沈阳:东北大学出版社,1998:305-307.

