刺参26S蛋白酶体S7亚基基因的克隆与组织表达分析
赵祥吉,秦艳杰,李霞,刘洋,丁鉴峰
(1.大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,辽宁大连116023;2.大连海洋大学农业部北方海水增养殖重点实验室,辽宁大连116023;3.江西省上饶市鄱阳中学,江西上饶 333100)
刺参26S蛋白酶体S7亚基基因的克隆与组织表达分析
赵祥吉1、3,秦艳杰1、2,李霞1,刘洋1,丁鉴峰1
(1.大连海洋大学辽宁省海洋生物资源恢复与生境修复重点实验室,辽宁大连116023;2.大连海洋大学农业部北方海水增养殖重点实验室,辽宁大连116023;3.江西省上饶市鄱阳中学,江西上饶 333100)
应用RACE法从刺参Apostichopus japonicus体腔细胞中克隆出26S蛋白酶体组成19S亚复合体亚基之一的S7亚基基因 (GenBank登录号:JQ922514)。该基因的cDNA序列全长为2 445 bp,其中5'UTR为70 bp,3'UTR为1 070 bp,开放阅读框为1 305 bp,编码435个氨基酸;保守区具有典型的ATP结合和水解基序,氨基酸序列分别为GPPGTGKT和DEID,具有MAT和VRPGRLDR的RNA/DNA螺旋酶的特征性基序;刺参26S蛋白酶体S7亚基基因与紫海胆的氨基酸序列同源性最高 (95%),与脊椎动物、节肢动物的同源性较高 (88%~89%),与线虫、拟南芥和酵母同源性较低 (85%、77%和73%);S7亚基编码的蛋白没有信号肽,也没有跨膜结构域,为非跨膜蛋白,定位于细胞中的液态基质中。采用实时定量PCR方法检测了26S蛋白酶体S7亚基基因在刺参5种组织中的表达情况,结果显示,在肠、呼吸树、表皮、体腔细胞、纵肌中26S蛋白酶体S7基因均有明显表达,且体腔细胞中表达量最高,其次是纵肌和肠,在体壁和呼吸树中表达量较低。本研究中首次在刺参体内发现并克隆了26S蛋白酶体S7亚基基因,同时采用Realtime PCR技术对该基因的表达部位进行了研究,所得结果将为研究棘皮动物蛋白质代谢途径提供新的着眼点,为刺参生理功能的调控研究奠定了基础。
刺参;26S蛋白酶体S7亚基;基因克隆;组织表达
在真核生物中,26S蛋白酶体 (26S proteasome,26S PSM)是一种依赖ATP的蛋白酶复合体,该蛋白酶体分子由催化颗粒 (Catalytic particle,CP,又称 20S降解复合体)和调节颗粒(Regulatory particle,RP,又称19S调节复合体)构成,其中20S复合体具有蛋白质水解功能,而19S调节复合体负责对蛋白质底物的识别、去折叠和变性。哺乳动物19S调节复合体由至少20个亚基构成,其中至少有6个具有ATPase酶活性的亚基 (包 括 S7、S4、S6b、S10b、S6a 和 S8)[1-2],而S4、S6、S7、S8还可作为转录因子,调节细胞周期蛋白等功能蛋白基因,参与细胞的程序性死亡[2-3]。26S蛋白酶体已在哺乳动物 (尤其是癌症患者)、植物等的研究中成为热点,这是因为该蛋白酶体能够对多种细胞活动进行调控,调控机理是通过“泛肽 (Ubiquitin)依赖性蛋白降解途径”对细胞内的异常蛋白以及参与细胞周期、代谢调控等的功能蛋白进行选择性降解。在水产动物中仅见Horiguchi等[4]利用免疫印迹方法对金鱼26S蛋白酶体α4亚基的磷酸化进行了初步研究;姜铁民等[5]对合浦珠母贝26S蛋白酶体S4、S7亚基基因的部分片段进行了分离与分析。有关其他水产动物26S蛋白酶体的研究国内外尚未见报道。
刺参Apostichopus japonicus又名仿刺参,隶属于棘皮动物门、海参纲、刺参科、仿刺参属,是中国黄渤海地区的主要养殖品种之一,经济价值较高。刺参的再生能力较强,近几年关于其再生的生物学特征和分子机制等方面的研究已成为国内外研究的热点[6-7],也为医学研究提供了重要启示。国内对刺参再生的研究主要集中在其形态学、组织学等方面[8-9],而对再生的分子机理探讨则刚刚起步。周遵春等[10]构建了刺参多个组织的全长cDNA文库并进行了EST初步分析;Sun等[11]对刺参再生的肠道和体壁进行了转录组测序研究。李霞等[12]首次构建了刺参体壁再生的差减文库,并在此基础上进行了再生相关基因的克隆研究。这些研究工作都为刺参功能基因的研究以及再生分子机理的探讨奠定了基础。作者在通过差减文库研究刺参体壁再生的过程中,发现了刺参26S蛋白酶体S7亚基部分序列在刺参再生体壁中上调表达,由此首次克隆得到了该基因的全长,并进行了该基因在刺参不同组织中的表达分析。鉴于S7亚基基因在哺乳动物和植物中调节细胞周期、信号转导等方面的重要作用,结合刺参具有超强再生能力的特点,本研究结果将有助于揭示刺参再生的分子机理,并将揭开棘皮动物蛋白质降解途径研究的序幕。
1 材料与方法
1.1 材料
试验用刺参 (体长为6~8 cm)于2012年3月采自旅顺龙王塘海区,暂养在大连海洋大学农业部北方海水增养殖重点实验室300 L的塑料水槽中,培育用水为沙滤海水,每天换水1/2,投喂人工配合饵料,水温控制在 (18±1)℃,连续充气,暂养1个月以上。
1.2 方法
1.2.1 总RNA的提取和cDNA合成 挑选健康刺参,用注射器抽取体腔液,将体腔液以4 500 r/min离心10 min,弃上清收集细胞,按照李霞等[12]的方法进行RNA提取、纯化和电泳检测。将纯化后的RNA于-80℃下保存备用。按照反转录试剂盒(TaKaRa公司)说明书进行RNA反转录。
1.2.2 RACE扩增 由李霞等[12]构建的刺参体壁再生差减文库,得到一段长330 bp的序列,经Blastx比对分析,发现该序列与26S蛋白酶体S7亚基基因的同源性较高。以此为目的片段,采用SMART RACE试剂盒 (Clontech公司)进行基因全长的克隆,操作方法按照说明书进行,其中涉及到的引物见表1。扩增体系共25 μL,包括2.5 μL 10×PCR Buffer,2.0 μL MgCl2(25 mmol/L),2.0 μL dNTP Mixture(各 2.5 mmol/L),引物 UPM/NUP 2.5 μL,引物调节亚基3'或5'/调节亚基3'EN或 5'EN 1.0 μL,ddH2O 13.0 μL。PCR 反应条件为:94℃下预变性5 min;94℃下变性30 s,51℃ (3'RACE)/55℃ (5'RACE)下退火30 s,72℃下延伸2 min,共进行35个循环;最后在72℃下延伸10 min。
取5 μL PCR产物用10 g/L琼脂糖凝胶进行电泳,采用琼脂糖凝胶DNA回收试剂盒 (TaKaRa公司)切胶回收产物。将PCR产物纯化后连接到PMD19-T(TaKaRa公司)载体中,于16℃下连接过夜。将连接好的载体热转化至感受态细胞E.Coli DH5α(TaKaRa公司)中,并在涂有IPTG、Amp的平板上过夜培养 (培养箱温度为37℃),然后挑取蓝菌加入到含有Amp的1 mL培养液中进行扩大培养。用引物Rv-m、M13-47(表1)对菌液进行PCR检测,并将检测合格的菌液送上海生工生物工程有限公司进行测序。
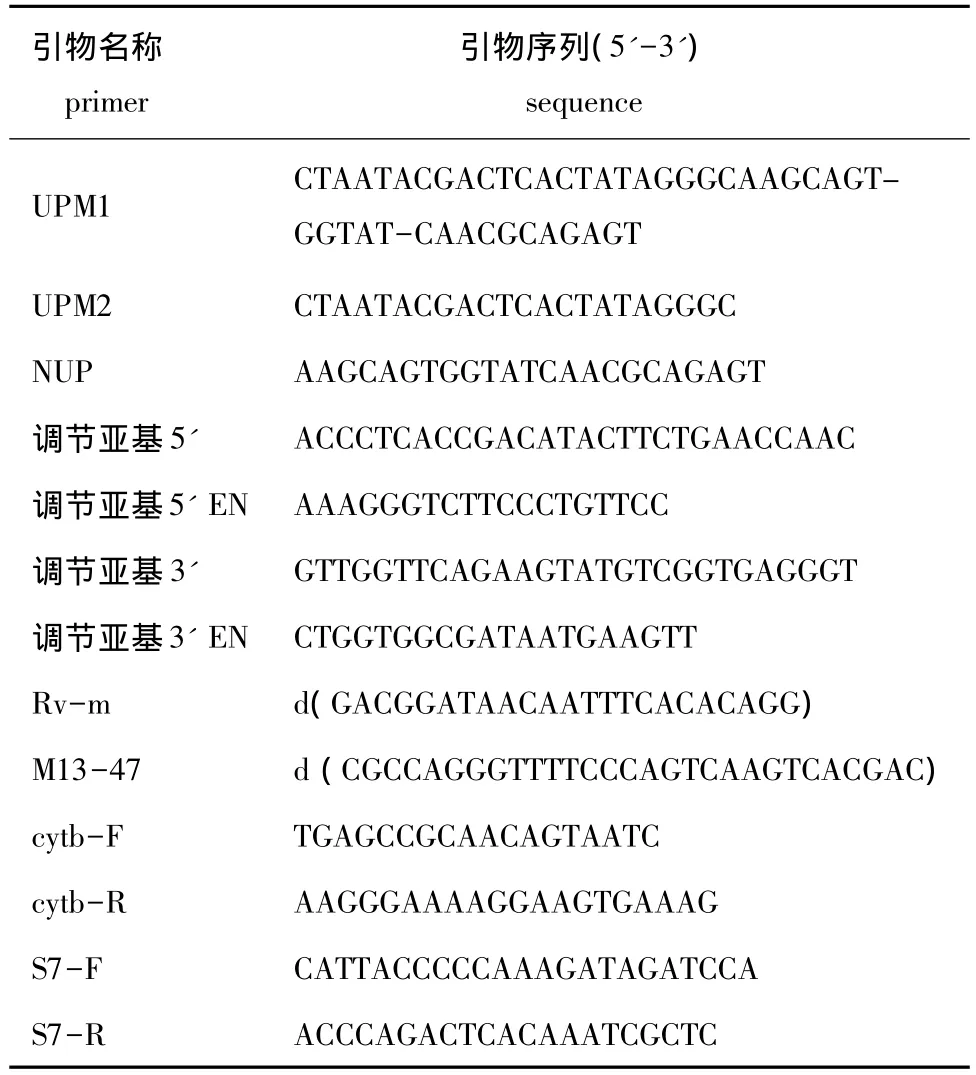
表1 试验引物序列Tab.1 Sequences of PCR primers in this study
1.2.3 生物信息学分析与系统进化树的构建 用DNAStar软件将S7亚基基因cDNA序列翻译成氨基酸序列,并确定刺参26S蛋白酶体S7亚基基因的开放阅读框;在Genbank数据库中用Blastx程序进行序列比对;采用 ProtParam、ProtScale、TMHMM Server 2.0软件预测该基因编码蛋白的理化性质、亲疏水性和跨膜结构域;采用SingnalP 3.0 Server、ExPaSy-motifscan和 Interproscan软件进行motify区域和信号肽的预测。在www.predictprotein.org网页上进行蛋白质二级结构的预测。用Clustal W软件将本研究中获得的刺参26S蛋白酶体S7亚基基因序列与其他物种相应序列进行比对,并用Mega 5.0软件绘制26S蛋白酶体S7亚基基因的系统进化树。
1.2.4 26S蛋白酶体S7亚基基因在刺参5种组织中的表达检测 随机选取实验室培育的刺参5只,割取体壁并解剖,取其肠、纵肌、呼吸树各约200 mg,同时抽取体腔液,按照李霞等[12]的方法提取各组织的RNA并反转录得到cDNA。根据已获得的刺参26S蛋白酶体S7亚基基因全长序列设计Real-time PCR 引物 S7-F和S7-R,选取 cytb为内参基因,其引物序列见表1。
荧光定量 PCR反应体系共50 μL,包括2×PCR buffer 25 μL,Primers(25 pmol/μL)2 ×1 μL,Sybr green 0.5 μL,模板 (cDNA)2 μL,DEPC 水20.5 μL。荧光定量PCR扩增条件为:94℃下预变性4 min;94℃下变性20 s,60℃下退火30 s,72℃下延伸30 s,共进行35个循环;在72℃下检测信号。采用FTC2000(Canada公司)实时荧光定量PCR仪进行扩增,每个样品重复扩增检测3次。最后将得到的样品内参基因和目的基因的C1值分别代入各自的标准曲线,得出目的基因mRNA的相对表达量。
1.3 数据处理
各组织的表达量采用SPSS 16.0软件进行单因素方差分析,显著性水平设为0.05。
2 结果
2.1 刺参26S蛋白酶体S7亚基基因序列及结构分析
刺参26S蛋白酶体S7亚基基因的cDNA序列全长为2 445 bp,其中5'UTR为70 bp,3'UTR为1 070 bp,开放阅读框为1 305 bp,编码435个氨基酸。保守序列搜索及序列分析表明,该基因的ATP酶组件序列区 (M1)位于第212~395位氨基酸处,其中第217~224位氨基酸 (GPPGTGKT)为ATP的结合基序,第276~279位氨基酸(DEID)为ATP的水解基序,第320~322位和第333~340位氨基酸分别为RNA/DNA螺旋酶的特征性基序MAT和VRPGRLDR。序列已提交至Genbank中 (GenBank登录号:JQ922514)。该基因在部分生物的保守区序列及特征序列比对结果见图1,经比较发现,各种生物的ATP酶结合基序与水解基序完全相同,而RNA/DNA螺旋酶的特征性基序有所差异,其中绝大多数生物均为MAT序列,仅酵母为FAT序列;另一个螺旋酶特征基序在脊椎动物、节肢动物和线虫中是MRPGRLDR,在棘皮动物 (海参、海胆)中为VRPGRLDR,在酵母和拟南芥中则分别为LRPGRIDR和LRSGRLDR。
S7亚基的分子式为C2134H3445N601O648S19,原子总数为6 847,相对分子量为48 498.6,等电点为6.02,正电荷残基 (Arg+Lys)总数为64,负电荷残基 (Asp+Glu)总数为68,不稳定系数为35.11,通过计算,该蛋白属于稳定蛋白,脂肪系数为85.85,亲水评估为-0.419,可见该蛋白为亲水蛋白。亚基亚细胞定位的预测结果显示,S7亚基没有信号肽,也没有跨膜结构域,为非跨膜蛋白,定位于细胞中的液态基质中。采用PROF法预测S7亚基的二级结构,有44.24%的α螺旋,10.14%的β折叠,45.62%的α螺旋和β折叠复合体。S7亚基的三级结构如图2所示。
2.2 同源性比较及生物进化
将刺参26S蛋白酶体S7亚基氨基酸序列使用在线Blastx软件进行同源性比较,得到结果见表2。该基因序列与紫海胆Strongylocentrotus purpuratus同源性最高,达到95%,与高等哺乳动物、鸟类、鱼类、两栖类和节肢动物等模式动物之间的同源性为88%~89%,与酵母和拟南芥的同源性较低,分别为73%和77%。设置Bootstrap置信值为1 000,应用Mega 5.0软件构建的系统进化树见图3,其中刺参和紫海胆的亲缘关系最近,独立聚为一支,脊椎动物和节肢动物各独立聚为一支,线虫、拟南芥和酵母则单独聚为一支。
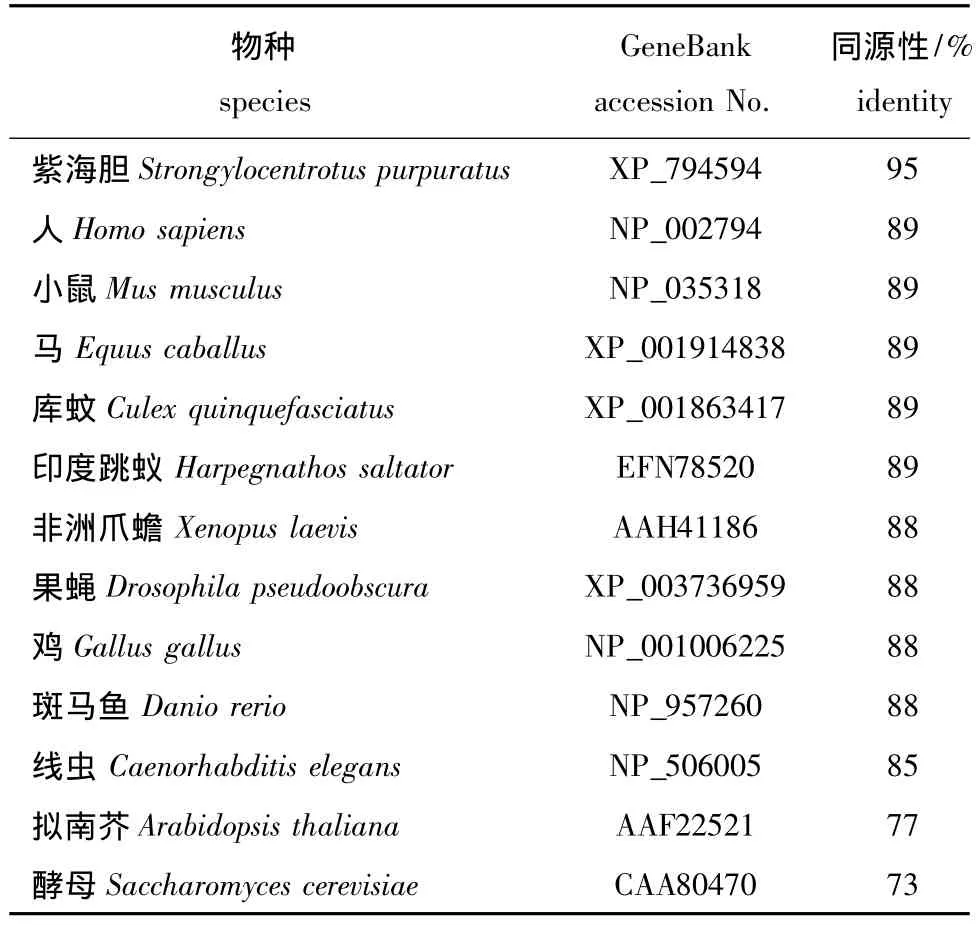
表2 不同物种氨基酸序列的同源性比对结果Tab.2 The amino acid sequence identities of different species
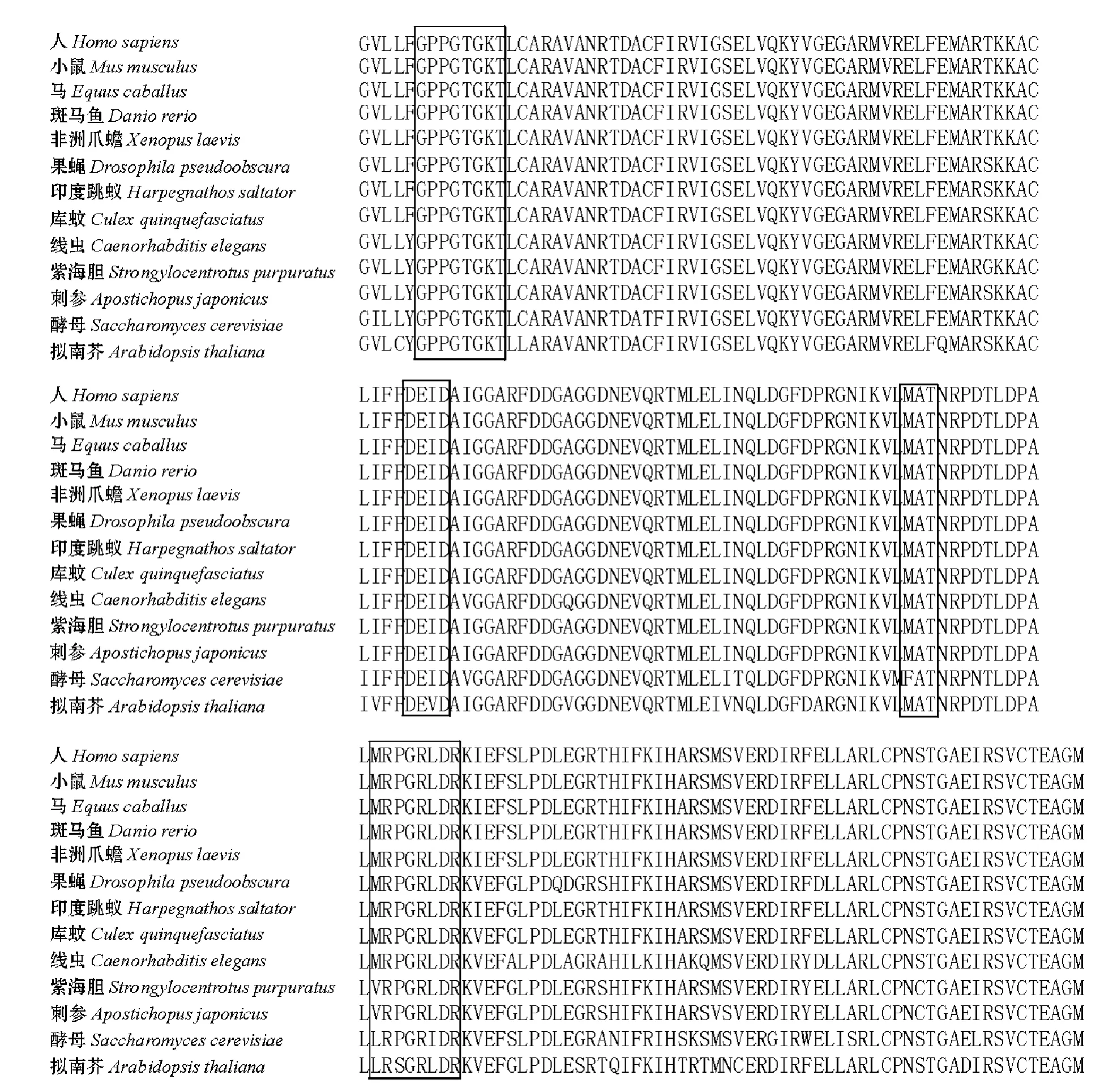
图1 不同生物来源的S7亚基ATP酶组件的氨基酸序列比较Fig.1 Amino acid sequence alignment of subunit 7 ATPase from various organisms
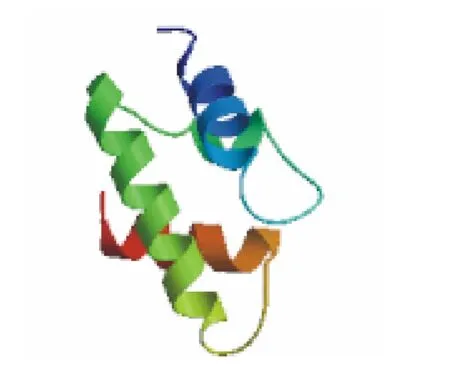
图2 S7亚基的三级结构Fig.2 The three-dimensional constitution of S7 subunit predicted by Swiss-mode
2.3 26S蛋白酶体S7亚基基因在刺参不同组织中的表达
荧光实时定量PCR检测结果表明,S7亚基基因在刺参体壁、肠、呼吸树、纵肌和体腔细胞中都有表达 (图4)。经单因素方差分析表明,各组织中S7亚基基因的表达量依次为体腔细胞>纵肌带>肠>体壁>呼吸树 (P<0.05)。可见,该蛋白酶体的调节颗粒中ATP酶活性亚基S7在刺参体腔液中表达量最高 (P<0.05),而在体壁和呼吸树中该基因的表达量最低 (P<0.05)。

图3 刺参S7亚基与其同源体的系统发育树Fig.3 The phylogenetic tree between S7 subunit unit in sea cucumber Apostichopus japonicus and their homologies from other species

图4 刺参各组织中S7亚基基因的mRNA相对表达量Fig.4 Relative quantity of S7 subunit gene expressions in the different tissues of sea cucumber Apostichopus japonicus
3 讨论
本研究中,首次在刺参中发现了26S蛋白酶体S7亚基基因的部分序列,并对该基因进行了全长克隆,发现刺参S7亚基基因编码的氨基酸序列与其他物种的S7亚基基因有着高度的同源性 (73%以上)。这些生物都具有相似的ATP酶亚基保守序列和功能位点,且ATP酶组件中都具有ATP结合基序和水解基序 (氨基酸序列分别为GPPGTGKT和DEID)两个特征结构域,也同时存在RNA/DNA螺旋酶的特征性基序MAT和VRPGRLDR。而且,这些生物GPPGTGKT和DEID之间的氨基酸残基均为51个,DEID与MAT之间相隔的氨基酸残基均为40个,而MAT与VRPGRLDR之间间隔的氨基酸残基数目均为10个。这些极其类似的结构特征说明这4个保守序列在S7亚基发挥生物学功能上起着关键作用,同时也说明26S蛋白酶体S7亚基在结构和功能上保守性较高。真核细胞中,蛋白质降解的途径一般有两条:一条是通过溶酶体进行非特异性降解,主要涉及的蛋白包括膜蛋白和通过胞吞作用获取的外源蛋白;另一条是通过26S蛋白酶体实现,胞内的绝大多数蛋白 (80%~90%)都是通过这条途径进行降解[13]。当然,只有被多聚泛肽链标记的蛋白才会被26S蛋白酶体降解,这一系统被称为泛素-蛋白酶体系统[14]。泛素-蛋白酶体通路是真核细胞中新蛋白合成的主要数量控制通路[15]。通过调节蛋白的特异性降解,这个通路参与了细胞增殖和生理功能的调控。例如:细胞周期的运行必须在细胞周期蛋白和细胞周期蛋白依赖性激酶 (Cyclin-dependent kinase,CDK)抑制因子及时降解的前提下才能进行[16]。鉴于S7亚基的保守性,作者认为,刺参的蛋白质降解途径可能也涉及26S蛋白酶体,但由于本研究只在刺参中发现了26S蛋白酶体的一个亚基,至于其他亚基结构以及26S蛋白酶体是否与泛素作用乃至作用方式等还有待于深入系统地研究。
有报道指出,S7亚基常常与S4和S2亚基结合在一起形成三聚体,在大麦中发现这两个基因的突变会影响有丝分裂,而在果蝇中发现这两个亚基与细胞增殖和变态相关[17-18];S7亚基同源基因在菠菜发芽期和子叶衰老时表达量增加,分析认为,子叶衰老时该亚基的高表达导致蛋白质降解活动增加,使降解后所得的氨基酸重复利用[19];在患恶性肿瘤病的小鼠中,S7亚基的同源亚基 (MSS1)表达量较其他调节亚基高,因而被认为促进了肌肉蛋白质的水解[20]。也有研究表明,MSS1亚基在患癌小鼠体内的表达量增加与20S亚基并无关联,并不会导致蛋白酶体的组装及蛋白质水解的增加,观察发现,MSS1亚基的mRNA主要分布在多聚核糖体上,表明这个亚基参与了翻译的激活[21]。目前,对水产动物中26S蛋白酶体的功能研究不多,主要集中在基因表达水平的研究上,如Place等[22]研究发现,温度会影响北美西海岸四个地区的贻贝Mytilus californianus蛋白酶体不同亚基的表达模式。对瓷蟹Petrolisthes cinctipes和鰕虎鱼Gillichthys mirabilis的研究发现,在接近生物高温极限时泛素-蛋白酶体通路中的相关基因上调表达[23-24]。长期提高温度会使鳉Austrofundulus limnaeus具有一定的环境适应性,从而蛋白酶体调节亚基表达反应较小[25]。可见对该基因的表达研究可以辅助揭示生物生理适应方面的机制。
刺参作为一种棘皮动物,其体腔细胞功能类似于高等动物的血细胞,涉及营养物质的运输、生理功能的调节、再生和免疫防御等多种生理功能。近年来,关于刺参体腔细胞的种类、吞噬和细胞免疫功能的研究报道较多[26],而本研究中发现,S7亚基在刺参体腔细胞中表达量最高,表明该亚基可能与体腔细胞各项生理活动所需要的转录翻译以及蛋白质水解有关。另外,S7亚基在刺参纵肌带、肠、体壁和呼吸树中均有表达。秦艳杰等[27]采用差减文库的方法发现,S7亚基以及Cdc2同源基因在刺参再生体壁中高表达量,由此推测,该亚基在刺参再生过程中的细胞增殖调控中发挥重要的作用,这可能与刺参较强的再生能力相关。
[1] Ferrell K,Wilkinson G R M,Dubiel W,et al.Rugulatory subunit interactions of the 26S proteasome,a complex problem[J].Trends in Biochemical Science,2000,25:83-89.
[2] Voges D,Zwickl P,Baumeister W.The 26S proteasome:a molecular machine designed for controlled proteolysis[J].Annual Review of Biochemistry,1999,68:1015-1068.
[3] Tanaka K,Tsurumi C.The 26S proteasome:subunits and functions[J].Molecular Biology Reports,1997,24:3-11.
[4] Horiguchi R,Yoshikuni M,Tokumoto M,et al.Identification of a protein kinase which phosphorylate α4 subunit of the 26S proteasome in goldfish oocytes[J].Fish Physiology and Biochemistry,2003,29:255-256.
[5] 姜铁民,张勇,黄洁,等.合浦珠母贝26S蛋白酶体S4,S7亚基基因片段的分离与分析[J].海洋科学,2003,27(4):28-34.
[6] García-Arrarás J E,Valentín-Tirado G,Flores J E,et al.Cell dedifferentiation and epithelial to mesenchymal transition during intestinal regeneration in H.glaberrima[J].BMC Developmental Biology,2011,11:61.
[7] Yuan Z,Dahms H U,Han L L,et al.Cloning and characterization of a trypsin-like serine protease gene,a noval regeneration-related gene from Apostichopus japonicus[J].Gene,2012,502:46-52.
[8] 李霞,周海燕,秦艳杰,等.仿刺参体壁再生形态学和组织学的研究[J].大连海洋大学学报,2011,26(4):287-290.
[9] 王霞,李霞.仿刺参消化道的再生形态学与组织学[J].大连水产学院学报,2007,22(5):340-346.
[10] 周遵春,赫崇波,杨爱馥,等.仿刺参体壁、肠和呼吸树全长cDNA文库的构建及部分 ESTs初步分析[J].水产科学,2009,28(2):55-58.
[11] Sun L,Chen M Y,Yang H S,et al.Large scale gene expression profiling during intestine and body wall regeneration in the sea cucumber Apostichopus japonicus[J].Comparative Biochemistry and Physiology,Part D,2011,6:195-205.
[12] 李霞,王雪,秦艳杰,等.仿刺参EGFR基因的克隆与表达分析[J].水产学报,2012,36(1):41-49.
[13] Craiu A,Gaczynska M,Akopian T,et al.Lactacystin and clastolactacystin beta-lactone modify multiple proteasome beta-subunits and inhibit intracellular protein degradation and major histocompatibility complex class I antigen presentation[J].Journal of Biological Chemistry,1997,272(20):13437-13445.
[14] Glickman M H,Ciechanover A.The ubiquitin-proteasome proteolytic pathway:destruction for the sake of construction[J].Physiological Reviews,2002,82:373-428.
[15] Hershko A,Ciechanover A.The ubiquitin system[J].Annual Review of Biochemistry,1998,67:425-479.
[16] King R W,Deshaies R J,Peters J M,et al.How proteolysis drives the cell cycle[J].Science,1996,274:1652-1659.
[17] Gorbea C,Taillandier D,Rechsteiner M.Mapping subunit contracts in the regulatory complex of the 26S proteasome[J].The Journal of Biological Chemistry,2000,275(2):875-882.
[18] Allen J,Colleaux L,Davidson D,et al.Expression and mapping of the mouse S7/Pmsc2 gene,homolog of essential mitotic gene in yeast[J].Mammalian Genome,1997,8:352-354.
[19] Ito N,Tomizawa K I,Tanaka K,et al.Characterization of 26S proteasome α- and β - type and ATPase subunits from spinach and their expression during early stages of seeding development[J].Plant Molecular Biology,1997,34:307-316.
[20] Combaret L,Ralliére C,Taillandier D,et al.Manipulation of the ubiquitin-proteasome pathway in cachexia:pentoxifylline suppresses the activation of 20S and 26S proteasomes in muscles from tumor-bearing rats[J].Molecular Biology Reports,1999,26:95-101.
[21] Attaix D,Taillandier D,Combaret L,et al.Expression of subunits of the 19S complex and of the PA28 activator in rat skeletal mus-cle[J].Molecular Biology Reports,1997,24:95-98.
[22] Place S P,O'Donnell M J,Hofmann G E.Gene expression in the intertidal mussel Mytilus californianus:physiological response to environmental factors on a biogeographic scale[J].Marine Ecology Progress Series,2008,356:1-14.
[23] Teranishi K S,Stillman J H A.cDNA microarray analysis of the response to heat stress in hepatopancreas tissue of the porcelain crab Petrolisthes cinctipes[J].Comparative Biochemistry and Physiology,Part D,2007,2:53-62.
[24] Buckley B A,Gracey A Y,Somero G N.The cellular response to heat stress in the goby Gillichthys mirabilis:a cDNA microarray and protein-level analysis[J].Journal of Experimental Biology,2006,209:2660-2677.
[25] Podrabsky J E,Somero G N.Changes in gene expression associated with acclimation to constant temperatures and fluctuating daily temperatures in an annual killifish Austrofundulus limnaeus[J].Journal of Experimental Biology,2004,207:2237-2254.
[26] 李华,陈静,李强,等.仿刺参体腔细胞吞噬与凝集功能及其与温度的关系[J].大连水产学院学报,2009,24(3):189-194.
[27] 秦艳杰,李霞,张慧敏,等.仿刺参体壁创伤修复消减文库的构建与分析[J].大连海洋大学学报,2013,28(3):224-229.
Molecular cloning and tissue expression analysis of S7 subunit gene of 26S proteasome in sea cucumber Apostichopus japonicus
ZHAO Xiang-ji1,3,QIN Yan-jie1,2,LI Xia1,LIU Yang1,DING Jian-feng1
(1.Key Laboratory of Marine Bio-resources Restoration and Habitat Reparation in Liaoning Province,Dalian Ocean University,Dalian 116023,China;2.Key Laboratory of Mariculture& Stock Enhancement in North China's Sea,Ministry of Agriculture,Dalian Ocean University,Dalian 116023,China;3.Poyang Senior Middle School,Shangrao 333100,China)
In this study,S7 subunit gene in 26S proteasome was cloned from coelomocytes in sea cucumberApostichopus japonicususing RACE method for the first time.It was found that the cDNA of the gene had a full length of 2 445 bases in all,including 70 bp of the 5'UTR,1 070 bp of 3'UTR,and open reading frame of 1 305 bp encoding 435 amino acids.The S7 subunit was shown to have typical sequences of ATP -binding motif GPPGTGKT,ATP hydrolytic region DEID and RNA/DNA helicase region MAT and VRPGRLDR,sharing 95%amino acid identity with those in sea urchin,88%-89%with vertebrate,arthropodand and nematode,73%-85%with yeast andArabidopsis thaliana.There was not any signal peptides and trans-membrane domain in the protein coded by S7 gene.The real-time PCR quantification revealed that the expression of S7 subunit gene was observed in 5 tissues of sea cucumber,the maximal level in the coelomocytes,followed by longitudinal muscle and intestine,and the minimum in body wall and respiratory tree.The findings provide the molecular bases for understanding of the function of 26S proteasome in physiological regulation,regeneration and immune mechanisms in sea cucumber.
Apostichopus japonicus;S7 subunit of 26S proteasome;molecular clone;tissue expression
S917.4
A
2095-1388(2013)06-0528-07
2013-03-28
国家自然科学基金资助项目 (30371099);辽宁省教育厅实验室专项 (2008S064)
赵祥吉 (1986-),男,硕士研究生。E-mail:30965469@qq.com
秦艳杰 (1977-),女,副教授,博士。E-mail:qinyanjie@dlou.edu.cn

