共有序列简并杂合寡核苷酸引物聚合酶链反应在呼吸道副黏病毒科病毒诊断中的应用
赵百慧,王春,沈佳仁,俞雪莲,高烨,滕峥,朱兆奎,储维,宋黎黎 ,张泓,张曦
1. 上海市疾病预防控制中心,上海 200336; 2. 上海交通大学附属上海市儿童医院,上海 200040; 3. 上海市黄浦区疾病预防控制中心,上海 200023
由病毒引起的呼吸道感染是全球范围内导致人类发病和死亡的主要原因之一,对呼吸道病毒的早期、快速诊断一直是临床和公共卫生领域的研究热点[1]。在过去40年中,新发病原体已超过50种,且多数为病毒[2,3]。新型呼吸道病毒以其不确定性、突然性、难以控制性及易在人群中引起暴发流行等特点严重危害人类健康,对社会经济造成巨大损失。这就要求呼吸道病毒检测技术不仅要快速、准确,同时还要向高通量和可覆盖潜在新型呼吸道病毒的方向发展。
共有序列简并杂合寡核苷酸引物聚合酶链反应(consensus-degenerate hybrid oligonucleotide primer polymerase chain reaction,CODEHOP PCR)是1998年研发的一种新实验技术,主要用于寻找具有相同功能的蛋白质家族新成员[4-6]。该方法巧妙解决了在未知核酸序列情况下无法设计引物的问题,近5年来在病毒领域的应用主要集中在乳头瘤病毒、单纯疱疹病毒和肠道病毒[7-9]。美国疾病预防控制中心(Centers for Disease Control and Prevention,CDC)童苏香等利用该技术建立了一套检测副黏病毒科新病毒的PCR体系,并用相关病毒株进行了灵敏度和特异度验证[10]。本研究分别应用CODEHOP PCR体系和商品化试剂盒对572份儿童下呼吸道分泌物标本进行检测,比较2种方法的检测符合率;对10倍倍比稀释的呼吸道合胞病毒A(respiratory syncytial virus A,RSVA)细胞收获液检测,比较2种方法的检测灵敏度,从而综合探讨CODEHOP PCR在副黏病毒检测中的临床应用价值。
1 材料与方法
1.1 标本采集
选择2011年2~12月上海交通大学附属上海市儿童医院因急性呼吸道感染住院的患儿572例,入院3 d内以负压吸取方法获得下呼吸道分泌物,放入MEM转运培养液(含1 mg/ml庆大霉素、1 000 U/ml青霉素、2.5 g/ml两性霉素B和2%胎牛血清),暂存于4 ℃,48 h冷链转运至实验室。
1.2 核酸抽提和反转录
使用罗氏核酸自动抽提仪Roche MagNA Pure LC 2.0抽提标本RNA,200 μl标本的洗脱体积设置为100 μl。抽提好的核酸置-70 ℃保存备用。应用ReverAid First Strand cDNA Synthesis Kit(Fermentas)进行反转录,体系为20 μl,包括4 μl 5×缓冲液、2 μl dNTP(10 mmol/L)、1 μl RNA酶抑制剂(20 μg/μl)、1 μl RNA反转录酶、1 μl随机引物(0.2 μg/μl)、3 μl焦碳酸二乙酯(diethypyrocarbonate,DEPC)处理水和8 μl抽提核酸。反应参数:80 ℃ 3 min;37 ℃ 90 min;94 ℃ 2 min。反转录后cDNA置于-20 ℃保存备用。
1.3 CODEHOP PCR检测副黏病毒
将文献中的引物序列送至上海博尚生物技术有限公司合成,该系列引物均为半套式引物,引物名称和对应的副黏病毒属见表1[10]。引物合成后用DEPC处理水分别稀释至50 μmol/L、20 μmol/L备用。使用SuperScript Ⅲ One-Step RT-PCR with Platium Taq(Invitrogen)进行第1轮反转录PCR(reverse transcriptase-PCR,RT-PCR)扩增,引物为50 μmol/L Prime-F1、50 μmol/L Prime-R,反应体系和反应条件与文献报道相同。第2轮PCR扩增使用Premix Ex Taq Version 2.0(TaKaRa),反应体系为50 μl,包括25 μl Premix Ex Taq、1 μl 20 μmol/L Primer-F2、1 μl 20 μmol/L Primer-R、5 μl第1轮扩增产物、8 μl DEPC处理水,反应条件与文献相同。扩增产物进行2%琼脂糖凝胶电泳。将出现目的条带的阳性PCR产物送上海博尚生物技术有限公司测序,测序结果用GenBank Blastn进行比对,>95%同源性确定为该副黏病毒属。
表1检测副黏病毒科病毒的CODEHOPPCR体系引物(半套式)
Tab.1PrimersusedinCODEHOPPCRfordetectionofParamyxoviridae
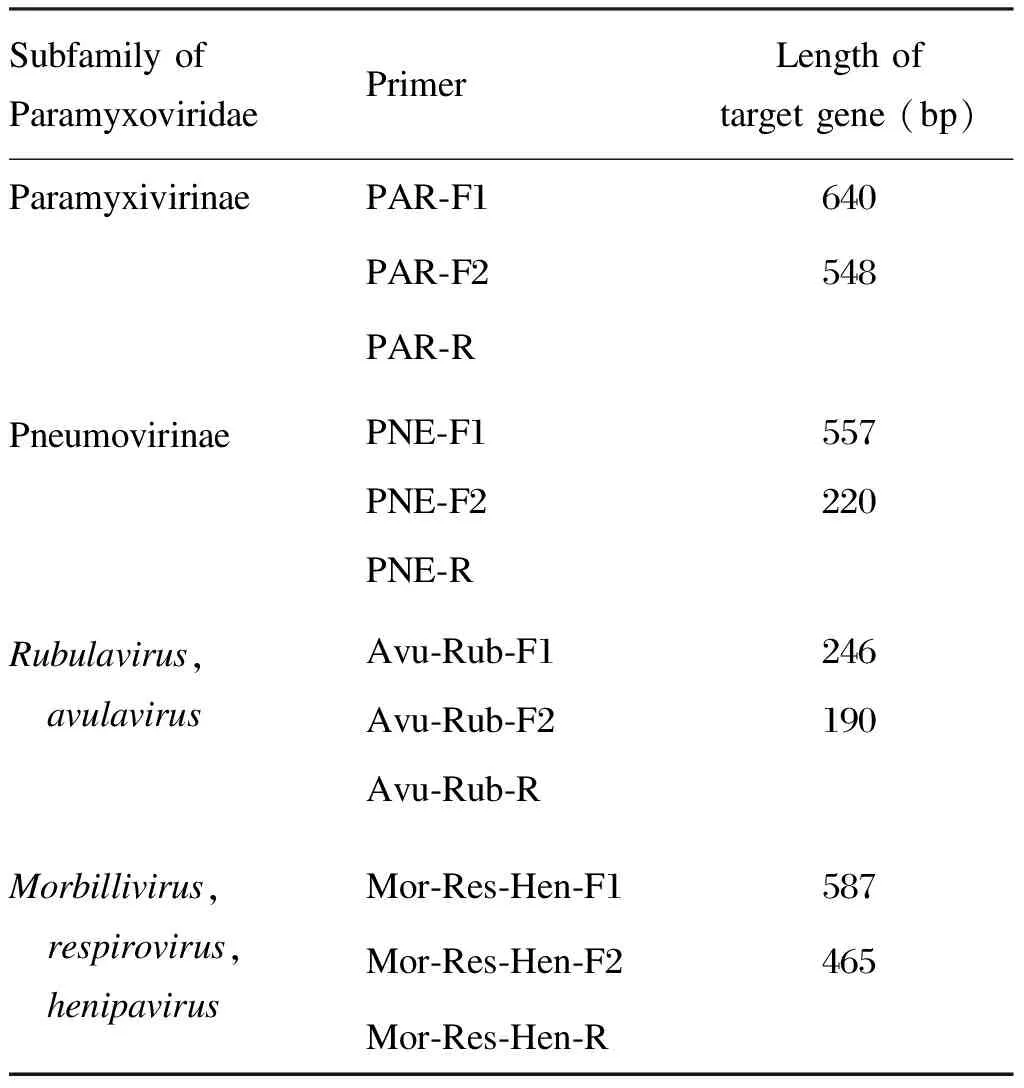
Subfamily of ParamyxoviridaePrimerLength of target gene (bp)ParamyxivirinaePAR-F1640PAR-F2548PAR-RPneumovirinaePNE-F1557PNE-F2220PNE-RRubulavirus, avulavirusAvu-Rub-F1246Avu-Rub-F2190Avu-Rub-RMorbillivirus, respirovirus, henipavirusMor-Res-Hen-F1587Mor-Res-Hen-F2465Mor-Res-Hen-R
1.4 RV12试剂盒检测副黏病毒
采用Seegene®RV12 ACE Detection试剂盒(杭州纽罗西敏生物科技有限公司)对反转录后cDNA进行扩增。该商品化试剂盒是多重PCR试剂盒,可检测常见的12种呼吸道致病原,包括副黏病毒科的RSVA、RSVB,副流感病毒1型(parainfluenza virus type 1,PIV-1)、PIV-2、PIV-3,以及人偏肺病毒(human metapneumovirus,HMPV)。该试剂盒将同一标本分成A管和B管进行扩增,A管可扩增8种常见呼吸道致病原,包括HMPV、PIV-1、PIV-2、PIV-3等;B管可扩增7种常见呼吸道致病原,包括RSVA、RSVB等。A、B管反应体系相同,20 μl反应体系包括10 μl 2×Multiple Master Mix、3 μl 8-MOP solution、4 μl 5×RV15 RACE PM(A或B)和3 μl反转录后cDNA。反应参数:94 ℃ 预变性15 min;94 ℃变性 30 s,60 ℃复性1.5 min,72 ℃延伸1.5 min,40个循环;72 ℃延伸10 min。扩增产物进行2%琼脂糖凝胶电泳,根据阳性对照和试剂盒内分子量标记判断致病原种类。
1.5 CODEHOP PCR与RV12试剂盒检测的符合率及敏感度比较
以RV12试剂盒为参考,分析CODEHOP PCR 对572份临床标本中副黏病毒亚科和肺炎病毒亚科的检测结果,计算该方法的敏感度、特异度和总符合率,计算方法见表2。将细胞培养获得的RSVA阳性细胞收获液以10倍倍比稀释,稀释度为10-1~10-11。取上述稀释液200 μl,用Roche MagNa Pure LC 2.0抽提核酸,方法同上。将抽提的核酸分别用肺炎病毒亚科CODEHOP PCR和RV12试剂盒A管进行检测,扩增产物用QIAxcel BioCalculator System毛细管电泳仪(Qiagen)电泳,卡夹选择AM420,比较2种方法的检出下限。
表2CODEHOPPCR与RV12试剂盒检测副黏病毒结果的比较
Tab.2TheresultsofCODEHOPPCRandRV12kitfordetectionof572clinicalspecimens

RV12(Reference kit)ParamyxovirinaePneumovirinaeNegative§TotalCODEHOP PCRParamyxovirinae11 (A)/10 (B)21Pneumovirinae/56 (C)37 (D)93Negative†20 (E)17 (F)424 (G)461Total3173471575∗
*indicates one co-infection sample with RSVA/PIV-3 by CODEHOP PCR and two co-infection samples with RSVA/PIV-3 and RSVA/PIV-1 by RV12 kit. The number of samples with RV12-negative and CODEHOP PCR-positive was marked by §. The number of samples with CODEHOP PCR-negative and RV12-positive was marked by †. Sensitivity of CODEHOP PCR in detection of Paramyxivirinae=A/(A+E)×100%. Sensitivity of CODEHOP PCR in detection of Pneumovirinae = C/(C+F)×100%. Specificity of CODEHOP PCR=G/(B+D+G)×100%. General coincidence rate=(A+C+G)/(A+B+C+D+E+F+G)×100%.
1.6 对2种方法检测结果不一致标本的进一步检测
1.6.1实时荧光PCR检测选用上海之江生物科技有限公司的呼吸道合胞病毒核酸测定试剂盒, PIV-1、PIV-2和PIV-3型核酸测定试剂盒及HMPV核酸测定试剂盒对上述2种方法检测结果不统一的标本作进一步分析。考虑到CODEHOP PCR结果判断需依据测序,而测序是目前分子生物学技术的“金标准”,故对CODEHOP PCR检测阳性、RV12试剂盒检测阴性的标本没有进一步分析。只选择RV12试剂盒检测阳性、CODEHOP PCR检测阴性的标本(肺炎病毒亚科17份、副黏病毒亚科18份)进行荧光PCR检测。反应参数和反应条件严格按照试剂盒操作说明,在每个反应的同时加入试剂盒含有的阴、阳性对照。PCR反应在Roche LightCycler 480荧光PCR仪上进行,依操作说明推荐的循环阈值(cycle threshold, Ct)对结果进行判断。
1.6.2副黏病毒亚科病毒属的2组半套式CODEHOPPCR检测考虑到副黏病毒亚科有5个可引起动物和人类致病的病毒属,且近几年报道的新感染人类的副黏病毒多来自这5个病毒属,为降低半套式PCR的假阴性率,进一步对这5个病毒属设计引物并进行分组扩增。其中一组为针对德国麻疹病毒属和禽副黏病毒属(Avu-Rub)的半套式PCR引物Avu-Rub-F1、Avu-Rub-F2和Avu-Rub-R,另一组为针对麻疹病毒属、呼吸道病毒属和亨尼巴病毒属(Mor-Res-Hen)的半套式引物Mor-Res-Hen-F1、Mor-Res-Hen-F2和Mor-Res-Hen-R(表1)。选取RV12试剂盒检测阳性、副黏病毒亚科CODEHOP PCR检测阴性的标本再分析,PCR反应体系和反应参数见参考文献[10],产物进行2%琼脂糖凝胶电泳。将出现目的条带的阳性PCR产物送上海博尚生物科技有限公司测序,测序结果用GenBank Blastn比对,>95%同源性确定为该副黏病毒属。
2 结果
2.1 CODEHOP PCR检测572份临床标本的结果
572份临床标本中,113份检测出副黏病毒科病毒,阳性率为19.76%,其中1例为RSVA/PIV-3混合感染。副黏病毒亚科阳性标本21份(3份PIV-1、1份PIV-2、17份PIV-3),肺炎病毒亚科阳性标本93份(16份HMPV、66份RSVA、11份RSVB)。PCR扩增产物的电泳结果见图1。
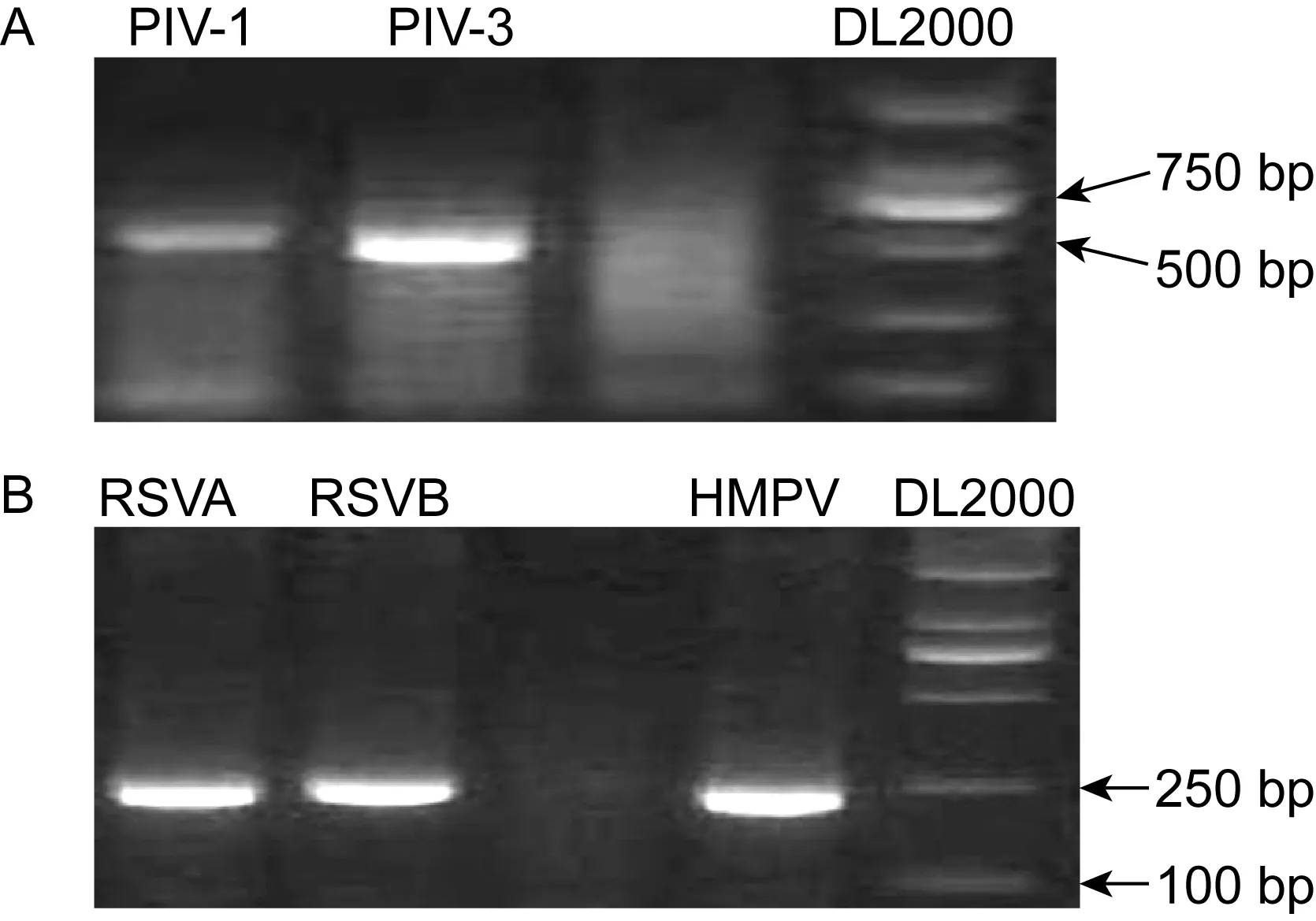
图1采用CODEHOPPCR对572份临床标本抽提核酸的检测结果
Fig.1AmplificationofRNAsfrom572clinicalspecimensbyusingsubfamilyParamyxivirinaesemi-nestedPCR(A)andsubfamilyPneumovirinaesemi-nestedPCR(B)
2.2 RV12试剂盒检测572份临床标本的结果
572份临床标本中,102份检测出副黏病毒科病毒,阳性率为17.83%,其中副黏亚科病毒31份(13份PIV-1、1份PIV-2、17份PIV-3),肺炎亚科病毒73份(3份HMPV、59份RSVA、12份RSVB,其中1份为RSVA/RSVB混合感染)。副黏亚科病毒和肺炎亚科病毒同时阳性标本2份,分别是RSVA/PIV-3和RSVA/PIV-1。572份临床标本中,63份检测出其他常见呼吸道病毒,副黏病毒科病毒阳性标本占总阳性标本的61.82%(102/165)。PCR扩增条带电泳结果见图2。
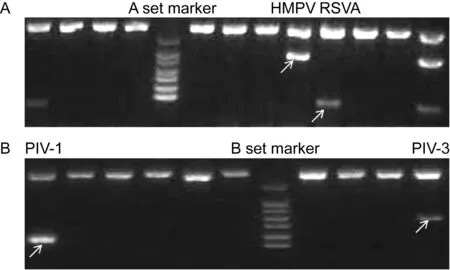
The virus types of the positive samples were identified by the size of the amplicons according to the marker. The targeted PCR products were marked with white arrow.
图2采用RV12试剂盒对572份临床标本抽提核酸的检测结果
Fig.2AmplificationofRNAsfrom572clinicalspecimensbyAset(A)andBset(B)ofRV12kit
2.3 CODEHOP PCR与RV12试剂盒的符合率和敏感度
以RV12试剂盒为参考,计算CODEHOP PCR检测的敏感度、特异度及与RV12试剂盒相比的总符合率。计算公式如下:敏感度=真阳性/(真阳性+假阴性)×100%,特异度=真阴性/(真阴性+肺炎病毒亚科真阴性+副黏病毒亚科真阴性)×100%,总符合率=(真阳性+真阴性)/标本总数×100%。572份临床标本中,副黏病毒亚科的敏感度为35.48%,肺炎病毒亚科的敏感度为76.71%,特异度为90.02%,总符合率为85.39%(表2)。对10-1~10-11倍比稀释的RSVA细胞收获液检测结果显示,肺炎病毒亚科的CODEHOP PCR检测下限为10-8,RV12试剂盒为10-6,电泳结果见图3。
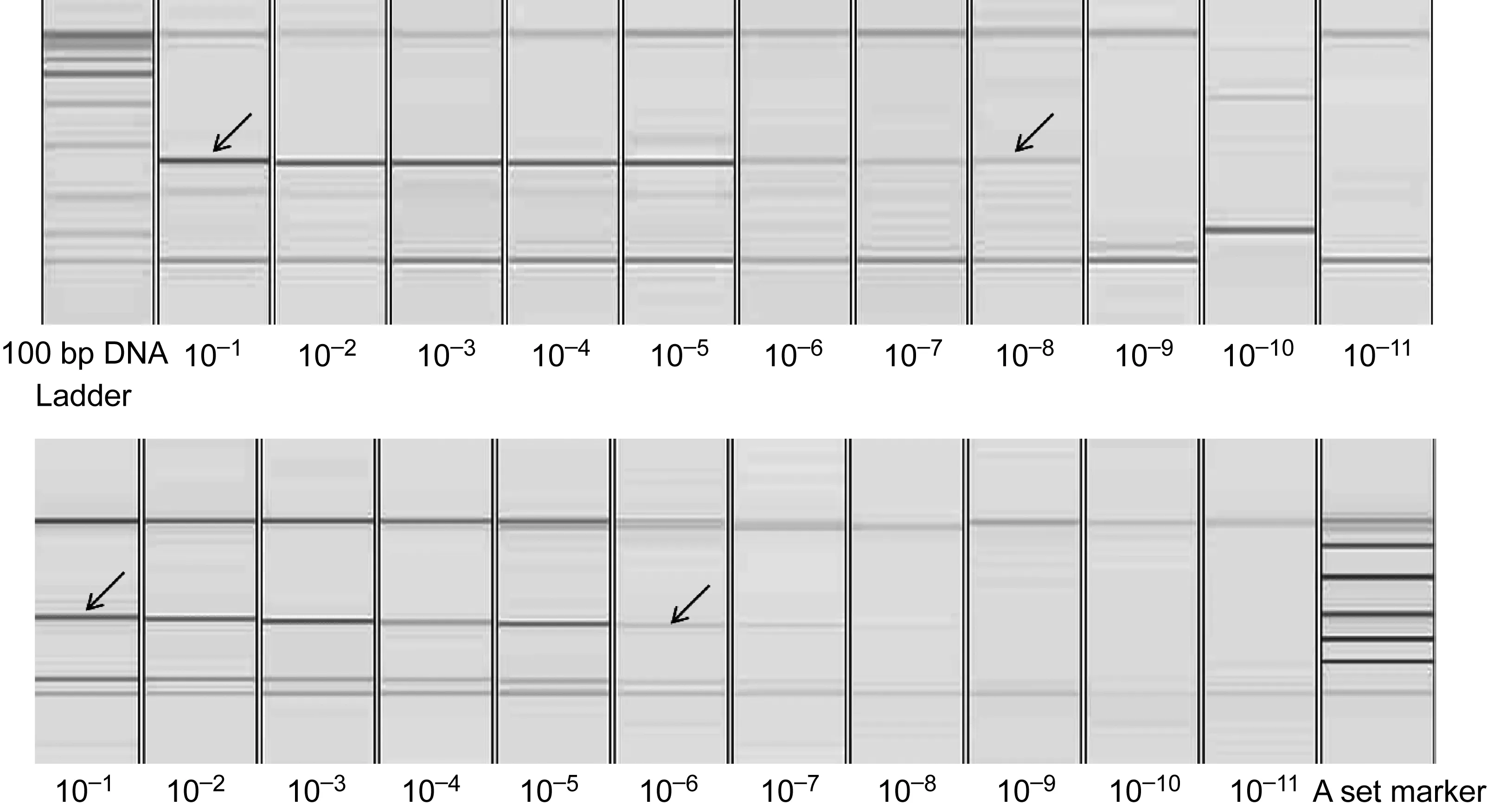
The target products were marked with black arrow.
图3CODEHOPPCR和RV12试剂盒对10倍倍比稀释的RSVA感染细胞收获液的检测结果
Fig.3AmplificationofRNAsfrom10-foldserialdilutionofRSVA-infectedcellculturebyCODEHOPPCR(A)andRV12kit(B)
2.4 初始检测结果不一致标本的进一步判断
2.4.1商品化荧光PCR试剂盒检测结果对17份肺炎病毒亚科CODEHOP PCR检测阴性、RV12试剂盒检测阳性的标本,进行荧光PCR检测,结果10份阳性,包括5份RSVA、4份RSVB、1份HMPV。对18份副黏病毒亚科CODEHOP PCR检测结果阴性、RV12试剂盒检测阳性标本,进行荧光PCR检测,结果13份阳性,包括10份PIV-3、2份PIV-1和1份PIV-2。
2.4.2副黏病毒亚科病毒属的2组半套式CODEHOPPCR结果对18份副黏病毒亚科RV12试剂盒检测阳性、CODEHOP PCR检测阴性的标本,采用副黏病毒亚科的属检测引物,检测出3份阳性。其中Avu-Rub引物套检测出1份阳性,经测序证实为PIV-2;Mor-Res-Hen引物套检测出2份阳性,经测序证实均为PIV-3。PCR产物电泳结果见图4。
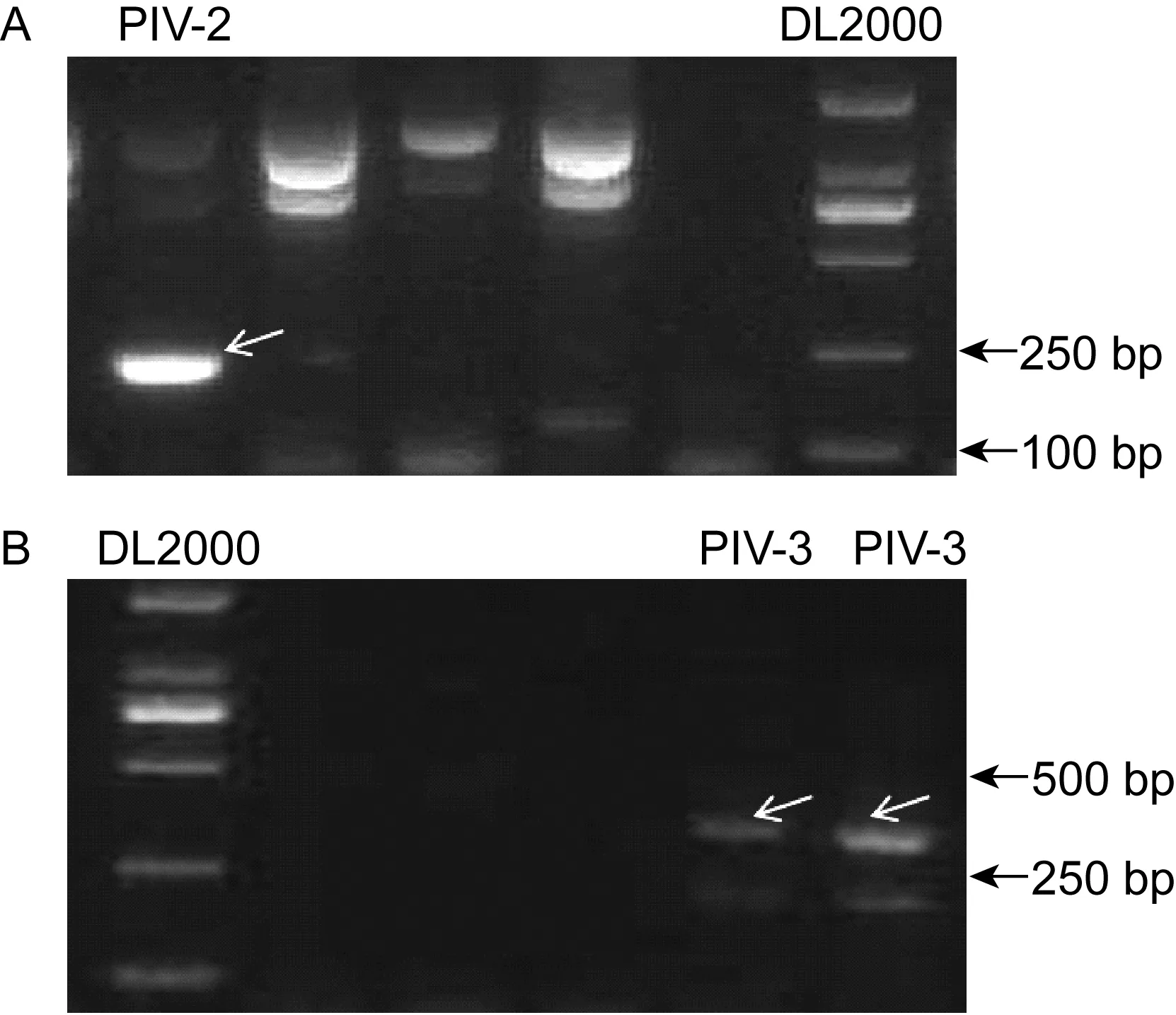
图4CODEHOPPCR对18份副黏病毒亚科CODEHOPPCR检测阴性、RV12试剂盒检测阳性标本的检测结果
Fig.4AmplificationofRNAsfrom18sampleswhichwereParamyxivirinaeCODEHOPPCR-negativebutRV12-positivebyAvu-Rub-F1,Avu-Rub-F2,Avu-Rub-Rprimers(A)andMor-Res-Hen-F1,Mor-Res-Hen-F2,Mor-Res-Hen-Rprimers(B)
3 讨论
副黏病毒科病毒是导致儿童急性下呼吸道感染的主要致病原。2009年以来对上海市儿童下呼吸道疾病谱的研究数据表明,副黏病毒科病毒在5岁以下儿童呼吸道疾病谱中占60%以上[11,12]。最近文献报道,感染人类的Nipah病毒和Hendra病毒也属副黏病毒科,提示在呼吸道感染中副黏病毒科家族的重要性越来越大。副黏病毒科分2个亚科7个病毒属,病毒种类繁多,覆盖的宿主范围广,对该类病毒的诊断方法要求同时具备高通量和可覆盖新的未知病毒的特点[13]。
尽管目前高通量分子诊断试剂已日趋成熟,但检测未知新病毒的手段还不尽如人意。新病毒因其未知核酸序列的存在而成为分子诊断技术的瓶颈。目前用于检测未知新病毒的方法,如随机引物库筛选和高通量测序技术,因工作量大、需特殊仪器设备而不易在基层实验室推广[14-16]。美国CDC在2008年建立的CODEHOP PCR方法,使用普通PCR仪,实验操作简单,是适用于CDC和临床检验科的具备高通量、可筛选潜在新病毒的方法。本研究对文献中的反应体系略作调整,并应用CODEHOP PCR和目前检测呼吸道病毒的多重商品化RV12试剂盒对来自上海市儿童医院的572份下呼吸道吸出物进行检测。其中CODEHOP PCR检测副黏病毒亚科的灵敏度只有35.48%,这与文献报道的使用副黏病毒亚科病毒株确定的敏感度有很大差异。CODEHOP PCR方法为半套式PCR,RV12试剂盒为RT-PCR,理论上讲CODEHOP PCR灵敏度应高于RV12试剂盒。造成这一现象的主要原因是由于该灵敏度是以RV12试剂盒为基准计算得到的,RV12检测到副黏病毒亚科阳性标本29份,CODEHOP PCR检测到阳性标本21份,相差不大,但2种方法同时检测为阳性的标本只有11份。如果以其中一种方法作为基准,那么另一种方法的假阴性比例大,造成灵敏度较低。为验证这一推测,本研究首先以CODEHOP PCR为基准,计算出RV12试剂盒检测副黏病毒亚科的灵敏度为52.38%,远低于试剂盒研发要求的>95%。其次,对RSVA感染的细胞收获液10倍倍比稀释的核酸进行检测,结果显示CODEHOP PCR检测下限为10-8,RV12试剂盒检测下限为10-6,这也证实CODEHOP PCR检测灵敏度低主要是因为与RV12试剂盒结果的符合率较低。
CODEHOP PCR与RV12试剂盒检测的总符合率为85.39%,其中CODEHOP PCR检测阳性、RV12试剂盒检测阴性的47份标本(副黏病毒亚科10份、肺炎病毒亚科37份)经测序确定为真阳性,而RV12试剂盒检测阳性、CODEHOP PCR检测阴性的35份标本(副黏病毒亚科18份、肺炎病毒亚科17份)因无法获得RV12试剂盒的引物序列故没有进行测序。为进一步明确这35份标本是否为真阳性,选择商品化荧光试剂盒进行检测。结果显示,副黏病毒亚科18份标本中13份阳性,肺炎病毒亚科17份标本中10份阳性。造成这一结果的原因如下:其一,CODEHOP PCR使用的引物为简并引物,存在扩增过程中与靶片段特异性相结合的引物会在达到PCR扩增平台期前发生耗竭的可能;其二,从电泳图谱中可看出,CODEHOP PCR针对临床标本的检测在出现目的条带的同时常伴随非特异性扩增,这会影响实验阳性结果。为排除因副黏病毒亚科病毒属过多而造成阳性率降低的可能,进一步对副黏病毒亚科CODEHOP PCR检测阴性、RV12试剂盒检测阳性的18份标本进行2组属引物扩增。在18份标本中,仅扩增出3份阳性,说明副黏病毒亚科病毒引物覆盖面已足够广。在对临床标本检测过程中,可先选择针对亚科的PCR体系,如果某一亚科检测阳性率过低,再进一步选择属PCR体系。CODEHOP PCR、RV12试剂盒与上海之江生物科技有限公司的荧光PCR试剂盒存在不同的适用性。前者是基于高度简并设计的引物,的确存在非特异性扩增和引物非特异性耗竭的现象,因此在日常临床标本检测中的优势不明显,或需进一步优化。但该方法的优势在于有发现新致病原的能力,在其他报道中已显示,因此CODEHOP PCR更适于在新发、突发传染病疫情暴发时使用,且建议与其他方法组合应用以达到鉴定新病原体的目的。
本研究在572份临床标本中并未检测到新病毒,主要系临床标本数量较少所致。CODEHOP PCR与RV12试剂盒使用的仪器都是普通PCR扩增仪,实验所需时间和操作复杂性相当,但CODEHOP PCR成本要低于RV12试剂盒。本研究表明,CODEHOP PCR技术可同时满足高通量和对未知新病原体筛查的要求,该方法通过测序确定病原体种类,融合了PCR的易操作性与测序的精准性。尽管目前该方法在临床标本检测中的灵敏度不是很高,但可进一步与目前一些快速高通量的分子生物学诊断手段,如基因芯片、多重实时荧光定量PCR等技术相结合,值得进一步优化并在病毒检测领域推广。另外,该方法的核心点是引物设计,目前已有美国癌症国立研究院在线软件iCODEHOP辅助对该引物的设计(http://dbmi-icode-01.dbmi.pitt.edu/i-codehop-context/Welcome)[6],使其推广成为可能。童苏香等利用该原理建立了检测正黏病毒科的PCR体系,在来源于蝙蝠的标本中发现了疑似流感病毒新亚型H17,进一步验证了其在发现呼吸道新病毒领域中的应用价值[17]。
致谢
感谢美国CDC童苏香教授在该研究中对实验环节的指导。
[1] Liu L, Johnson HL, Cousens S, Perin J, Scott S, Lawn JE, Rudan I, Campbell H, Cibulskis R, Li M, Mathers C, Black RE, Child Health Epidemiology Reference Group of WHO and UNICEF. Global, regional, and national causes of child mortality: an updated systematic analysis for 2010 with time trends since 2000 [J]. Lancet, 2012, 379(9832): 2151-2161.
[2] 张欣欣, 孔晓慧.新发现的呼吸道病毒研究现状[J].中华儿科杂志, 2010, 48(02):112-114.
[3] Jartti T, Jartti L, Ruuskanen O, Soderlund-Venermo M. New respiratory viral infections [J]. Curr Opin Pulm Med, 2012, 18(3): 271-278.
[4] Rose TM, Henikoff JG, Henikoff S. CODEHOP (consensus-degenerate hybrid oligonucleotide primer) PCR primer design [J]. Nucleic Acids Res, 2003, 31(13): 3763-3766.
[5] Preston GM. Polymerase chain reaction with degenerate oligonucleotide primers to clone gene family members [J]. Methods Mol Biol, 1996, 58: 303-312.
[6] Boyce R, Chilana P, Rose TM. iCODEHOP: a new interactive program for designing consensus-degenerate hybrid oligonucleotide primers from multiply aligned protein sequences [J]. Nucleic Acids Res, 2009, 37(Web Server issue): W222-W228.
[7] Staheli JP, Ryan JT, Bruce AG, Boyce R, Rose TM. Consensus-degenerate hybrid oligonucleotide primers (CODEHOPs) for the detection of novel viruses in non-human primates [J]. Methods, 2009, 49(1): 32-41.
[8] Zlateva KT, Crusio KM, Leontovich AM, Lauber C, Claas E, Kravchenko AA, Spaan WJ, Gorbalenya AE. Design and validation of consensus-degenerate hybrid oligonucleotide primers for broad and sensitive detection of corona- and toroviruses [J]. J Virol Methods, 2011, 177(2): 174-183.
[9] Rose TM. CODEHOP-mediated PCR — a powerful technique for the identification and characterization of viral genomes [J].Virol J, 2005, 2: 20.
[10] Tong S, Chern SW, Li Y, Pallansch MA, Anderson LJ. Sensitive and broadly reactive reverse transcription-PCR assays to detect novel paramyxoviruses [J]. J Clin Microbiol, 2008,46(8): 2652-2658.
[11] 何静, 龚燕, 张万菊, 徐磊, 刘炜, 钱方兴, 揭志军, 俞慧菊, 李扬, 周志统, 胡芸文. 2009~2010年上海地区急性呼吸道感染病毒病原谱分析[J]. 微生物与感染, 2011, 6(2):90-96.
[12] 曾玫, 王晓红, 俞蕙, 朱启镕. 上海地区儿童急性呼吸道病毒感染的流行特征[J]. 中华传染病杂志, 2008, 26(9):527-532.
[13] Pomeroy LW, Bjørnstad ON, Holmes EC. The evolutionary and epidemiological dynamics of the paramyxoviridae [J]. J Mol Evol, 2008, 66(2): 98-106.
[14] Jartti T, Hedman K, Jartti L, Ruuskanen O, Allander T, Söderlund-Venermo M. Human Bocavirus — the first 5 years [J]. Rev Med Virol, 2012, 22(1): 46-64.
[15] Towner JS, Sealy TK, Khristova ML, Albario CG, Conlan S, Reeder SA, Quan PL, Lipkin WI, Downing R, Tappero JW, Okware S, Lutwama J, Bakamutumaho B, Kayiwa J, Comer JA, Rollin PE, Ksiazek TG, Nichol ST. Newly discovered Ebola virus associated with hemorrhagic fever outbreak in Uganda. [J]. PLoS Pathog, 2008, 4(11): e1000212.
[16] Palacios G, Druce J, DU L, Tran T, Birch C, Colan S, Quan PL, Hui J, Marshall J, Simons JF, Eqholmn M, Paddock CD, Shich WJ, Goldsmith CS, Zaki SR, Catton M, Lipkin WI. A new arenavirus in a cluster of fatal transplant-associated diseases [J]. N Engl J Med, 2008, 358(10): 991-998.
[17] Tong S, Li Y, Rivailler P, Conrardy C, Castillo DA, Chen LM, Recuenco S, Ellison JA, Davis CT, York IA, Turmelle AS, Moran D, Rogers S, Shi M, Tao Y, Weil MR, Tang K, Rowe LA, Sammons S, Xu X, France M, Lindblade KA, Cox NJ, Anderson LJ, Rupprecht CE, Donis RO. A distinct lineage of influenza A virus from bats [J].Proc Natl Acad Sci USA, 2012,109(11): 4269-4274.

