基于线粒体DNA D-loop序列分析养殖刀鲚与湖鲚的遗传多样性
徐钢春,魏广莲,李建林,徐跑,张呈祥,顾若波
(1.中国水产科学研究院淡水渔业研究中心,农业部淡水渔业和种质资源利用重点实验室,江苏无锡214081;2.南京农业大学无锡渔业学院,江苏无锡214081;3.江阴市水产指导站,江苏江阴214431)
刀鲚Coilia nasus隶属于鲱形目、鳀科、鲚属,俗称刀鱼,为中国长江重要的经济洄游性鱼类[1-2],其脂肪丰满、肉质细嫩鲜美,享有“长江三鲜”之一的美誉;但近年来刀鲚天然资源量急剧减少,种质小型化明显,远远不能满足市场的需求[3-4]。随着刀鲚灌江纳苗[5]、幼鱼采捕[6]的成功实施后,人工繁殖试验也取得了成功[7],刀鲚的池塘养殖正逐渐成为新兴的产业。程起群等[8]以线粒体细胞色素b基因片段序列为分子标记,分析了长江野生刀鲚和湖鲚Coilia nasus taihuensis的遗传关系,认为湖鲚是刀鲚的一个地理种群且遗传多样性水平增加,同时还认为,隔离和适应是形态差异形成的重要因素[9]。随着刀鲚人工养殖规模的扩大,很有必要对刀鲚淡水养殖群体和湖鲚的遗传多样性和遗传分化状况进行深入研究,从而促进刀鲚人工养殖的可持续发展。
在mtDNA上,D-loop区的进化速度最快,用它来进行种内、种群间的系统进化分析,是一种在分子水平上研究鱼类种群遗传多样性比较经济有效的手段[10-14]。本研究中,作者以灌江纳苗后完全在淡水池塘环境中繁育、养殖的刀鲚群体与湖泊定居的湖鲚为试验材料,运用PCR技术对D-loop区段进行扩增,旨在从分子水平上分析其遗传多样性及相互间的差异水平,为保护刀鲚种质资源和有效地开展刀鲚遗传育种研究提供参考资料。
1 材料与方法
1.1 材料
试验用养殖刀鲚11尾,为2004年灌江纳苗养殖并繁育的子三代,于2010年8月采自中国水产科学研究院长江三鲜养殖与示范基地,体质量为17.7~85.5 g,平均为36.2 g;湖鲚8尾,于2010年9月采自太湖梅梁湾段,体质量为25.7~40.4 g,平均为33.5 g。从每尾鱼剪取少许背部肌肉分别放入Eppendorf管中,于-20℃下保存。
1.2 方法
1.2.1 DNA提取 将肌肉剪碎,分别放入1.5 mL Eppendorf管中,加入470 μL SET缓冲液,再依次加入12.5 μL 20% 的 SDS 和 10 μL(10 mg/mL)蛋白酶K,混合后在55℃水浴中消化过夜,再分别用饱和酚、酚氯仿、氯仿/异戊醇抽提去除蛋白,最后用预冷的无水乙醇沉淀DNA,烘干后用TE溶解。所得基因组DNA样品用紫外分光光度计测定DNA样品的浓度和纯度,同时用琼脂糖凝胶电泳检测基因组DNA的完整性并估测分子量。将DNA原样稀释至50 ng/μL,于4℃下保存备用。
1.2.2 PCR扩增及测序 使用两个通用引物DF1和DR2扩增D-loop区段基因,引物序列DF1:5'-CTAACTCCCAAAGCTAGAATTCT-3',DR2:5'-ATCTTAGCATCTTCAGTG-3'[15]。扩增反应混合物总体积25 μL,包括 DNA 模板 1.5 μL,10×Reaction buffer 2.5 μL,200 μmol/L dNTP 0.5 μL,引物(浓度 0.2 μmol/L)各 1 μL,Taq 酶 (1 U)0.2 μL,用无菌ddH2O补至25 μL。PCR扩增程序为:94℃下预变性3 min;94℃下变性30 s,50℃下退火1 min,72℃下延伸1 min,共进行35个循环;最后在72℃下再延伸8 min。PCR产物用14 g/L琼脂糖凝胶电泳检测,并拍照记录。目标扩增产物用试剂盒回收纯化后,由上海博尚生物有限公司进行双向测序,本研究中,除用引物DF1和DR2由两端向中部测序外,还根据所得的序列再设计一对引物 (F:5'-GCGGATTCTCCGTAGACAAC-3',R:5'-GTAAAATTGTCTGGGTCTCC-3')由序列中部向两端进行测序,以增加测序的准确性。
1.3 数据处理
序列用Clustal W 1.83软件[16]分析比对。利用DnaSP 4.0软件[17]统计单倍型及变异位点(S)、计算单倍型多样性 (H)、核苷酸多样性 (π)及平均核苷酸差异数 (K)。用Mega 3.0软件[18]中的Kimura双参数法计算各单倍型间的遗传距离。采用NJ法 (Neighbor-joining)对基因序列数据进行分析,Bootstrap置信值估算重复次数为1 000次。
2 结果
2.1 线粒体D-loop区段基因扩增结果
养殖刀鲚和湖鲚的D-loop区段基因均能被清晰稳定地扩增,PCR产物电泳图谱见图1。从图1可见,D-loop区段基因长度为1 200 bp左右。PCR产物经回收后测序,测出的序列参照GenBank中已有的刀鲚D-loop区段序列 (登录号为NC_009579),经Clastal X比对分析,结果表明同源性都高达99%以上,确定所得序列为刀鲚mtDNA D-loop区序列。
养殖刀鲚和湖鲚两个群体D-loop区段基因片段中,A、T、G、C碱基含量分别为 33.3%、33.3%、14.2%、19.2%。其中,A+T碱基含量(66.6%)高于C+G的含量 (33.4%)。
图1 养殖刀鲚和湖鲚mtDNA D-loop区段PCR电泳图Fig.1 The PCR eletrophoretogram of mtDNA D-loop in the populations of farmed Coilia nasus and Coilia nasus taihuensis
2.2 遗传多样性分析
通过序列分析,养殖刀鲚和湖鲚的D-loop序列全长都有较大的变异,养殖刀鲚的D-loop序列长度为1 210~1 252 bp,若以刀鲚 (NC_009579)D-loop序列全长1 252 bp为标准,则11尾养殖刀鲚中,只有2尾鱼序列长度没有变化,其他9个个体存在1、3、38 bp 3种片段的缺失;湖鲚的D-loop序列长度为1 252~1 290 bp,8尾湖鲚中有3个个体存在38 bp片段的插入。
在两个群体19尾个体中,共检测到变异位点(S)35个,占全部序列的2.79%,其中单一多态位点14个,简约信息位点21个。绘制碱基变异位点分布图如图2所示。由图2可以看出,除了碱基在100以内的多态位点外,其他多态位点十分一致,这与D-loop区段基因具有一定的保守性和稳定性相符合。本研究中共检测到12种不同的单倍型,单倍型多态性 (H)为0.924;核苷酸多样性(π)为0.0099,平均核苷酸差异数 (K)为4.154;遗传距离为0~0.020,平均遗传距离为0.015;养殖刀鲚、湖鲚与七丝鲚 (EF_419803)的平均遗传距离分别为0.0528、0.0537,而养殖刀鲚与湖鲚的平均遗传距离为0.0148,明显小于它们与七丝鲚的平均遗传距离。养殖刀鲚和湖鲚群体内的各遗传多样性参数如表1所示。由表1可以看出,养殖刀鲚的各遗传多样性参数稍高于湖鲚,说明养殖刀鲚比湖鲚的遗传多样性要丰富。
2.3 分子系统树
以七丝鲚(EF_419803)和刀鲚的D-loop区段序列(NC_009579)为参考进行系统分析,结果显示,整个群体聚类成两大支,七丝鲚为一大支,养殖刀鲚和湖鲚聚在一起组成另一大分支。在养殖刀鲚和湖鲚聚在一起组成的分支中,养殖刀鲚和湖鲚又各自聚在一起,形成独立的单系类群(图3)。
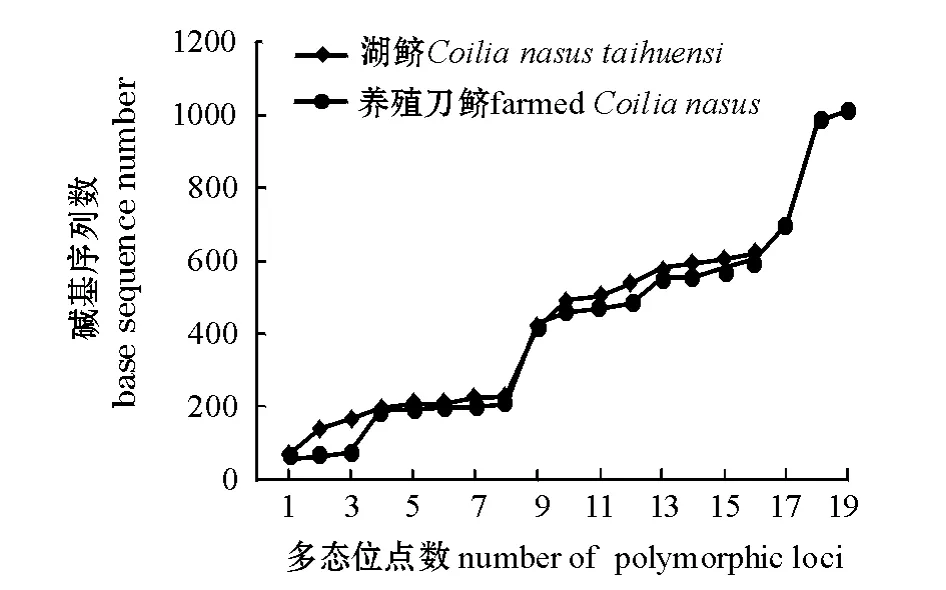
图2 刀鲚两个群体多态位点序列分布图Fig.2 The base sequence of polymorphic fragments between this two populations

表1 养殖刀鲚和湖鲚两个群体的遗传多样性Tab.1 Populational genetic diversity in farmed Coilia nasus and Coilia nasus taihuensis

图3 基于D-loop区段序列构建的几种鲚属鱼类的邻接树Fig.3 Molecular phylogenetic tree in several anchovy species based on the D-loop sequences construced by NJ method
3 讨论
3.1 刀鲚和湖鲚种群控制区序列的碱基组成
所测定的19尾刀鲚mtDNA D-loop区序列中,碱基 A、T、G、C的平均含量分别为 33.3%、33.3%、14.2%、19.2%,从碱基组成可以看出,D-loop区段表现出很强的碱基组成偏向性,即在A、T、G、C 4种碱基中,G的含量明显低于其他3种碱基的含量;此外,刀鲚mtDNA D-loop区中A+T碱基的含量 (66.6%)高于G+C碱基(33.4%),符合脊椎动物mtDNA D-loop区碱基组成的特点[19],并与刘焕章[20]、韩虎峰等[21]关于鱼类mtDNA控制区的研究结果一致,同时与鲚属其他种类一样[22],具有高比例的 A+T可能是mtDNA D-loop序列变异较快的原因之一。
3.2 遗传变异及遗传多样性
本试验中测定的养殖刀鲚和湖鲚两个群体的D-loop序列中共检测出35个变异位点,占全部序列的2.79%,共定义了12个单倍型,显示出较为丰富的遗传多样性,与程起群等[8]、葛家春等[23]和马春艳等[24]的研究结果一致。
人工养殖环境中因基础群体数量过小、养殖过程中出现的近交衰退等因素可能导致养殖群体遗传多样性降低,进而影响养殖鱼类的经济性状,最终对养殖业造成较大的危害[25-26]。本试验中养殖刀鲚各遗传多样性参数稍高于湖鲚,说明养殖刀鲚的遗传多样性比湖鲚更为丰富,这点从养殖刀鲚D-loop序列长度存在比湖鲚更多样的变化也能得到印证。湖鲚是长时期陆封型刀鲚群体[8],由于奠基者效应 (Founder effect)等因素的影响,湖鲚的遗传多样性水平可能会较低。养殖刀鲚虽然没有和外界群体发生基因交流,但可能由于其亲本是由灌江纳苗而培育繁殖的子三代,其有着较强的环境适应能力、生存能力和进化潜力,仍还保持着较高的遗传多样性。然而,在经过数代或者更长时间的人工繁育后,刀鲚种群的遗传多样性水平仍然可能因受到影响而出现衰退的现象。此外,本试验中还发现,养殖刀鲚和湖鲚两个群体的个体间D-loop序列长度的多态性,序列长度差异主要源于以38 bp为基本单位的不同次数的片段重复,唐文乔等[15]的研究也出现过相似的结果。有学者认为,这是由于某些鱼类增加遗传变异的一种方式,对维持物种的生存有一定的作用[27]。
Billington等[28]报道鱼类种内遗传距离一般在10%以内,对其他一些动物的D-loop区段基因系列分析表明,种内个体间的序列差异一般在0~4.06%,差异超过6%的个体间已有明显的亚种或者种的分化[29],而本试验中养殖刀鲚、湖鲚与七丝鲚的差异分别为5.28%和5.37%,超出了一般种内个体间的序列差异范围,但养殖刀鲚与湖鲚的平均遗传距离为1.48%,仍属于种内个体间的序列差异,未达到种或亚种的分化,这与唐文乔等[15]的研究结果相似。虽然系统树中养殖刀鲚和湖鲚各自聚在一起,形成独立的单系类群,但养殖刀鲚和湖鲚共同构成1个单系群,说明它们为两种生态型种群,并非两种有效物种,这与程起群等[8]的研究结果一致。
当然,由于野生刀鲚资源量急剧减少使得人工繁育、养殖、放流成为一种新的保护刀鲚资源的有效方法,所以,应该保持一定数量的人工繁育刀鲚亲本,使其适时与洄游刀鲚进行基因交流,以保障养殖刀鲚较高的遗传多样性,同时防止养殖群体出现种质退化等现象,科学地保护刀鲚种质资源,达到资源的可持续利用。
[1]Whitehead P J P.FAO species catalogue.Vol.7.Clupeoid fishes of the world(suborder Clupeioidei).An annotated and illustrated catalogue of the herrings,sardines,pilchards,sprats,shads,anchovies and wolf-herrings.Part 1-Chirocentridae,Clupeidae and Pristigasteridae[M].Rome:United Nations Development Programme,1985:470-471.
[2]张敏莹,徐东坡,刘凯,等.长江下游刀鲚生物学及最大持续产量研究[J].长江流域资源与环境,2005,14(6):694-698.
[3]Shi D,Gong D.Strategies of resource conservation for Coilia ectenes in the Yangtze Estuary[J].Marine Science,2003,25:96-97.
[4]Li Y X,Xie S G,Li Z J,et al.Gonad development of an anadromous fish Coilia ectenes(Engraulidae)in lower reach of Yangtze River,China[J].Fisheries Science,2007,73(6):1224-1230.
[5]张呈祥,郑金良.长江刀鲚灌江纳苗与养殖[J].科学养鱼,2006(7):26.
[6]沈林宏,戴玉红,顾树信,等.长江刀鲚幼鱼的采集与运输技术研究[J].水产养殖,2011(5):4-6.
[7]Xu G C,Tang X,Zhang C X,et al.First studies of embryonic and larval development of Coilia nasus(Engraulidae)under controlled conditions[J].Aquaculture Research,2011,42:593-601.
[8]程起群,温俊娥,王云龙.刀鲚与湖鲚线粒体细胞色素b基因片段多态性及遗传关系[J].湖泊科学,2006,18(4):425-430.
[9]程起群,韩金娣.鲚属两种群的形态变异及综合判别[J].湖泊科学,2004,16(4):356-364.
[10]肖武汉,张亚平.鱼类线粒体DNA的遗传与进化[J].水生生物学报,2000,24(4):384-391.
[11]Sugaya T,Ikeda M,Fujio Y.Comparison for the genetic variabilities of natural and seed populations of Japanese flounder based on PCR-RFLP analysis of mtDNA D-loop[J].Fish Genetics Breeding Science,1999,28:65-73.
[12]单淇,董仕,吴海防,等.3个群体鳙鱼mtDNA D-1oop区段的限制性片断长度多态性分析[J].中国水产科学,2006,13(2):174-180.
[13]谭书贞,董仕,边春媛,等.长江流域3个群体草鱼mtDNA D-loop区段的PCR-RFLP分析[J].南开大学学报:自然科学版,2007,40(3):106-112.
[14]杨正玲,赵世海,龚疏影,等.州河鲤、建鲤和框鳞镜鲤的mtDNA D-loop的 RFLP 分析[J].淡水渔业,2008,38(3):40-45.
[15]唐文乔,胡雪莲,杨金权.从线粒体控制区全序列变异看短颌鲚和湖鲚的物种有效性[J].生物多样性,2007,15(3):224-231.
[16]Thompson J D,Gibson T J,Plewwniak F,et al.The Clustal X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Research,1997,25(24):4876-4882.
[17]Rozas J,Sánchez-delbarrio J C,Messeguer X,et al.DnaSP,DNA polymerism analyses by the coalescent and other methods[J].Bioinformatics,2003,19(18):2496-2497.
[18]Kumar S,Tamura K,Nei M.MEGA3:Integrated software for molecular evolutionary genetics analysis and sequence alignment[J].Brief Bioinform,2004,5(2):150-163.
[19]Broughton R E,Milam J E,Roe B A.The complete sequence of the zebrafish(Danio rerio)mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA[J].Genome Research,2001,11(11):1958-1967.
[20]刘焕章.鱼类线粒体DNA控制区的结构与进化:以鳑鲏鱼类为例[J].自然科学进展,2002,12(3):266-270.
[21]韩虎峰,代应贵.珠江水系光唇裂腹鱼可渡河种群mtDNA D-loop序列多态性分析[J].中国水产科学,2010,17(1):143-149.
[22]诸廷俊,杨金权,唐文乔.长江口鲚属鱼类线粒体DNA控制区结构分析[J].上海水产大学学报,2008,17(2):152-157.
[23]葛家春,曹延,陈婵娟,等.利用扩增片段长度多态性技术分析长江刀鲚的遗传多样性[J].南京大学学报,2008,44(3):333-337.
[24]马春艳,刘敏,马凌波.长江口刀鲚遗传多样性扩增多态DNA(RAPD)分析[J].海洋水产研究,2004,25(5):20-24.
[25]谢振宇,杜继曾,陈学群,等.线粒体控制区在鱼类种内遗传分化中的意义[J].遗传,2006,28(3):362-368.
[26]王军,全成干,苏永全,等.宫井洋大黄鱼遗传多样性的RAPD分析[J].海洋学报,2001,23(3):87-91.
[27]Zhang S M,Deng H,Wang D Q,et al.Mitochondrial DNA length variation and heteroplasmy in Chinese sturgeon Acipenser sinensis[J].Acta Genetica Sinica,1999,26:489-496.
[28]Billington N,Hebert P D N.Mitochondrial DNA diversity in fishes and its implications for introductions[J].Canadian Journal of Fisheries Aquatic Sciences,1991,48:80-94.
[29]Yang X G,Wang Y Q,Zhou K Y.The authentication of oviductus ranae and their original animals by using molecular marker[J].Biological﹠ Pharmaceutical Bulletin,2002,25:1035-1039.

