DNA的滚环扩增技术研究进展
王晓亮,王星宇,梁长城,董 平,梁兴国
(中国海洋大学食品科学与工程学院,山东青岛266003)
DNA的滚环扩增技术研究进展
王晓亮,王星宇,梁长城,董 平,梁兴国*
(中国海洋大学食品科学与工程学院,山东青岛266003)
作为一种等温DNA扩增技术,滚环扩增(Rolling Circle Amplification,RCA),以其简单和高效的特点得到了广泛的研究和应用。一般通过与靶序列进行杂交,特异性地生成环状DNA来达到DNA检测的目的。RCA已经广泛应用于免疫检测、原位检测、SNP检测等方面。本文将对RCA的工作原理、技术进展、应用前景以及存在的问题等进行介绍。
核酸检测,恒温核酸扩增,滚环扩增,锁式探针,核酸复制
Abstract:As an isothermal DNA amplification technology,the well-known RCA (rolling circle amplification) has gained wide attention for its simplicity and high efficiency.Generally,forming specifically the circular DNA through hybridizing to the target molecule is the key point to realize specific detection.RCA has been extensively applied to immunoassay,in situ detection,SNP investigation and microarray assay.This review would introduce the principle,technological development,applicable prospect and the present problems of RCA.
Key words:nucleic acid detection;isothermal nucleic acid amplification;RCA;padlock probe;nucleic acid replication
核酸扩增是一项基本的现代生物技术,不仅在生命科学各个领域的科学研究中发挥了重要作用,并且在医疗、农业、环境监测、取证等各个方面都得到了广泛应用。众所周知,上世纪八十年代发展起来的PCR(Polymerase chain reaction),作为第一个体外核酸扩增技术,对现代分子生物学的发展起到非常重要的作用,可以说PCR技术是生物医学领域中的一项革命性创举和里程碑。但是PCR方法仍然有不足之处如:扩增过程中需要对双链DNA进行变性、需要特制的可快速升降温的PCR仪、因非特异性扩增而产生假阳性或假阴性结果等。因此,近十几年来发展起来了一些新的核酸扩增技术以取代PCR技术或弥补它的不足。如:环介导等温扩增(Loop-mediated isothermal amplification,LAMP)、滚环扩增(Rolling circle amplification,RCA)、基于核酸序列的扩增(Nucleic acid sequence-based amplification,NASBA)、链置换扩增(Strand displacement amplification,SDA)和依赖解旋酶的扩增(Helicase-dependent amplification,HAD)等。这些方法各有所长,综合利用这些技术将使便携式核酸检测装置等的开发成为可能。滚环扩增(RCA)技术以其简单和高效的特点受到了越来越多的重视。由于引物可以连接于抗体等其他生物大分子上,使滚环扩增产生的信号可固定于基质;引物也可以固定于固相基质,使其与DNA芯片技术结合并使高通量检测成为可能,例如进行微阵列上的单核苷酸多态性(Single Nucleotide Polymorphism,SNP)检测。国外关于RCA技术的综述文章多见于2006年以前,主要介绍RCA在生物技术领域的具体应用方法。最近几年,RCA已在纳米材料、生物传感器和功能性核酸的制备等领域有很多应用[1]。国内虽然也有综述文章发表,但都只是进行了简单的介绍,而且对于RCA技术存在问题的阐述也不够详细,例如高灵敏度和高特性的矛盾,要很好地解决还需巧妙的分子设计;RCA技术的广泛应用还有赖于其自身的发展以及同其他高新技术(如纳米技术,表面修饰技术,高灵敏度荧光检测技术等)的有机结合[2]。本文对RCA技术及其应用进行较全面的介绍与分析,着重介绍其存在的问题和发展前景,以期对大家开发和应用新的核酸扩增技术有所启发。
1 滚环扩增技术
1.1 滚环扩增技术简介
滚环扩增技术发展于上世纪九十年代中期,几个研究小组证实[3-5],以几十个碱基长的单链环状DNA为模板,在具有链置换性的DNA聚合酶的作用下,可以产生与环状DNA模板互补的单链DNA序列(图1a)。Lizardi等最先将结合锁式探针的滚环扩增应用于突变检测[6],发展了使用单条引物的线性RCA和使用双引物的指数RCA(图1b)。
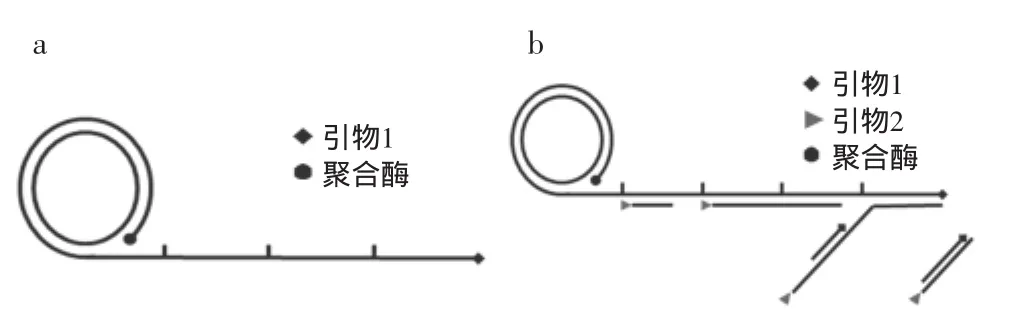
图1 RCA扩增与DNA检测原理Fig.1 Principle of RCA amplification and DNA detection
线性RCA又称单引物RCA,一条结合于环状DNA模板的引物沿着环延伸,产物为单环长度的数千倍的串联重复拷贝,最高可产生105倍的扩增[7]。线性RCA技术由于产物始终连接在起始引物上,所以信号易于固定是它的一大优势,非常适合于微阵列上的扩增检测。
指数RCA,也被称作超分支扩增HRCA(Hyper branched RCA)或RAM(Ramified RCA)[8-10],可使目标物扩增109倍以上,甚至实现单分子的核酸检测[11-13]。在指数RCA中,一条引物扩增出RCA产物,第二条引物与RCA产物杂化并延伸,置换已经结合在RCA产物上的下游引物延伸链,反复进行延伸和置换,产生树状的RCA扩增产物(图1b)[14]。在指数RCA扩增过程中引入或产生新的环状DNA作为模板,可以进行下一轮的RCA反应,进一步提高扩增的灵敏度[15]。
1.2 特异性成环-RCA技术进行核酸检测的关键
利用RCA技术进行基因诊断,关键是特异性地生成环状DNA。即只在有检测对象存在的条件下方可成环,而且只有成环的DNA才能进行扩增。一般将几十个碱基长的寡核苷酸,部分杂化到目标DNA上以后,使寡核苷酸的两末端相邻,形成一个缝隙(Nick),经过连接酶连接后形成闭合环状DNA(图2)。这一过程类似于将DNA分子特异性地锁定在目标DNA上,因此所用寡核苷酸探针被称作锁式探针(Padlock-probe)。理论上只有锁式探针的两端与检测对象的序列完全互补才能成环,但是在实际应用中,当离开连接处较远的部位出现不互补的碱基对时,探针也会成环,RCA扩增也会进行,造成假阳性的结果。尤其是在使用忠实度较低的T4 DNA连接酶时,甚至在没有任何靶序列作为模板的情况下也会成环[16]。为了提高成环的特异性,可以采用耐热性连接酶(如Ampligase,Taq ligase)在较高温度下进行连接[6,17]。有时需要进一步提高其特异性,如区分单核苷酸多态性。图3所示的改进方法可以达到检测SNP的要求,杂化后留一个6~10nt长的大缺口(Gap),只有另外一条短DNA与靶序列的缺口处完全互补时才能成环[6]。由于这条短DNA链中只要有一个错配,就不会杂化,特异性得到了很大提高。
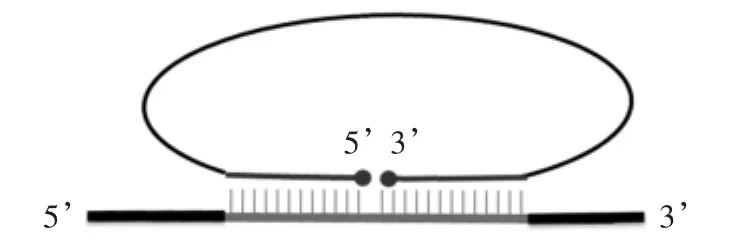
图2 锁式探针的特异性环化Fig.2 Specific circularization of the pad-lock probe
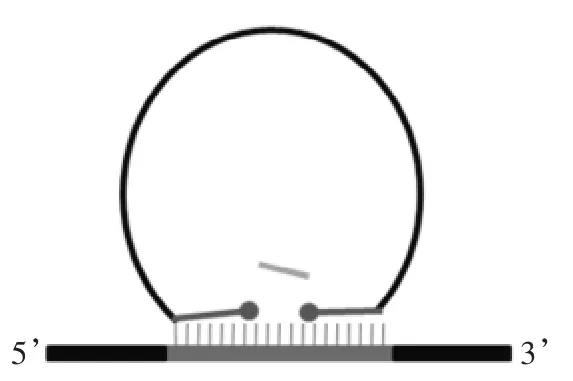
图3 中部留有一个大缺口的探针成环方式Fig.3 Circularization with a big gap at the middle position
在使用RCA技术检测双链DNA时,一般要对DNA进行高温变性,这无疑会增加非特异性成环和非特异性扩增的可能性。为了解决这个问题,Smolina等人使用特殊序列的PNA(Peptide nucleic acid)在不使双链DNA变性的条件下实现了特异性成环[18]。如图4所示,只在特定的部位通过与双链DNA中的一条链形成三螺旋(Triplex)而把DNA双链打开以形成DNA-loop,然后再将C-loop(Circular loop)在此部位连接成环。也可以先用一条被称作Capture probe的DNA将相关序列固定于固体或粒子表面,再进行RCA反应[11]。这样可以通过洗脱除去没有成环的非目标DNA,来提高检测的特异性。当用来检测较短的微小RNA(Mircro RNA)时,RNA可以直接作为引物进行线性RCA反应,即使在较长的非目标核酸(杂化处无自由的3’末端)上通过部分错配成环,所成环也不能进行RCA扩增,从而保证了检测反应的忠实度[19-20]。
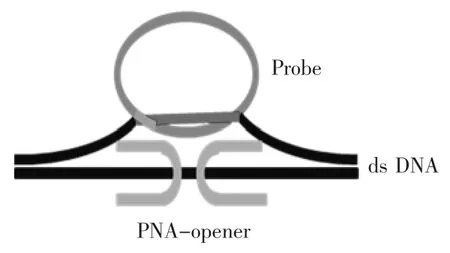
图4 PNA-opener用于原位检测的原理Fig.4 Principle of PNA-opener for in situ detection
1.3 高效RCA反应的技术要求
从RCA的扩增机理可以看出,无论是线性RCA还是指数型RCA,都要求DNA聚合酶具有很强的持续性(Good processivity)和链置换(Strand-displacement)能力。Phi29、Phage T7和Sequenase等聚合酶都可以满足以上要求[5]。其中Phi29聚合酶的效率最高,可以在30min内对靶核酸分子的扩增达到1000倍。但由于Phi29等聚合酶有较强的外切酶活性,有时需要对引物进行修饰以避免被降解。RCA反应也可以在热稳定性的聚合酶如Vent(exo-)和Bst DNA polymerase的作用下高温进行,但同时也容易发生非特异性的扩增[21-22]。有时加入可以同单链DNA结合的蛋白质有利于提高RCA反应的效率。
锁式探针的长度一般为30~100nt。过短则DNA双螺旋很难弯曲,在进行RCA过程中将会产生较大的张力和拓扑阻力,降低RCA的反应效率。扩增过程中生成的DNA将不断缠绕在环状DNA上,环过小将使扩增中途停滞[23],环过大时,拓扑障碍不复存在,但扩增的倍数将成比例降低,尤其对指数RCA影响较大。只要引物和DNA环结合,聚合酶的强大催化能力使RCA反应可在单分子水平进行。但是,当靶核酸浓度较低(fmol/L或amol/L数量级)时,成环反应一般要求探针的浓度较高(nmol/L数量级)。另一方面,加入较高浓度的待成环DNA探针,会产生非特异性成环和生成探针线性二聚体(两个探针分子在一个靶物分子上连接)等问题。使用较高浓度的高反应活性连接酶也有利于提高灵敏度,但同时也会使非特异性成环的概率增加。可以看出,在实际应用中灵敏度和高特异性总是一对矛盾,需根据具体情况合理选择反应条件。
1.4 RCA反应产物的检测
传统的RCA反应与PCR最大的不同在于:PCR靠引物本身与模板的特异性结合实现扩增的特异性;而RCA只是对加入的探针(C-probe)进行扩增。在用RCA进行基因检测时,一般靶序列的有无决定探针是否成环并进行RCA扩增。因此一旦成环,RCA扩增就会顺利进行,这往往是假阳性产生的重要原因。
检测RCA扩增产物的常规方法是凝胶电泳。线性RCA产物在凝胶电泳上呈现弥散状分布,指数RCA产物在凝胶电泳上的图样呈现阶梯状。在预先设计的锁式探针上引入限制性内切酶的酶切位点,将RAM信号扩增产物经过酶切后进行凝胶电泳,就会呈现单一条带[13]。同时在RCA反应中,也可以加入SYBR Green等染料对RCA反应进行实时检测。但SYBR Green染色无特异性,同非目的核酸结合也发出荧光,因此在使用过程中,应注意避免背景被核酸污染。
采用荧光标记的寡核苷酸探针可特异性检测RCA扩增产物。只有与标记的探针互补的DNA产物才能产生检测信号,有很好的特异性。常用的探针有分子信标(Molecular Beacon)、分子拉链(Molecular Zipper)等[24-26]。其中分子信标主要用于检测线性RCA;而分子拉链主要用于指数RCA的检测。分子拉链含有两条长度不同的寡核苷酸链,短链的3’末端接有一个荧光分子,长链的5’端带有荧光淬灭基团。两条链杂化后形成带有单链突出末端(Overhang)的双螺旋,而且荧光分子和淬灭基团相邻抑制荧光产生。在RCA过程中,突出的3’末端作为引物参与RCA反应,这样扩增过程中短链被置换下来,荧光发射基团和淬灭基团分离而产生荧光,生成可以检测到的信号。
以纳米金粒子标记的DNA探针还可以实现目测检测[27]。Li等利用毛细管电泳方法检测RCA产物,可以检测到少于2fmol的RCA产物[28]。Hu等用纳米金探针标记RCA产物后,利用表面增强拉曼光谱对产物进行高灵敏度检测[29]。即使有一个碱基的差别,也可以被识别,显示了较高的特异性。Long等将RCA产物固定于固相磁珠表面,利用电化学的方法检测RCA产物,最低能够检测到10amol/L的目标DNA和大约0.2pg/μL的基因组DNA[30]。
1.5 利用RCA技术进行环状基因组的全扩增
在生命科学领域,对基因组内所有的DNA进行同比例的全扩增(全基因组DNA扩增)越来越受到重视。最初全基因组扩增方法主要依靠大肠杆菌宿主体内自然扩增和PCR反应。采用宿主体内扩增的缺点是操作复杂,扩增周期长,而基于PCR的全基因组扩增方法经常导致非专一性扩增和染色体位点缺失,扩增产物量也较少[31-34]。RCA技术也可以用于基因组全扩增。对于大的环型DNA模板,例如质粒、噬菌体DNA、叶绿素和线粒体DNA等,采用多个引物或随机性引物进行指数RCA反应,可以达到较高质量的全扩增[35]。Amersham Biosciences公司的Templiphi DNA测序模板扩增试剂盒能够在几小时内从皮克量的起始材料得到微克量的DNA。由于Phi29 DNA聚合酶的高保真率和高延伸性使得扩增产物的长度和染色体位点保留都得到了极大提高,扩增产物可以直接用于测序[36]。
根据指数RCA类似的原理,使用随机序列的多种引物可对线性全基因组进行全扩增。Dean等人使用核酸酶抗性随机引物和Phi29 DNA聚合酶进行全基因组扩增,在30℃反应6h,能够从少至10个拷贝的基因组DNA中扩增DNA至20~30μg,产物的平均长度大于10kb[37]。这种方法甚至可以从全血样品中较均匀地扩增人类基因组DNA。对于降解较严重的DNA样品,Wang等开发了一种改进的全基因组扩增技术,RCA-RCA(限制性酶切和辅助环化滚环扩增)[38],即先使用限制性内切酶去降解DNA成小的片段,然后进行环化,再加入随机引物进行指数RCA扩增,扩增效果相对于以往方法有了很大改善。
2 RCA在传统检测领域的应用
RCA技术以其独特的优势在基因检测、基因诊断、原位检测、免疫检测和微阵列固相检测等领域表现出了极大的应用潜力。这些领域中的大量相关研究开辟了RCA技术良好的应用前景。
原位检测技术是利用核酸杂交方法,特异地与细胞内的目标核酸进行结合并用显微镜观测目标核酸的位置[39]。Schweitzer和Wiltshire等研究小组利用RCA技术对传统的免疫测定技术进行了改进[40-41]。具体有以下四个步骤:a.样品蛋白被固定在芯片上的抗体捕捉;b.生物素化(Biotinylated)的二次抗体与被捕捉的样品蛋白进行特异性结合;c.带有引物的通用抗体和二次抗体结合;d.加入环状DNA进行RCA信号扩增。近来也有研究小组利用筛选的适配体(Aptamer)和被检测蛋白直接亲和,然后进行滚环扩增Aptamer的检测方法,能够省去繁杂的蛋白亲和过程[42]。Nilsson最初研究利用锁式探针检测细胞中期和间期的染色体[7]。后来Christian研究小组等利用限制性内切酶处理细胞内的双链目标模板,使得用于探针结合的单链模板暴露,原位检测的准确度有了很大提升[43]。Somlina等人利用两个PNA openers打开双链DNA中的特定序列用于探针结合,省去了多步骤的酶切处理过程,简化了操作,同时也保证了准确性[18]。但是此方法只有在易形成三螺旋(Triplex)的特殊序列处才可使用,有一定的局限性。
基于SNP在分子生物学和遗传学中的重要地位,快速、高通量、廉价的SNPs分型方法有着广阔的应用前景。RCA技术可以很好地满足这些要求。Faruqi等通过特殊设计的锁式探针,缩短3’端的杂交臂长,升高杂交温度,可达到1∶100000的SNPs等位基因识别率[25]。由于采用了强力的扩增手段和高效的实时检测方法,使得研究者可以从1.0ng的基因组DNA中检测出SNP。Pickering等采用相同的方法,能够从单个同源基因中对所有等位基因实现同步检测[44]。
在检测单核苷酸多态性和新近出现的检测MicroRNA方面,RCA技术也具有显著的优势[45]。RNAi和各种小RNA调节作用的发现使人们开始重新审视RNA的功能,并有发现RNA新大陆的说法。因此小RNA的检测引起了人们的广泛关注。目前利用锁式探针检测MicroRNA的方法主要有三种形式:a.只是利用目标RNA作为引发引物进行扩增,锁式探针预先利用单链连接酶连接成环[19-20];b.锁式探针以目标RNA为模板成环,同时目标RNA作为引物进行扩增[46-47];c.预先反转录目标RNA,再利用反转录的DNA为模板进行RCA反应[48]。在以RNA为模板进行成环时,连接酶T4 RNA Ligase2较T4 DNA Ligase具有更高的特异性[46-47]。
Murakami等研究出了一种新的检测MicroRNA的方法,称作Primer-generation RCA(PG-RCA)[49]。它依靠MicroRNA模板特异性地产生可用作锁式探针的引物,同时在引物中引入酶切位点使得RCA的效率得到极大的提高,能够特异性地检测到浓度为15.9zmol的MicroRNA分子。另外Lagunavicius等[50]证明Phi 29 DNA聚合酶具有3’→5’单链RNA外切核酸酶活性,能够将目标RNA转化成扩增引物。Merkiene等利用E.Coli RNase III专一性辅助Phi29 DNA聚合酶,先将目标RNA和探针的杂交物进行RNase III处理,随后加入Phi29 DNA聚合酶,进行RCA产物延伸[51]。这种方法特异性良好,适用于待检测的RNA序列远离其3’末端时的检测。
芯片上的DNA扩增可以实现高通量检测。Nallu等利用RCA反应扩增固定于微阵列上的引物,能够在微阵列芯片上检测到最低浓度480fmol/L的固定引物,比相同条件下均相中的杂交反应敏感度增加了8000倍[52]。同时在微阵列上进行SNP检测,比不采用RCA的直接杂交检测的灵敏度提高2个数量级(1nmol/L到10pmol/L)。但如何将大量不同序列的待检测引物在芯片上固定仍是一大难题。如果能在芯片上特异性成环并进行特异性扩增,无疑将大大拓展这一技术的应用前景。
目前RCA技术已经有被应用到食品中的例子,利用锁式探针特异地与从食品中提取的转基因模板杂交,连接成环后进行RCA[53]。这种方法可以快速检测进出口食品中的转基因成分,其特异性好,灵敏度高。利用RCA技术可检测食品中的卵清蛋白,基于抗原抗体特异结合后利用RCA进行扩增[54]。也有研究者使用RCA检测重金属铅,利用Aptamer同铅离子的特异性结合,然后用RCA进行信号扩增以判断目标物的存在与否[55]。
3 核酸纳米技术与RCA
纳米技术与RCA技术结合可以取得非常好的检测效果。有研究人员利用纳米金结合的寡核苷酸探针来检测滚环扩增产物,操作简便,灵敏度高[56];Cheng等利用Quantum Dot来检测RCA扩增产物,将此种方法应用于免疫测定,能够检测到100μL内的16个样品分子,在1.0amol/L到1.0pmol/L的范围内呈现良好线性关系[57]。除了纳米级金属材料应用于滚环扩增产物检测以外,纳米级的生物材料也被应用于滚环扩增中。Lin等利用Aptamer和RCA技术结合检测溶液中的ATP,最低能够检测到10μmol/L的ATP分子[58]。
Edda等利用滚环扩增技术扩增具有交叉结构的DNA纳米结构分子,即以RCA技术制备DNA纳米材料[59]。其团队引入一种限制性内切酶辅助将RCA产物酶切成单个拷贝,然后利用连接酶连接成环,重复RCA扩增和酶切过程,得到大量的目标DNA分子。但是,RCA技术仍很难解决核酸纳米材料在实际应用上的诸多难题。
4 结论与展望
滚环扩增作为一种高效的核酸扩增手段,因其等温特性和引物末端易固定的特点,既可实现溶液中的特异性扩增,又可用于微阵列芯片进行高通量检测。在基因诊断与检测、原位检测等领域的应用潜力使之可称为具有广泛应用前景的先端生物技术。滚环扩增技术引物设计简单,产物分析手段多样,符合后基因组时代核酸检测的快速、准确、高通量等要求。尤其是它可用于小RNA分子的检测,在快速发展RNA研究领域可以得到较大的发展。而且RCA技术可方便地同纳米技术、荧光探针、核酸芯片等技术相结合,使其应用前景更广阔。另一方面,目前RCA技术也存在难以同时达到高特异性、高灵敏度和高通量的问题。在实际应用中也有操作繁琐和成本高的弱点,难以取得巨大的经济效益,仍然需要进行有针对性的深入研究以解决上述问题,最终使之成为强有力的技术工具。
[1]Zhao W,Ali M M,Brook M A.Rolling Circle Amplification:Applications in nanotechnology and biodetection with functional[J].Angewandte Chemie International Edition,2008,47 (34):6330-6337.
[2]CaoW G.Recentdevelopmentsin ligase-mediated amplification and detection[J].Trends in Biotechnology,2004,22(1):38-44.
[3]Fire A,Xu S Q.Rolling replication of short DNA circles[J].Proceedings of the National Academy of Sciences,1995,92(10):4641-4645.
[4]Liu D Y,Sarah L,Daubendiek S L,Zillman M A,et al.Rolling Circle DNA synthesis:small circular oligonucleotides as efficient templates for DNA polymerases[J].Journal of American Chemical Society,1996,118(7):1587-1594.
[5]Sarah L D,Kevin R,Eric T K.Rolling-Circle RNA Synthesis:circular oligonucleotides as efficient substrates for T7 RNApolymerase[J].Journal of American Chemical Society,1995,117(29):7818-7819.
[6]Lizardi P M,Huang X H,Zhu Z R,et al.Mutation detection and single-molecule counting using isothermal rolling-circle amplification[J].Nature Genetics,1998,19(3):225-232.
[7]BanérJ,Nilsson M,Mendel-HartvigM,etal.Signal amplification of padlock probes by rolling circle replication[J].Nucleic Acids Research,1998,26(22):5073-5078.
[8]Zhang D Y,Brandwein M,Hsuih T,et al.Ramification amplification:a novel isothermal DNA amplification method[J].Molecular Diagnosis,2001,6(1):141-150.
[9]Thomas D C,Nardone G A,Randall S K.Amplification of padlock probes for DNA diagnostics by cascade rolling circle amplification or the polymerase chain reaction[J].Archives of Pathology&Laboratory Medicine,1999,123(12):1170-1176.
[10]Murakami T,Sumaoka J,Komiyama M.Sensitive isothermal detection of nucleic-acid sequence by primer generation-rolling circle amplification[J].Nucleic Acids Research,2009,37(3):e19.
[11]Goransson J,Wahlby C,Isaksson M,et al.A single molecule array for digital targeted molecular analyses[J].Nucleic Acids Research,2009,37(1):e7.
[12]Schopf E,Chen Y.Attomole DNA detection assay via rolling circle amplification and single molecule detection[J].Analytical Biochemistry,2010,397(2):115-117.
[13]Schwartz J J,Quake S R.High density single molecule surface patterning with colloidal epitaxy[J].Applied Physics letters,2007,91(8):083902.
[14]Zhang D Y,Brandwein M,Hsuih T C,et al.Amplification of target-specific,ligation-dependent circular probe[J].Gene,1998,211(3):277-285.
[15]Zou B J,Ma Y J,Wu H P,Zhou G H.Signal amplification by rolling circle amplification on universal flaps yielded from target-specific invasive reaction[J].Analyst,2012,137(3):729-734.
[16]Kuhn H,Frank-Kamenetskii M D.Template-independent ligation of single-stranded DNA by T4 DNA ligase[J].Federation of European Biochemical Societies Journal,2005,272(23):5991-6000.
[17]Qi X Q,Saleha B,Katrien M D.L-RCA (ligation-rolling circle amplification):a general method for genotyping of single nucleotide polymorphisms(SNPs)[J].Nucleic Acids Research,2001,29(22):e116.
[18]Smolina I,Lee C,Frank-Kamenetskii M,et al.Detection of low-copy-number genomic DNA sequences in individual bacterial cells by using peptide nucleic acid-assisted rolling-circle amplification and fluorescence in situ hybridization[J].Applied and Environment Microbiology,2007,73(7):2324-2328.
[19]Mashimo Y,Mie M,Kobatake E,et al.Detection of small RNA molecules by a combination of branched rolling circle amplification and bioluminescentpyrophosphate assay[J].Analytical and Bioanalytical Chemistry,2011,401(1):221-227.
[20]Takahashi H,Matsumoto A,Sugiyama S,et al.Direct detection of green flourescent protein messenger RNA expressed in Escherichia coli by rolling circle amplification[J].Analytical Biochemistry,2010,401:242-249.
[21]Vigneault F,Drouin R.Optimal conditions and specific characteristics of Vent exo-DNA polymerase in ligation-mediated polymerase chain reaction protocols[J].Biochemistry and Cell Biology,2005,83(2):147-165.
[22]Aliotta J M,Pelletier J J,Ware J L,et al.Thermostable Bst DNA polymerase I lacks a 3’ →5’proofreading exonuclease activity[J].Genetic Analysis:Biomolecular Engineering,1996,12(5):185-195.
[23]Kuhn H,Demidov V V,Frank-Kamenetskii M D.Rollingcircle amplification under topological constraints[J].Nucleic Acid Research,2002,30(2):574-580.
[24]Nilsson M,Gullberg M,Dahl F,et al.Real-time monitoring of rolling-circle amplification using a modified molecular beacon design[J].Nucleic Acids Research,2002,30(14):e66.
[25]Faruqi F A,Hosono S,Driscoll M D,et al.High-throughput genotyping of single nucleotide polymorphisms with rolling circle amplification[J].BMC Genomics,2001,2(1):4-9.
[26]Yi J Z,Zhang W D,Zhang D Y.Molecular zipper:a fluorescent probe for real-time isothermal DNA amplification[J].Nucleic Acids Research,2006,34(11):e81
[27]Li J S,Deng T,Chu X,et al.Rolling circle amplication combined with gold nanoparticle aggregates for highly sensitive identification of single nucleotide polymorphisms[J].Analytical Chemisrty,2010,82(7):2811-2816.
[28]Li N,Li J S,Zhong W W.CE combined with rolling circle amplification for sensitive DNA detection[J].Electrophoresis,2008,29(2):424-432.
[29]Hu J,Zhang C Y.Sensitive detection of nucleic acids with rolling circle amplification and surface-enhanced raman scattering spectroscopy[J].Analytical Chemisrty,2010,82(21):8991-8997.
[30]Long Y,Zhou X M,Xing D.Sensitive and isothermal electrochemiluminescence gene-sensing of Listeria monocytogeneswith hyper-branching rolling circle amplification technology[J].Biosensors and Bioelectronics,2011,26(6):2897-2904.
[31]Cheung V G,Nelson S F.Whole genome amplification using a degenerate oligonucleotide primer allows hundreds of genotypes to be performed on less than one nanogram of genomic DNA[J].Proceedings of the National Academy of Sciences,1996,93(25):14676-14679.
[32]Paunio T,Reima I,Syvanen A.Preimplantation diagnosis by whole-genome amplification,PCR amplification,and solid-phase minisequencing of blastomere DNA [J].Clinical Chemistry,1996,42(9):1382-1390.
[33]Telenius H,Carter N P,Bebb C E,et al.Degenerate oligonucleotide-primed PCR:General amplification of target DNA by a single degenerate primer[J].Genomics,1992,13(3):718-725.
[34]Zhang Lin,Cui X F,Schmitt K,et al.Whole genome amplification from a single cell:Implications for genetic analysis[J].Proceedings of the National Academy of Sciences,1992,89(13):5847-5851.
[35]Dean F B,Nelson J R,Giesler T L,et al.Rapid amplification of plasmid and phage DNA using Phi29 DNA polymerase and multiply-primed rolling circle amplification[J].Genome Research,2001,11(6):1095-1099.
[36]Garmendia C,Bernad A,Esteban J A,et al.The Bacteriophage φ29 DNA polymerase,a proofreading enzyme[J].Journal of Biological Chemistry,1992,267(4):2594-2599.
[37]Dean F B,Hosono S,Fang L H,et al.Comprehensive human genome amplification using multiple displacement amplification[J].Proceedings of the National Academy of Sciences,2002,99(8):5261-5266.
[38]Wang G,Maher E,Brennan C,et al.DNA amplification method tolerant to sample degradation[J].Genome Research,2010,14(11):2357-2366.
[39]Haase A T,Retzel E F,Staskus K A.Amplification and detection of lentiviral DNA inside cells[J].Proceedings of the National Academy of Sciences,1990,87(13):4971-4975.
[40]Schweitzer B,Wiltshire S,Lambert J,et al.Immunoassays with rolling circle DNA amplification:A versatile platform for ultrasensitive antigen detection[J].Proceedings of the National Academy of Sciences,2000,97(18):10113-10119.
[41]Kingsmore S F,Patel D D.Multiplexed protein profiling on antibody-based microarrays by rolling circle amplification[J].Current Opinion in Biotechnology,2003,14(1):74-81.
[42]Cho E J,Yang L,Levy M,et al.Using a deoxyribozyme ligase and rolling circle amplification to detect a non-nucleic acid analyte,ATP[J].Journal of American Chemical Society,2005,127(7):2022-2023.
[43]Christian A T,Pattee M S,Attix C M,et al.Detection of DNA point mutations and mRNA expression levels by rolling circle amplification in individual cells[J].Applied Biological Sciences,2001,98(25):14238-14243.
[44]Pickering J,Bamford A,Godbole V,et al.Integration of DNA ligation and rolling circle amplification for the homogeneous,end-point detection of single nucleotide polymorphisms[J].Nucleic Acid Research,2002,30(12):60.
[45]Nilsson M,Antson D O,Barbany G,et al.RNA-templated DNA ligation for transcript analysis[J].Nucleic Acids Research,2001,29(2):578-581.
[46]Jonstrup S P,Koch J,Kjems J.A microRNA detection system based on padlock probes and rolling circle amplification[J].RNA 2006,12(9):1747-1752.
[47]Cheng Y Q,Zhang X,Li Z P,et al.Highly sensitive determination of microRNA using target-primed and branched rolling-circle amplification[J].Angewandte Chemie International Edition,2009,48(18):3268-3272.
[48]Yao B,Li J,Huang H,et al.Quantitative analysis of zeptomole microRNAs based on isothermal ramification amplification[J].RNA,2009,15(9):1787-1794.
[49]Murakami T,Sumaoka J,Komiyama M.Sensitive RNA detection by combining three-way junction formation and primer generation-rolling circle amplification[J].Nucleic Acids Research,2012,40(3):e22.
[50]Lagunavicius,A Kiveryte,Z Zimbaite-Ruskuliene,et al.Duality of polynucleotide substrates for Phi29 DNA polymerase:3’→5’ RNase activity of the enzyme[J].RNA,2008,14(3):503-513.
[51]Merkiene E,Gaidamaviciute E,Riauba L,et al.Direct detection of RNA in vitro and in situ by target-primed RCA:the impact of E.coli RNase III on the detection efficiency of RNA sequences distanced far from the 3’-end[J].RNA,2010,16(8):1508-1515.
[52]Nallur G,Luo C H,Fang L H,et al.Signal amplification by rolling circle amplification on DNA microarrays[J].Nucleic Acids Research,2001,29(23):e118.
[53]Pang S,Qureshi F,Shanahan D.Investigation of the use of rolling circle amplification for the detection of GM food[J].European Food Research and Technology,2007,225(1):59-66.
[54]Kobori T,Matsumoto A,Takahashi H,et al.Rolling Circle Amplification for signal enhancement in ovalbumin detection[J].Analytical Sciences December,2009,25(12):1381-1383.
[55]Jacobi Z E,Li L,Liu J W.Visual detection of lead (II)using a label-free DNA-based sensor and its immobilization within a monolithic hydrogel[J].Analyst,2012,137(3):704-709.
[56]Hsu H Y,Huang Y Y.RCA combined nanoparticle-based optical detection technique for protein microarray:a novel approach[J].Biosensors and Bioelectronics,2004,20(1):123-126.
[57]Cheng W,Yan F,Ding L,et al.Cascade Signal Amplification strategy for subattomolar protein detection by rolling circle amplification and quantum dots tagging[J].Analytical Chemistry,2010,82(8):3337-3342.
[58]Lin C X,Wang X,Liu Y,et al.Rolling circle enzymatic replication of a complex multi-crossover DNA nanostructure[J].Journal of American Chemical Society,2007,129(3):14475-1448.
[59]Edda Reiβ,Ralph Holzel,Frank F Bier.Synthesis and stretching of rolling circle amplification products in a flow-through system[J].Small,2009,5(20):2316-2322.
Research progress of rolling circle amplification of DNA
WANG Xiao-liang,WANG Xing-yu,LIANG Chang-cheng,DONG Ping,LIANG Xing-guo*
(College of Food Science and Engineering,Ocean University of China,Qingdao 266003,China)
Q556
A
1002-0306(2012)14-0358-06
2012-03-10 *通讯联系人
王晓亮(1986年-),男,硕士研究生,研究方向:食品化学与营养。
“国家青年千人计划”;“长江学者和创新团队发展计划”;“山东省万人计划”。

