青枯菌Po82菌株Ⅲ型分泌系统调控基因hrpB的功能研究
刘 蕾, 徐 进, 许景升, 陈匡宇, 张丽勍, 张 昊, 冯 洁
(中国农业科学院植物保护研究所,植物病虫害生物学国家重点实验室,北京 100193)
青枯菌Po82菌株Ⅲ型分泌系统调控基因hrpB的功能研究
刘 蕾, 徐 进, 许景升, 陈匡宇, 张丽勍, 张 昊, 冯 洁*
(中国农业科学院植物保护研究所,植物病虫害生物学国家重点实验室,北京 100193)
本文以青枯菌致病力分化菌株Po82的Ⅲ型分泌系统调控基因hrpB为研究对象,采用同源重组双交换法,构建获得青枯菌Po82菌株的hrpB基因缺失突变株Po82ΔhrpB及其互补菌株Po82ΔhrpB-pML123-hrpB,并对野生型菌株、突变株和互补菌株进行了致病力及生物学功能的验证。致病力测定结果表明,青枯菌hrpB基因缺失突变株Po82ΔhrpB的致病力较Po82野生型菌株显著下降,而互补菌株Po82ΔhrpB-pML123-hrpB能够部分恢复突变菌株的致病力。生长曲线测定结果表明,在营养贫瘠型培养基中,hrpB基因缺失突变株Po82ΔhrpB的生长速率较野生型青枯菌Po82菌株快,但是在营养丰富型培养基中,两者生长速率基本一致。野生型和突变株的运动性测定结果显示,两者的运动性无显著差异。表明hrpB基因在青枯菌致病过程中具有重要影响,并对进一步发掘鉴定Po82菌株中新的Ⅲ型效应子,进而深入解析其致病力分化的分子机理具有重要的作用。
青枯菌; Ⅲ型分泌系统;hrpB基因; 致病性
茄科雷尔氏菌[Ralstoniasolanacearum(Smith) Yabuuchi et al.],简称青枯菌,是一种地域分布广且寄主范围非常大的毁灭性植物病原细菌[1]。进入寄主植物根部后,青枯菌侵入木质部导管,随后通过维管束系统快速扩展至植物的地上部分。青枯病典型的快速萎蔫症状是由病原菌在维管束中产生大量胞外多糖从而阻塞水分运输所致。青枯菌作为研究寄主植物与病原细菌分子互作的模式系统之一,目前通过分子遗传学的研究手段已经鉴定出了多个与致病相关的基因[2-3]。与其他革兰氏阴性菌相同,Ⅲ型分泌系统(typeⅢsecretion system,T3SS)在青枯菌致病过程中起重要作用。Ⅲ型分泌系统通过高度保守的装置将蛋白分泌至细胞外,并且通过可变的胞外装置将效应子蛋白输送至植物细胞内,干扰宿主细胞信号转导通路,为病原菌在细胞内存活创造有利条件[4-5]。试验证明,Ⅲ型分泌系统缺失突变株无法引起寄主的病害特征[6-7]。
青枯菌菌体细胞与植物细胞物理接触后,感知来自寄主植物的信号,继而启动复杂的调控网络系统控制Ⅲ型分泌系统及其效应子编码基因的表达。近年来,针对植物信号所调控的hrp(hypersensitive response and pathogenicity)基因簇的研究[8-9]明确了细菌的胞外受体蛋白PrhA(plant regulator of hrp gene)接收植物细胞信号,并将信号依次转导并激活调控基因prhJ、prhJ和hrpG基因的表达,并最终调控hrpB基因的表达。其中,PrhA、PrhJ、HrpG是植物信号的中间递体,信号的最终受体是hrpB基因[10]。在基础培养基(minimal medium)中或者与植物细胞共培养,hrp基因簇的表达依赖于转录 激 活 因 子 HrpB[9,11]。HrpB 调 控 因 子 属 于AraC家族,位于青枯菌调控网络的最下游。它不仅激发hrp基因簇中各转录单元及其上游popABC操纵子的表达,同时还调控了其他多个基因的表达,其中包括许多依赖T3SS分泌系统释放的效应蛋白。作为调控因子,HrpB的作用原理是通过与Ⅲ型分泌系统装置蛋白及效应蛋白编码基因上游的hrpII框(TTCGN16TTCG)顺式作用元件结合,从而启动这些基因的表达[6]。近年来,对于hrpB突变株的转录组学分析表明,HrpB调控因子不仅控制Ⅲ型分泌系统组成基因以及超过60种的效应子基因的表达,而且控制依赖于Ⅲ分泌系统输出途径相关基因的表达[5]。因此,调控因子HrpB参与青枯菌侵染植物过程,并且对青枯菌的致病性有重要影响。
茄科雷尔氏菌是一个高度复杂的种群。2009年,本实验室依照国际新近提出的演化型分类框架对保存的286株青枯菌进行了遗传多样性的研究[12-13]。并从中发现一株编号为Po82的菌株,该菌株的演化型分类归属为演化型Ⅱ/序列变种4,不仅可以对番茄、马铃薯、茄子等茄科植物致病,而且同时又对香蕉致病。Po82菌株具有NPB菌株(no pathogenic to banana)和传统香蕉菌株的致病特征,因此,Po82菌株是一株珍贵的致病力分化菌株[14-15]。
本试验以青枯菌致病力分化菌株Po82为研究对象,采用同源重组双交换的方法,使用庆大霉素抗性基因对Ⅲ型分泌系统调控基因hrpB进行了替换。获得了青枯菌菌株Po82的hrpB基因突变株,命名为Po82ΔhrpB。通过致病力测定以及相关生物学研究,明确了Po82ΔhrpB的生物学特性,为进一步研究青枯菌株Po82的Ⅲ型分泌系统及发掘新的Ⅲ型效应子奠定了基础。
1 材料与方法
1.1 材料
1.1.1 供试菌株、质粒及培养条件
本研究所使用的菌株、质粒特征及来源见表1。青枯菌株Po82采用NA培养基,28℃条件下培养24~48h。大肠杆菌DH5α采用LB培养基,37℃培养12~16h。
表1 试验中所使用的菌株和质粒的来源及特征
1.1.2 培养基
突变株筛选培养基为NA培养基附加蔗糖贮存液至终浓度10%。抗生素使用终浓度为:氨苄青霉素(Amp):100μg/mL;卡那霉素(Kan):50μg/mL;庆大
霉素(Gm):50μg/mL。基础培养基(minimal medium):Boucher培养基[18]中加入谷氨酸至终浓度20 mmol/L;半固体培养基(SMM):葡萄糖100mg,蛋白胨100mg,乙二胺四乙酸二钠38mg,pH=7.0的磷酸盐缓冲液10mL,琼脂3.5g,用蒸馏水定容到1 000mL,不调酸度,121℃15min灭菌。
1.1.3 主要试剂
Taq酶,dNTP,GC bufferⅠ/Ⅱ,限制性内切酶,T4DNA连接酶,T-载体等均购自大连宝生物(TaKaRa)公司;DNA ladder,琼脂糖凝胶回收试剂盒,质粒小提试剂盒,细菌基因组DNA提取试剂盒等均购自北京天根生物工程公司;氨苄青霉素(Amp),卡那霉素(Kan),庆大霉素(Gm),IPTG,X-gal为Sigma公司产品;引物和测序均由上海生工生物工程技术服务有限公司合成;其余试剂为国产分析纯。
1.2 方法
1.2.1 青枯菌Po82菌株hrpB基因缺失突变株的构建
青枯菌菌株Po82基因组DNA的提取步骤参见北京天根生物工程公司说明书。突变菌株构建采用同源置换法,以庆大霉素抗性基因作为突变体筛选的抗性基因。将hrpB基因上游(UP)和下游(DOWN)各500bp左右片段,以及庆大霉素抗性基因 (gm),按 照 UP-gm-DOWN 的 顺 序 构 建 至pk18mobsacB载体中,进行重组自杀质粒的构建。根据NCBI中已发表的Po82的全基因组序列,分别提取hrpB基因上下游各500bp左右的序列,并设计 PCR 扩 增 引 物:HrpB-UP(forward):CCG GAATTC CGCGGCGATCAGAACACG(下划线部分为EcoR I酶切位点);HrpB-UP(reverse):CGC GGATCC GGCAATGCTC CTGAAGC(下划线部分为BamH I酶切位点);HrpB-DOWN(forward):TGC TCTAGA GCCTCTCCAGCGGACCG(下划线部分为XbaI酶切位点);HrpB-DOWN(reverse):CCC AAGCTT AGGTCGCGCA CCAGCTCGACC(下划线部分为HindⅢ酶切位点)。庆大霉素抗性基因:GMF:CGC GGATCC GGACGCACACCGTGGAAA(下划线部 分 为BamH I酶 切 位 点);GM-R:TGC TCTAGA GGCGGCGTTGTGACAATTT(下划线部分为XbaI酶切位点)。PCR扩增条件:94℃5min;94℃30s,58℃30s,72℃1min,共30个循环;72℃7min。构建好的重组自杀质粒(pk18-hr-pB-gm)交由上海生工进行序列测定。
青枯菌感受态细胞的制备以及电击转化参照Ausubel等[19]以及 Laiva[20]等方法。将构建好的重组自杀质粒pk18-hrpB-gm电击导入青枯菌Po82菌株中,经过3次筛选[21]及PCR验证,最终获得抗庆大霉素和蔗糖,并且对卡那霉素敏感的hrpB基因突变株Po82ΔhrpB。
挑取对卡那霉素敏感的菌落进行PCR扩增验证。PCR 引 物:HrpB-UP (forward)/ HrpBDOWN(reverse),以及根据hrpB基因ORF中间序列设计引物:BORF-F:ATCCGCCATTCGGATGACC;BORF-R:ACGACTTCCACCTTGGTCTG。PCR扩增条件:94℃5min;94℃30s,58℃30s,72℃2min,共30个循环;72℃7min。
1.2.2 互补菌株的构建
根据已发表的hrpB基因序列,设计PCR引物,扩增hrpB基因的ORF序列,经酶切连接至互补载体pML123中。hrpB基因引物序列为,BF:GC GGATCC ATGCTGGGAAACATCTACTTCG(下划线部分为BamH I酶切位点);BR:CCC AAGCTT TCAGCGCCAG ATGGTTTCGG AGG(下划线部分为HindⅢ酶切位点)。根据载体pML123序列设计引物PR:AGCCGAATAGCCTCTCCACCC,与BF进行PCR验证。PCR扩增条件:变性94℃5min;30个循环(94℃30s,58℃30s,72℃1min);延伸72℃7min。构建好的互补载体(pML123-hrpB)交由上海生工进行序列测定。
制备青枯菌Po82菌株电击感受态,将构建好的重组表达载体pML123-hrpB电击导入青枯菌Po82ΔhrpB菌株中,在含有卡那霉素和庆大霉素的平板上进行筛选,最终获得具有抗性的互补菌株Po82ΔhrpB-pML123-hrpB。
1.2.3 野生型、突变菌株以及互补菌株致病力测定
参照He等[22]伤根接种法,接种番茄感病品种‘中蔬5号’。野生型、突变株以及互补菌株各接种10株,重复3次。接种后的番茄植株置于光照培养箱中进行培养,培养条件为:16h光周期,(32±1)℃/(28±1)℃,相对湿度90%。每天调查发病情况,持续观察2~3周,记录病情指数。青枯病的病情分级及调查方法参照 Meng等[23]的方法。用SAS统计软件中LSD(p=0.05)进行数据分析。
1.2.4 突变菌株以及野生型菌株生长曲线的测定
挑取Po82野生型及hrpB基因突变株Po82ΔhrpB单菌落于NA液体培养基中过夜培养,离心收集菌体。生长曲线的测定参照 Kanda等[24]方法:(1)以基础液体培养基悬浮菌体,调节A600至相同大小(约为0.005),28℃,200r/min振荡培养,间隔6h测量A600的值,试验共重复3次。(2)以NA液体培养基悬浮菌体,调节A600至相同大小(约为0.005),28℃,200r/min振荡培养,间隔6h测量A600的值,试验共重复3次。
1.2.5 突变菌株以及野生型菌株运动性检测
运动性检测参照Kelman等方法[25]。各取5μL浓度为3×108cfu/mL的野生型和突变株的菌体悬浮液,滴加至半固体SMM平板上,28℃条件下培养3~5d。观察细菌的运动性情况,并使用直尺进行测量。
2 结果
2.1青枯菌Po82菌株hrpB基因缺失突变株的构建
本试验中,使用庆大霉素抗性基因作为突变体筛选的抗性基因。将基因上游(UP)和下游(DOWN)各500bp左右片段,以及庆大霉素抗性基因(gm,长度为855bp),按照 UP-gm-DOWN 的顺序构建至pk18mobsacB载体中,构建hrpB基因重组自杀质粒(pK18-hrpB-gm)。提取质粒使用限制性内切酶EcoR I和HindⅢ进行酶切以及测序验证。酶切结果如图1所示表明,泳道1中有2条带,分别位于6.0kb左右和1.8kb左右。其中,空载pk18mobsacB 长 度 约 为 5.6kb,片 段 UP-gm-DOWN长度为1.8kb左右。因此,片段 UP-gm-DOWN已成功连接至pk18mobsacB。
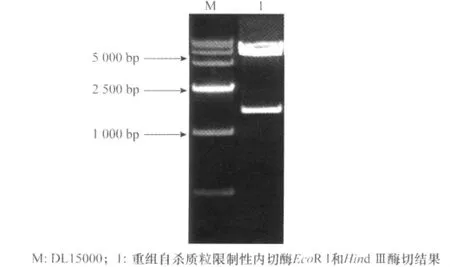
图1 hrpB基因重组自杀质粒的构建
2.2 青枯菌菌株Po82突变株的筛选与鉴定
将hrpB基因重组自杀质粒(pK18-hrpB-gm)电击转化至青枯菌菌株Po82中,经3步筛选,获得hrpB基因突变株Po82ΔhrpB。使用PCR方法,对突变株进行鉴定。分别以野生型菌株Po82的全基因组DNA,hrpB基因重组自杀质粒,以及hrpB基因突变株的全基因组DNA为模板,使用引物为HrpB-UP(forward)/HrpB-DOWN(reverse),以 及hrpB基因部分 ORF序列引物BORF-F/BORF-R。PCR鉴定结果如图2所示。从PCR结果可以看出,使用 引 物 HrpB-UP(forward)/HrpB-DOWN(reverse)进行PCR扩增,野生型菌株片段长度为2.4kb左右(图2,泳道3基因上游及下游序列各500bp,hrpB基因 ORF长度为1.4kb),hrpB基因重组自杀质粒和hrpB基因突变株扩增片段为1.8kb(图2,泳道1,2);使用引物BORF-F/BORFR进行扩增,野生型菌株扩增出约为600bp的片段(图2,泳道6),而重组自杀质粒(图2,泳道5)和hrpB基因突变株未能扩增出片段(图2,泳道4)。
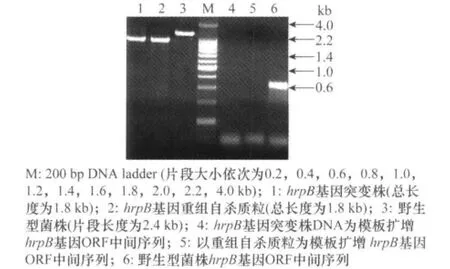
图2 hrpB基因突变株筛选结果
2.3 互补菌株的构建与鉴定
根据青枯菌Po82菌株hrpB基因的ORF序列设计引物,并连接至互补载体pML123中,构建载体pML123-hrpB。使用引物BF/PR进行PCR验证,PCR扩增所获得的片段长度约为1.6kb。结果表明,hrpB基因已成功连接至载体pML123中。随后,将载体pML123-hrpB电转化转入突变株Po82△hrpB中,于含有庆大霉素和卡那霉素的NA平板筛选转化子。用引物BF/PR进行PCR和测序验证,最后,将得到的互补菌株命名为Po82ΔhrpB-pML123-hrpB(图3)。
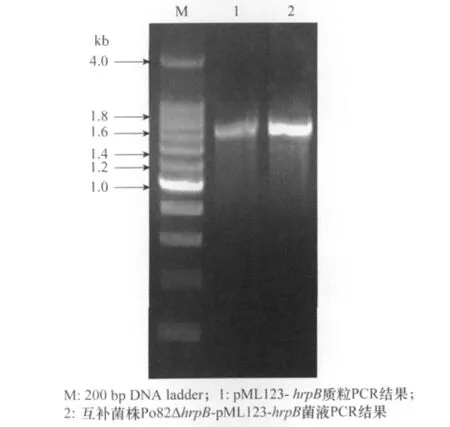
图3互补菌株筛选结果
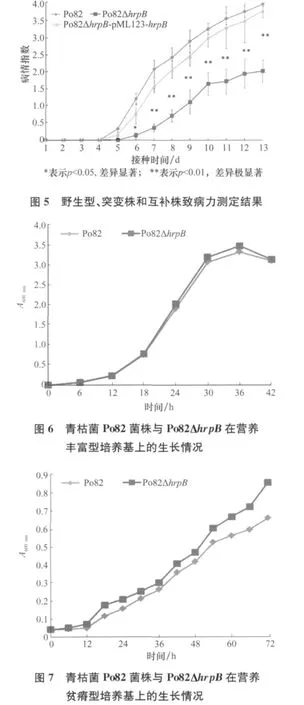
2.4 致病力测定
伤根浇注菌液接种法接种番茄进行致病力测定,统计结果表明,Po82野生型菌株接种后的番茄植株,于第5天开始发病,而突变株接种后植株则在第6天开始表现出明显萎蔫症状;接种后10d,接种野生型菌株植株的病情指数已高达3.73,而hrpB基因突变株Po82ΔhrpB仅为1.63,较野生型菌株降低了56.3%,SAS方差分析结果表明两者差异达极显著水平(图4、图5)。与野生型菌株相比,互补菌株Po82ΔhrpB-pML123-hrpB能够部分恢复突变菌株的致病力。

图4 野生型、突变株和互补株接种番茄第10天发病情况
2.5 生长曲线的测定
分别对青枯菌Po82野生型菌株和hrpB基因突变株Po82ΔhrpB在营养丰富型培养基(NA液体培养基)和营养贫瘠型培养基(Boucher基础培养基)中的生长曲线进行了测定。图6结果表明,青枯菌Po82野生型菌株和突变株Po82ΔhrpB在营养丰富型培养基中生长速率无明显差异;方差分析结果表明,在营养贫瘠型培养基中培养18h后,两者生长速率开始表现出明显差异(p<0.05),Po82ΔhrpB比野生型菌株生长速率要快。(图7)。
2.6 运动性测定
使用半固体培养基,研究青枯菌株Po82野生型菌株和hrpB基因突变株Po82ΔhrpB运动性。通过测量菌落直径,青枯菌株Po82野生型菌株和hrpB基因突变株Po82ΔhrpB的运动能力没有差异(图8)。结果表明,hrpB基因的突变对青枯菌Po82菌株的运动性没有影响。

图8 野生型菌株Po82与hrpB基因突变
3 结论与讨论
作为世界范围内最为重要的细菌病害之一,青枯病受到了各国植物病理学家的广泛关注。近年来随着全球经济一体化进程的加快,外来有害生物入侵日益成为各国关注的热点。其中,作为有害外来生物,香蕉细菌性枯萎病(茄雷尔氏菌2号小种)的高风险入侵途径主要为带菌植物材料,并且该病菌在我国属于具有潜在入侵风险的危险性外来有害生物,被列为我国的检疫对象[26]。本实验室保存的青枯菌Po82菌株由于其寄主范围的特殊性,本研究将其列为研究对象,对于深入解析青枯菌致病力进化的分子机理具有重要意义。
T3SS作为植物病原细菌分泌毒力因子的重要装置,已成为植物病理学界的研究热点之一[27]。hrpB起着转录激活因子的作用,不仅激发本基因簇中各转录单元及popABC操纵子的表达,同时调控了菌体中其他基因的表达,其中许多为依赖T3SS分泌系统释放的效应蛋白。近年来,针对hrpB基因的研究已于多个青枯菌株中展开。Vasse等[28]对青枯菌1号小种菌株GMI1000中的hrpB基因功能进行研究,结果表明,hrpB突变株在番茄根部维管束系统中的侵染、定殖和扩展能力都有所减弱。Kanda等[24]对烟草青枯菌OE1-1的hrpB基因进行突变,获得与之类似的结果。本试验利用同源基因置换法构建了青枯菌致病力分化菌株Po82的hrpB基因突变株Po82ΔhrpB,并对该突变株的致病力及生物学特性进行了分析。研究结果表明,hrpB基因突变株Po82ΔhrpB相对于野生型菌株Po82,致病力明显下降,而互补菌株Po82ΔhrpB-pML123-hrpB能够部分恢复突变株菌株的致病力。综上所述,hrpB基因对于青枯菌Po82菌株的致病性起关键作用。
Kanda等[24]对烟草青枯菌 OE1-1的生长曲线进行了测定,结果表明,无论是在营养丰富型培养基和营养贫瘠型培养基中,野生型菌株OE1-1与hrpB基因突变株的生长速率均保持一致。本试验对青枯菌Po82菌株的生长曲线进行研究,结果表明,在营养丰富型培养基中,野生型青枯菌Po82菌株的生长速率与hrpB基因突变株Po82ΔhrpB基本一致;而在营养贫瘠型培养基中,hrpB基因突变株Po82ΔhrpB的生长速率比野生型青枯菌Po82菌株快,这与Kanda等[24]研究结果不一致,而出现这种生长速率的差异尚未见报道,目前,还不清楚hrpB基因突变后青枯菌Po82菌株的代谢是否发生变化,因此,需进行下一步试验来探究其出现差异的原因。
在自然条件下,青枯菌的侵染过程包括识别、吸附、侵入、定殖、扩展和显症等步骤。伤根浇注菌液的接种方法包括以上全部过程被认为最接近于青枯菌自然发病情况;而茎部注射接种方法缺少识别、吸附、侵入过程,仅存在病原菌侵入以后的定殖、扩张及显症步骤。因此,运动性在青枯菌侵染初期具有重要的作用,而对于青枯菌侵入植物以后的致病过程不起作用。Vasse等[28]研究结果表明,青枯菌GMI1000菌株hrpB基因缺失突变株在侵染植物过程中的侵入、定殖和扩展的能力有所下降,而识别和吸附的过程与野生型没有差异。本试验对于野生型菌株和突变菌株的运动性试验表明,hrpB基因对青枯菌Po82菌株的运动性没有影响,与上述结论相一致。
hrpB基因不仅激发hrp基因簇中参与形成Ⅲ型分泌系统装置蛋白编码基因的表达,同时正调控依赖T3SS分泌系统泌出的效应蛋白,因此,在鉴定新的Ⅲ型效应子过程中,hrpB基因突变株的构建是至关重要的步骤。Cunnac等[29]在构建了青枯菌GMI1000菌株的hrpB基因突变株的基础上,鉴定出了48个新的依赖于hrpB基因的Ⅲ型效应子。因此,青枯菌Po82菌株hrpB基因缺失突变株的构建及其相关生物学功能的分析对进一步发掘鉴定Po82菌株中新的Ⅲ型效应子,进而深入解析其致病力进化的分子机理具有重要的作用。
[1]Hayward A C.Ralstoniasolanacearum[M]∥Lederberg J,ed.Encyclopedia of microbiology.San Diego:Academic Press,2000:32-42.
[2]Poueymiro M,Genin S.Secreted proteins fromRalstoniaso-lanacearum:a hundred tricks to kill a plant[J].Current Opinion in Microbiology,2009,12:44-52.
[3]Remigi P,Anisimova M,Guidot A,et al.Functional diversification of the GALA typeⅢeffector family contributes toRalstoniasolanacearumadaptation on different plant hosts[J].New Phytologist,2011,192:976-987.
[4]Cunnac S,Boucher C,Genin S.Characterization of thecis-acting regulatory element controllinghrpB-mediated activation of the typeⅢsecretion system and effector genes inRalstoniasolanacearum[J].Journal of Bacteriology,2004,186:2309-2318.
[5]Occhialini A,Cunnac S,Reymond N,et al.Genome-wide analysis of gene expression inRalstoniasolanacearumreveals that thehrpBgene acts as a regulatory switch controlling multiple virulence pathways[J].Molecular Plant-Microbe Interactions,2005,18:938-949.
[6]Aldon D,Brito B,Boucher C,et al.A bacterial sensor of plant cell contact controls the transcriptional induction ofRalstoniasolanacearumpathogenicity genes[J].The EMBO Journal,2000,19:2304-2314.
[7]Brito B,Aldon D,Barberis P,et al.A signal transfer system through three compartments transduces the plant cell contact-dependent signal controllingRalstoniasolanacearumhrpgenes[J].Molecular Plant-Microbe Interactions,2002,15:109-119.
[8]Brito B,Marenda M,Barberis P,et al.prhJandhrpGtwo new components of the plant signal-dependent regulatory cascade controlled by PrhA inRalstoniasolanacearum[J].Molecular Microbiology,1999,31:237-252.
[9]Genin S,Gough C L,Zischek C,et al.Evidence that thehrpBgene encodes a positive regulator of pathogenicity genes fromPseudomonassolanacearum[J]. Molecular Microbiology,1992,6:3065-3076.
[10]Valls M,Genin S,Boucher C.Integrated regulation of the typeⅢsecretion system and other virulence determinants inRalstoniasolanacearum[J].PLoS Pathogens,2006,2(8):798-807.
[11]Marenda M,Brito B,Callard D,et al.PrhA controls a novel regulatory pathway required for the specific induction ofRalstoniasolanacearumhrpgenes in the presence of plant cells[J].Molecular Microbiology,1998,27:437-453.
[12]Prior P,Fegan M.Recent development in the phylogeny and classification ofRalstoniasolanacearum[J].Acta Horticulturae,2005,695:127-136.
[13]Xu J,Pan Z C,Prior P,et al.Genetic diversity ofRalstonia solanacearumstrains from China[J].European Journal of Plant Pathology,2009,125:641-653.
[14]Wicker E,Grassart L,Coranson-Beaudu R,et al.Ralstonia solanacearumstrains from martinique (French West Indies)exhibiting a new pathogenic potential[J].Applied and Environmental Microbiology,2007,71:6790-6801.
[15]Xu J,Zheng H J,Liu L,et al.Complete genome sequence of the plant pathogenRalstoniasolanacearumstrain Po82[J].Journal of Bacteriology,2011,193:4261-4262.
[16]Schafer A A,Tauch W J,Kalinowsski G,et al.Small mobiliz-able multi-purpose cloning vectors derived fromEscherichiacoliplasmids pK18and pK19:selection of defined deletions in the chromosome ofCorynebacteriumglutamicum[J].Gene,1994,145:69-73.
[17]Block A,Guo M,Li G Y,et al.ThePseudomonassyringaetypeⅢfactor HopG1targets mitochondria,alters plant development,and suppresses plant innate immunity[J].Cell Microbiology,2010,12:318-330.
[18]Boucher C A,Barberis P A,Trigaret P A,et al.Transponson mutagenesis ofPseudomonassolanacearum:isolation of Tn5-induced avirulent mutants[J].Journal of General Microbiology,1985,131:2449-2457.
[19]Ausubel F M,Brent R,Kingston R E,et al.Short protocols in molecular biology[M].3rd ed.John Wiley and Sons,Chichester,1998.
[20]Lavie M,Shillington E,Eguiluz C,et al.PoP1,a new member of the YopJ/AvrRxv family of typeⅢeffector proteins,acts as a host specificity factor and modulates aggressiveness ofRalstoniasolanacearum[J].Molecular Plant Microbe Interactions,2002,15(10):1058-1068.
[21]张丽勍,许景升,徐进,等.植物青枯菌Ⅵ型分泌系统核心基因vasK突变株的构建及其致病性的测定[J].植物保护,2011,37(4):33-37.
[22]He L Y,Sequeira L,Kelman A.Characteristics of strains ofPseudomonassolanacearum[J].Plant Disease,1983,67:1357-1361.
[23]Meng F,Yao J,Allen C.A MotN mutant ofRalstoniasolanacearumis hypermotile and has reduced virulence[J].Journal of Bacteriology,2011,193(10):2477-2486.
[24]Kanda A,Ohnishi S,Tomiyama H,et al.TypeⅢsecretion machinery-deficient mutants ofRalstoniasolanacearumlose their ability to colonize resulting in loss of pathogenicity[J].Journal of General Plant Pathology,2003,69:250-257.
[25]Kelman A.The role of motility and aerotaxis in the selective increase of avirulent bacteria in still broth cultures ofPseudomanassolanacearum[J].Journal of General Microbiology,1990,76:177-188.
[26]徐进,陈林,许景升,等.香蕉细菌性枯萎病在中国的潜在适生区域[J].植物保护学报,2008,35(3):233-238.
[27]刘之洋,邹丽芳,邹华松,等.水稻条斑病菌hrpB1基因决定寄主致病性和非寄主过敏反应的功能研究[J].植物病理学报,2010,40(6):579-586.
[28]Vasse J,Genin S,Frey P,et al.ThehrpBandhrpGregulatory genes ofRalstoniasolanacearumare required for different stages of the tomato root infection process[J].Molecular Plant-Microbe Interactions,2000,13:259-267.
[29]Cunnac S,Occhialini A,Barberis P,et al.Inventory and functional analysis of the large Hrp regulon inRalstoniasolanacearum:identification of novel effector proteins translocated to plant host cells through the typeⅢsecretion system[J].Molecular Microbiology,2004,53(1):115-128.
The role of typeⅢsecretion system regulatorhrpBgene inRalstoniasolanacearumstrain Po82
Liu Lei, Xu Jin, Xu Jingsheng, Chen Kuangyu, Zhang Liqing, Zhang Hao, Feng Jie
(StateKeyLaboratoryforBiologyofPlantDiseaseandInsectPests,InstituteofPlantProtection,ChineseAcademyofAgriculturalSciences,Beijing100193,China)
Ralstoniasolanacearumstrain Po82,apathogenic variation strain,possesses pathogenic traits of both NPB andMokostrains.hrpBgene encodes a positive regulator and controls the expression of the typeⅢsecretion system (T3SS)and pathogenicity effectors transiting through this pathway.The mutant ofhrpBgene was constructed by using homologous recombination,and named Po82ΔhrpB.Based on the mutant strain,the complement strain was also constructed.The pathogenicity and the biological function of the wild type,the mutant strain and the complement strain were tested.Pathogenicity test showed that disease index of the mutant Po82ΔhrpBwas decreased compared with that of the wild type Po82.Growth curve analysis indicated that Po82ΔhrpBmutant grew as fast as the Po82strain in rich medium,whereas,in Boucher’s minimal medium,Po82ΔhrpBmutant grew faster than the Po82.There were no significant differences between the mutants and wild type strain in the ability of mobility.ThehrpBgene was a critical factor in pathogenicity ofR.solanacearumand played an important role in identifying new typeⅢeffectors and analyzing the molecular mechanism of pathogenicity variation.
Ralstoniasolanacearum; typeⅢsecretion system;hrpBgene; pathogenicity
Q 753
A
10.3969/j.issn.0529-1542.2012.05.002
2011-12-14
2012-01-18
国家重点基础研究发展计划(“973”)项目(2009CB119200);国家高技术研究与发展计划(“863”)项目(2012AA101501);国家自然科学基金(31101409,31272008)
* 通信作者E-mail:jfeng@ippcaas.cn

