贾鲁河水体微生物菌群结构季节动态变迁研究
王风芹,侯淑芬,谢 慧,于鲁冀,宋安东
(1.河南农业大学生命科学学院,河南郑州450002;2.农业部农业微生物酶工程重点实验室,河南郑州450002;3.郑州大学水利与环境学院,河南郑州450002)
贾鲁河是淮河的二级支流,发源于河南省新密市白寨乡圣水峪,流经郑州、中牟、开封、尉氏县,于周口市入沙颍河,全长276.5 km,总流域面积5 896 km2[1].贾鲁河无清水来源,沿途又接纳郑州市和中牟县的生活污水及工农业废水,造成其水体污染严重,自净能力差,水质属劣Ⅴ类,氨氮及COD的排放量占沙颖河的1/3,已成为沙颍河流域污染最严重的河流,被国家和河南省政府列为流域环境综合重点整治区域[2,3].微生物作为水质评价的一个重要指标,能及时反映整个水生环境的变化[4],对河流水体中的脱氮微生物数量进行检测,可以间接反映该水域的脱氮能力与自净能力.本研究利用传统的微生物分离计数方法和DGGE技术研究了贾鲁河水体及底泥微生物种群结构与多样性的季节动态变迁特征,以期为制定贾鲁河污水的生物治理方案提供理论依据.
1 材料与方法
1.1 样品采集
选取贾鲁河郑州示范工程第七标段上游和下游作为2个取样点,于2009-03—2009-12的春分、夏至、秋分、冬至4个时间点,参照文献[5]的方法,分别取河流水体(距水表面0~40 cm,5点取样混合均匀)和底泥(0~20 cm,5点取样混合均匀)以备试验.
1.2 微生物区系分析
1.2.1 细菌、真菌和放线菌数量测定 采用梯度稀释平板法测定水样和底泥样品中的细菌、真菌和放线菌数量.培养基分别选用牛肉膏蛋白胨培养基、马丁氏培养基和高氏一号培养基,于28~30℃,分别培养2~3 d、3~5 d和5~7 d,计数并记录结果.
1.2.2 脱氮功能微生物数量测定 采用最大或然数法(Most Probable Number,MPN)测定硝化细菌和反硝化细菌等脱氮功能微生物菌数.培养基分别采用硝化细菌培养基和反硝化细菌培养基.样品稀释梯度为 10-2,10-3,10-4,10-54 个梯度,每个梯度3个重复,接种量0.1 mL,并以不接菌的培养基为空白对照,28~30℃静置培养14 d后进行测定,培养基配方及试验方法参见文献[6],查MPN表,计算.
1.3 16S rDNA PCR-DGGE 分析
1.3.1 16S rDNA V3 区 PCR 扩增 采用 E.Z.N.A.TM Water/Soil DNA Kit Protocal(OMEGA)试剂盒提取水样及底泥的宏基因组DNA,提取方法参照说明书.选用细菌的通用引物F341-GC(5'-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGCCTACGGGAGGCAGCAG-3')和R518(5'-ATTACCGCGGCTGCTGG-3')[7~9]对水样和底泥样品的16s rDNA V3区进行PCR扩增.PCR扩增的反应体系如下:5 μL 10 × PCR Buffer(Mg2+15 mmol·L-1)、4 μL dNTP Mix(2.5 mmol·L-1)、1 μL 引物 F341-GC(10 μmol·L-1)、1 μL 引物 518 R(10 μmol·L-1)、0.5 μL Taq DNA 聚合酶、DNA模板10 ng,灭菌去离子水补至50 μL.PCR反应条件为:95℃下预变性3 min;94℃下变性30 s,56℃下退火30 s,72℃下延伸45 s,28个循环;94℃下30 s,56 ℃下30 s,72 ℃下延伸7 min.PCR 扩增产物经质量分数为1.0% 琼脂糖凝胶检测后,于-20℃保存备用.
1.3.2 DGGE 将45 μL 的 PCR 产物和5 μL 6 倍加样缓冲液混合,采用Bio-rad凝胶电泳系统,质量分数10%的聚丙烯酰胺凝胶,变性剂浓度为30%~60%(100%的变性剂含7 mol·L-1尿素和40%去离子甲酰胺),在200 V的电压下,电泳10 min,使样品快速跑出泳道后再在85 V电压下,60℃,电泳14 h.电泳结束后,采用SYBR GreenⅠ染色,获得DGGE指纹图谱.
1.3.3 目的条带的克隆与测序 分别切取DGGE图谱中的目的条带至不同的EP管中,以100 μL 70%的冰乙醇洗涤,重复2~3次,风干10 min后转移至另一新的 EP管中,加50 μL灭菌去离子水,4℃静置过夜;以此为模板,以 F341(5'-CCTACGGGAGGCAGCAG-3')和 R518为引物进行PCR扩增,其PCR反应体系和扩增程序同1.3.1.PCR产物经凝胶检测后,送北京鼎国生物技术有限公司测序.将测序结果进行处理后提交到GeneBank数据库,采用BLAST进行目标序列和基因库中所含序列的相似性分析,得到同源性最近的序列.
2 结果与分析
2.1 贾鲁河不同季节水体微生物数量分布
贾鲁河郑州段示范工程第七标段上下游水体不同季节各类群微生物数量统计见表1.由表1可知,贾鲁河水体中细菌数量最多,真菌次之,放线菌最少;上下游水体相比而言,细菌、放线菌和真菌数量未呈现规律性变化;硝化细菌和反硝化细菌数量基本相当,且下游数量略高于上游.贾鲁河水体中各类群微生物夏秋季数量高于冬春季.
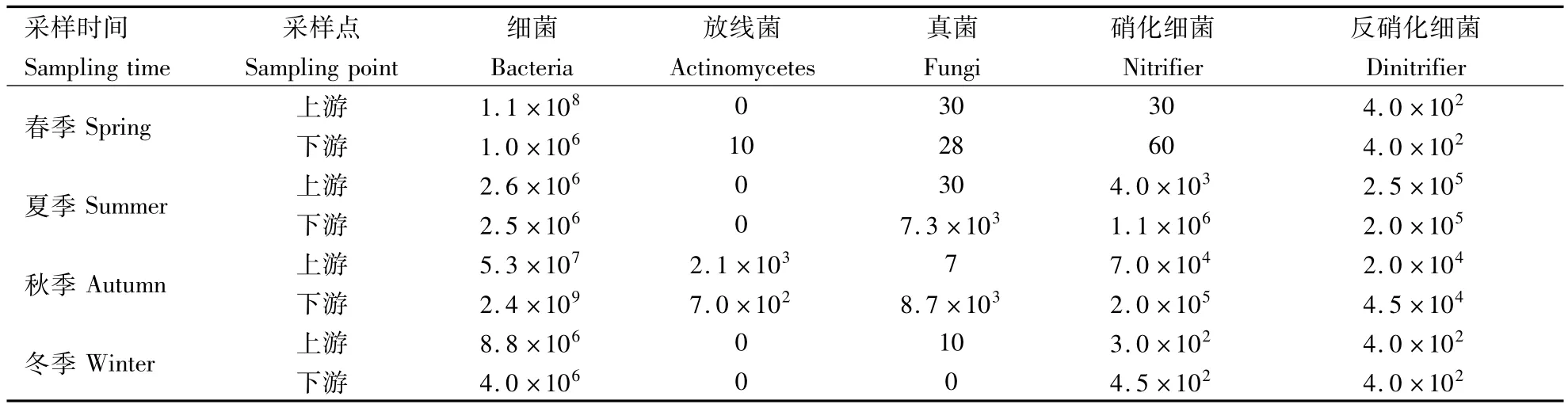
表1 不同季节水体微生物数量Table 1 Microbe population in the water column with various seasons CFU·mL-1
2.2 贾鲁河不同季节底泥微生物数量分布
贾鲁河不同季节上下游底泥中微生物数量分布情况见表2.由表2可知,底泥中细菌数量最高,其次是放线菌,真菌数量最少;硝化细菌和反硝化细菌数量基本相当.除冬季外,下游底泥中细菌数量显著高于上游数量;放线菌和真菌上下游数量之间差别不明显;夏秋季下游脱氮功能微生物数量显著高于上游,冬春季差别不明显.细菌、真菌和放线菌数量春夏季均略高于秋冬季,硝化细菌和反硝化细菌数量夏秋季高于冬春季.底泥中微生物数量季节变化较水体中微生物数量季节变化稳定.
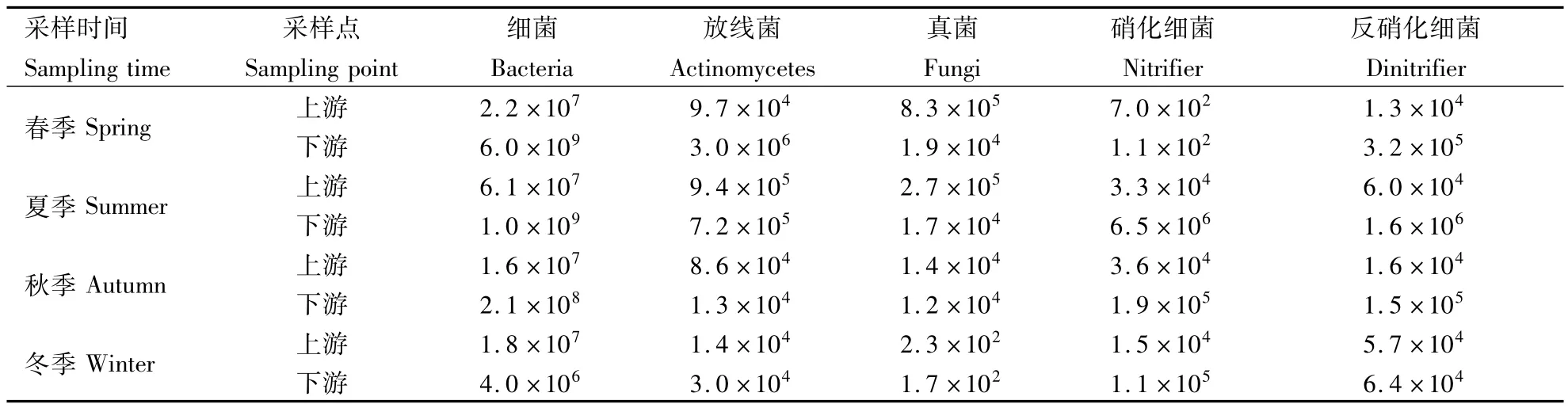
表2 不同季节底泥微生物数量Table 2 Microbe population in the sediment with various seasons CFU·g-1
2.3 底泥样品中细菌16S rDNA DGGE分析
2.3.1 16S rDNA DGGE指纹图谱分析 扩增底泥样品中细菌16S rDNA的V3区进行DGGE指纹图谱分析,明显可见各样品数量不等、强度不同的条带(图1),春、夏、秋、冬四季微生物种群结构差异显著,上下游底泥样品在春、夏、秋、冬四季优势菌群均存在明显的消涨变迁.
2.3.2 优势菌群鉴定结果 将DGGE图谱中的5条优势目的条带(图1)回收、纯化、测序,测得的16S rDNA V3区序列与GeneBank数据库中的已知序列用BLAST进行检索和同源性比较,结果见表3.
从表3可知,5个条带中有4条在GenBank中找到与其同源性很高(>98%)的相似种群.其中条带1和条带2为不可培养的未知种群细菌.条带3与弓形菌属(Arcobacter)亲缘关系最近,同源性达100%.条带4和条带5均为拟杆菌(Bacteroidetes),该种群微生物是活性污泥和好氧颗粒污泥系统中的优势菌群,在贾鲁河水体自净过程中可能起到重要作用.

图1 贾鲁河不同季节总细菌DGGE图谱Fig.1 DGGE profile of total bacteria in Jalu River various seasons

表3 DGGE优势条带的16S rDNA片段序列的比对结果Table 3 Comparison of 16S rDNA sequences in dominant DGGE bands by sequencing and BLAST analysis
3 结论与讨论
微生物监测是利用微生物资源对环境污染或变化所发生的反应,阐明环境污染状况,从生物学角度为环境质量的监测和评价提供依据.它不仅能反映多种因子污染的综合效应,还能反映环境污染的历史状况,成为污染物的暴露与效应最灵敏的监测指标,这对于环境污染的探查和快速筛选、环境健康的风险评价及污染物长期毒性效应的早期预报具有重要意义.
本研究发现贾鲁河水体和底泥中细菌数量显著高于真菌和放线菌数量,脱氮功能微生物硝化细菌和反硝化细菌数量相当;水体中各类群微生物数量及底泥中硝化细菌和反硝化细菌数量夏秋季高于冬春季,而底泥中细菌、真菌和放线菌数量春夏季略高于秋冬季.无论在水体还是底泥中,细菌总数在一年四季均达到了106CFU·mL-1以上,最高达到了109CFU·mL-1,表明贾鲁河水质一年四季均处于严重污染状态,为多污染带.这与贾鲁河暗渠多,大量生活污水和生产废水废物不断排入关系巨大.
吴春笃等[10]对镇江城市污水细菌多样性及其生物安全性研究时发现,近似弓形杆菌属(Arcobacter)的细菌比例高达74.2%,为城市污水中的主要优势菌群,进一步将其23S rDNA上的特异性序列进行PCR扩增后证实污水中的弓形菌属为致病性弓形杆菌.本研究利用16S rDNA PCR-DGGE指纹图谱分析,揭示出贾鲁河底泥中四季微生物种群结构差异显著,上下游底泥样品在春、夏、秋、冬四季优势菌群均存在明显的消涨变迁,且拟杆菌和弓形菌等是贾鲁河底泥的优势微生物.弓形菌等致病性细菌的存在将威胁周边群众及畜禽的健康,因此对于贾鲁河的污染治理应引起政府和科研部门的极大关注,制定相应的治理措施.
[1] 卢 暇,苏卫涛,王 楠.贾鲁河河道洪水计算[J].河南水利与南水北调,2009(8):54.
[2] 赵 辉,张建夫,谢东坡,等.贾鲁河水质的变化规律研究[J].周口师范学院学报,2005,22(5):68-70.
[3] 高红莉,李洪涛,赵凤兰.沙颍河(河南段)水污染的时空分布规律[J].水资源保护,2010,26(3):23-26.
[4] 陈国新.环境科学基础[M].上海:复旦大学出版社,1992:259-261.
[5] 国家环境保护总局.水和废水监测分析方法[M].北京:中国环境科学出版社,2002:38-42.
[6] 李振高,骆永明,滕 应.土壤与环境微生物研究法[M].北京:科学出版社,2008:92-103.
[7] 刘恩科,赵秉强,李秀英,等.不同施肥制度土壤微生物量碳氮变化及细菌群落16S rDNA V3片段PCR产物的DGGE 分析[J].生态学报,2007,27(3):1079-1085.
[8] NAKATSU C H,TORSVIK V,ØVREÁS L.Soil community analysis using DGGE of 16S rDNA polymerase chain reaction products[J].Soil Science Society of A-merica Journal,2000,64:1382-1388.
[9] MUYZER G,WAAL E C,UITTERLINDEN A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J].Applied and Environmental Microbiology,1993,59(3):695-700.
[10] 吴春笃,许小红,宁德刚,等.城市污水细菌多样性及其生物安全性研究[J].中国安全科学学报,2008,18(1):119-122.

