Lenzites gibbosa锰过氧化物酶1基因启动子序列的克隆1)
吴书景 池玉杰 闫洪波
(东北林业大学,哈尔滨,150040)
锰过氧化物酶(MnP;EC1.11.1.13)是一种依赖H2O2的含Fe3+的血红素五聚体糖蛋白酶,同时也是一种常见的降解木质素的过氧化物酶,广泛存在于白腐菌中,在真菌降解木质素的非特异性细胞外氧化还原酶中起着至关重要的作用。由于MnP对酚类和非酚类木质素和多环芳烃等多种异生物质都具有独特的降解能力,它能够有效地降解废水和土壤中很难被降解的多氯联苯、多环芳烃、DDT、染料、炸药和其它氯化物等,因此这种酶在生物制浆、污水处理等生物技术应用中具有重要意义。Lenzittes gibbosa是东北地区常见的一种白腐菌,也是一种生长速度较快、对木材和木质素分解能力较强的白腐菌,笔者及其小组以前的研究表明这种白腐菌能够产生锰过氧化物酶和漆酶[1],并首先克隆到了该菌种的MnP基因[2]。笔者根据本研究小组已克隆到的L.gibbosa MnP1 cDNA全长基因Lg-mnp1,利用染色体步移法首先克隆到了Lg-mnp1上游的启动子序列。对L.gibbosa MnP1基因Lg-mnp1启动子的克隆有助于进一步深入研究mnp表达调控的关系,同时也为外源基因表达载体系统的构建提供了重要依据。
1 材料与方法
1.1 菌种来源与培养
白腐菌(Lenzittes gibbosa)菌株CB1采自长白山,自行分离得到,保存在东北林业大学森林保护学科森林病虫病理实验室。试验前在PDA斜面培养基上培育的菌种冷藏于4℃冰箱。挑取小块试管内的菌种,接种于PDA平板培养基上,于28℃下培养7 d。
1.2 基因组DNA的提取
取长势较旺的一皿菌丝体,刮取新鲜菌丝,使用TIANGEN公司的 DNAquick_快捷型植物基因组DNA提取系统及相关方法提取真菌基因组DNA。将所得DNA取2 μL用1.0%琼脂糖凝胶电泳检测DNA的纯度和大小,然后将DNA稀释到终浓度为10~100 mg·L-1用于PCR反应。
1.3 Lg-mnp1启动子区域扩增
根据已克隆到的L.gibbosa MnP1 cDNA全长基因Lg-mnp1,在 5'端设计了3个巢式特异性引物SP1、SP2和SP3,设计方向为需要扩增的未知区域方向,SP2在SP1的上游,SP3位于SP2的上游。其中,SP1:5'-CGCAGCGTTCGTAGTGGTCT-3';SP2:5'-GCGAGGAGAGAAACGAAGGAAG-3';SP3:5'-ATTGTCGTTGTCTGATGAGGGATG-3'。利用染色体步移技术(Genome Walking Kit试剂盒,TaKaRa)克隆其上游的启动子序列。PCR的扩增体系、温度条件和循环数见表1。扩增完毕后,取第二、三轮PCR反应液各2 μL,使用1%琼脂糖凝胶进行电泳,观察电泳图,确定目的条带。使用Gel Extraction Kit(E.Z.N.A)试剂盒回收PCR产物的目的片段,再将DNA片段连接到 PMD20-T载体(TaKaRa)并转化至Top10感受态细胞中,所得菌液交北京英骏公司测序。将测定的序列去除载体后,与Lg-mnp1编码区原始序列进行拼接比对,确定为编码区上游序列。采用 Signal Scan(http://www-bimas.cit.nih.gov/molbio/signal/)对克隆到的Lg-mnp1启动子序列进行分析[3-4],确定其中的启动子元件及核心区域。
2 结果与分析
2.1 L.gibbosa MnP1基因Lg-mnp1的启动子序列与分析
筛选第2轮和第3轮PCR产物的目的片段,测序后得到1 568 bp的序列如图1所示,与Lg-mnp1编码区原始序列进行拼接比对,结果有90 bp的重叠,表明所得序列即为L.gibbosa MnP1基因Lgmnp1的启动子序列。将该启动子序列递交Gen-Bank,登录号为 HM369808。

表1 染色体步移PCR的扩增体系、温度条件和循环数
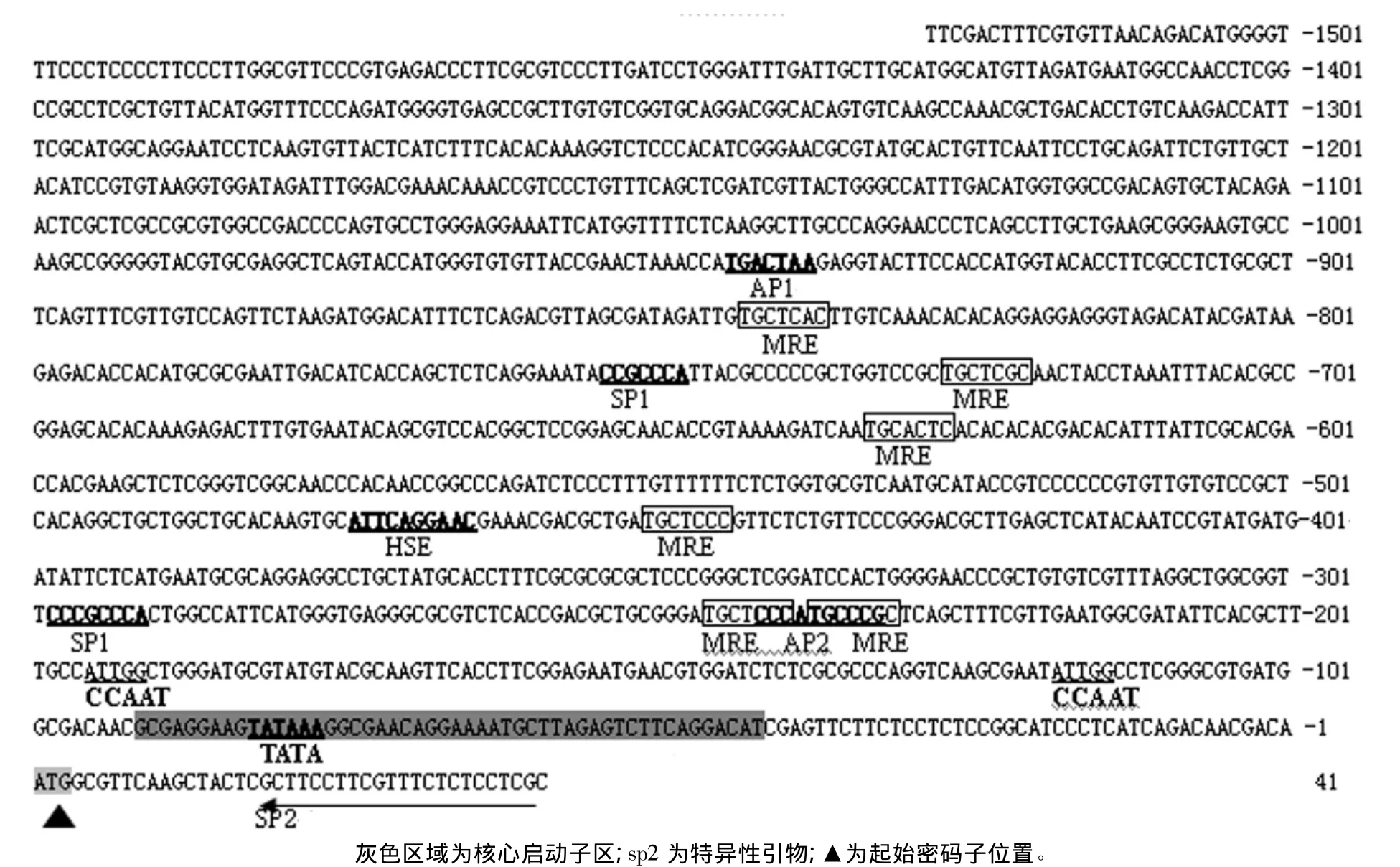
图1 L.gibbosa MnP1基因Lg-mnp1的启动子序列
启动子序列结构分析结果表明,Lg-mnp1 5'端上游包含了真核生物启动子的基本转录调控元件,见表2及图1。在起始密码子上游-83 bp处有1个TATA-box,-119、-196 bp有 2 个反向 CCAAT-box(ATTGG)。-92~43 bp为Lg-mnp1启动子的基础启动子区;在-52 bp处有一个转录起始位点。也存在多个顺式作用元件,包括SP-1(GGGCGG)、激活子蛋白AP-2位点(CCCMNSSS)、热激元件HSE(5'-NTTCNNGAAN-3')[5]及6个推定的金属响应MREs(TGCRCNC)[6]等。
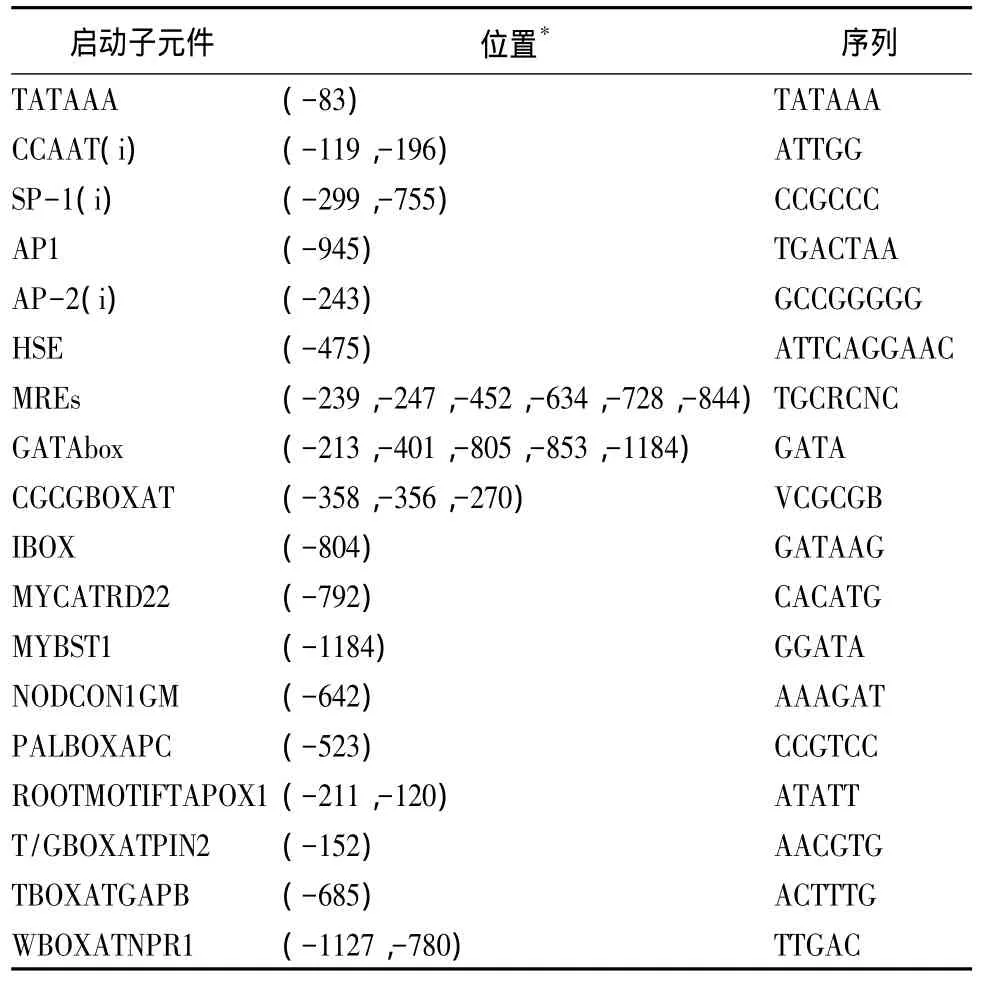
表2 Lg-mnp1启动子包含的作用元件
2.2 白腐菌mnp基因启动子元件的比较
将克隆到的白腐菌L.gibbosa MnP1基因Lgmnp1启动子序列与前人研究的8种白腐菌16个mnp启动子元件进行了比较,结果见图2。从图2可知,尽管Lg-mnp1的调控元件与其他记载的白腐菌mnp有很多相似之处,但元件分布、数目及方向都有所不同。有些菌株mnp启动子不包含MRE(如Lentinula edodes[7]),而有些菌株 mnp 启动子存在正向的CCAAT-box。
3 结论与讨论
本研究的白腐菌(Lenzites gibbosa)与偏肿栓菌(Trametes gibbosa)是同物异名,起初Persoon将其命名为偏肿迷孔菌(Daedalea gibbosa Pers.),后来由Fries转属作为 Trametes gibbosa(Pers.ex Fr.)Fr.,该命名后来一直被沿用。1939年,日本京都帝国大学农学部植物病理学研究室的逸見武雄(Hemmi Takewo)曾根据其子实体的形态特征,将其重新转属命名为 Lenzites gibbosa(Pers.)Hemmi[19],但并未被采用。Tomšovský等[20]对其进行系统发育分析后,发现其与Lenzites属的系统学关系较近,遂认定Lenzites gibbosa为该种的合法名称。由于该种已经转属到革裥菌属,可将其中文名改为偏肿革裥菌,偏肿栓菌作为同物异名。

图2 16个白腐菌mnp基因启动子元件的比较
利用染色体步移法克隆了偏肿革裥菌Lg-mnp1启动子序列,序列全长1 568 bp。经启动子在线分析软件分析可知,该5'端转录调控区序列包含了真核生物启动子的基本转录调控元件:1个TATA-box和2个反向的CCAAT-box(ATTGG),以及多个重要顺式作用元件,包括转录因子SP-1、AP-2、热诱导因子HSE、金属响应因子MRE以及氮因子结合元件(GATA)等,这表明偏肿栓菌MnP在转录水平可能受氮源、锰以及热休克调控,进一步的证据有待于对其5'端调控区域进行缺失分析研究。因此,对偏肿革裥菌Lg-mnp1启动子的克隆,可为进一步揭示mnp表达调控的分子机制提供启动子序列基础。再者,在构建外源表达载体时,所应用的可以是质粒中的启动子序列,也可以是自我基因的启动子序列,这可根据试验对比的结果筛选哪一个启动子序列更有利于该基因的外源表达。因此,对偏肿革裥菌L.gibbosa的锰过氧化物酶基因启动子序列的克隆,也为其外源基因表达载体系统的构建提供了基础依据。
[1]张玉龙,池玉杰,闫洪波.偏肿栓菌产锰过氧化物酶条件优化[J].林业科学,2011,47(8):88-94.
[2]闫洪波.偏肿拟栓菌锰过氧化物酶cDNA基因克隆及在毕赤酵母中的表达[D].东北林业大学,2009.
[3]Liu Yaoguang,Chen Yuanling.High-efficiency thermal asymmetric interlaced PCR for amplification of unknown flanking sequences[J].BioTechniques,2007,43(5):649-656.
[4]Wang Shiming,He Jian,Cui Zhongli,et al.Self-formed adaptor PCR:a simple and efficient method for chromosome walking[J].Applied and environmental microbiology,2007,73(15):5048-5051.
[5]Bonner J J,Ballou C,Fackenthal D L.Interactions between DNA-bound trimers of the yeast heat shock factor[J].Mol Cell Biol,1994,14(1):501-508.
[6]Culotta V C,Hamer D H.Fine mapping of a mouse metallothionein gene metal response element[J].Mol Cell Biol,1989,9(3):1376-1380.
[7]Nagai M,Sakamoto Y,Nakade K,et al.Isolation and characterization of the gene encoding a manganese peroxidase from Lentinula edodes[J].Mycoscience,2007,48:125-130.
[8]Alic M,Akileswaran L,Gold M H.Characterization of the gene encoding manganese peroxidase isozyme 3 from Phanerochaete chrysosporium[J].Biochimica et Biophysica Acta,1997,1338(1):1-7.
[9]Godfrey B J,Mayfield M B,Brown J A,et al.Characterization of a gene encoding a manganese peroxidase from Pkanerochaete chrysosporium[J].Gene,1990,93(1):119-124.
[10]Mayfield M B,Godfrey B J,Gold M H.Characterization of the MnP2 gene encoding manganese peroxidase isozyme 2 from the basidiomycete Phanerochaete chrysosporium[J].Gene,1994,142(2):231-235.
[11]Lobos S,Larrondo L,Salas L,et al.Cloning and molecular analysis of a cDNA and the Cs-MnP1 gene encoding a manganese peroxidase isoenzyme from the lignin-degrading basidiomycete Ceriporiopsis subvermispora[J].Gene,1998,206(2):185-193.
[12]Tello M,Corsini G,Larrondo L F,et al.Characterization of three new manganese peroxidase genes from the ligninolytic basidiomycete Ceriporiopsis subvermispora[J].Biochimica et Biophysica Acta,2000,1490(1/2):137-144.
[13]Li D,Li N,Mayfield M B,et al.Characterization of genes encoding two manganese peroxidases from the lignin-degrading fungus Dichomitus squalens[J].Biochimica et Biophysica Acta,1999,1434(2):356-364.
[14]Ruiz-Dueñas F J,Martínez M J,Martínez A T.Molecular characterization of a novel peroxidase isolated from the ligninolytic fungus Pleurotus eryngii[J].Molecular Microbiology,1999,31(1):223-235.
[15]Asada Y,Watanabe A,Irie T,et al.Structures of genomic and complementary DNAs coding for Pleurotus ostreatus manganese(II)peroxidase[J].Biochimica et Biophysica Acta,1995,1251(2):205-209.
[16]Giardina P,Palmieri G,Fontanella B,et al.Manganese peroxidase isoenzymes produced by Pleurotus ostreatus grown on wood sawdust[J].Archives of Biochemistry and Biophysics,2000,376(1):171-179.
[17]Hildén K,Martinez A T,Hatakka A,et al.The two manganese peroxidases Pr-MnP2 and Pr-MnP3 of Phlebia radiata,a lignindegrading basidiomycete,are phylogenetically and structurally divergent[J].Fungal Genetics and Biology,2005,42(5):403-419.
[18]Johansson T,Nyman P O.A cluster of genes encoding major isozymes of lignin peroxidase and manganese peroxidase from the white-rot fungus Trametes versicolor[J].Gene,1996,170(1):31-38.
[19]Hemmi T,Ikeya J.Studies on Lensites gibbosa(Pers.)Hemmi n.comb.causing wood-rot of deciduous trees[J].Japanese Journal of Phytopathology,1939,9(1):1-15.
[20]Tomšovský M,Kolaǐík M,Pažoutová S,et al.Molecular phylogeny of European Trametes(Basidiomycetes,Polyporales)species based on LSU and ITS(nrDNA)sequences[J].Nova Hedwigia,2006,82(3/4):269-280.

