湖南省猪源粪肠球菌AFLP分析
田世成,黎满香,蒋 伟,林荣高,郭洪香,屈泰龙
(湖南农业大学动物医学院,湖南长沙 410128)
粪肠球菌是栖居于人类或动物胃肠道的正常菌群,也可定植于口腔和阴道,且广泛分布于自然界中。它们能获得特定的基因决定簇,比如毒力因子和耐药性决定簇,使其适应各种生态系的能力增强[1]。目前,对粪肠球菌的种群结构和全球流行病学了解还是非常有限[2],特别是对动物来源菌株。在兽医临床中近年来不断有动物感染的报道[3-4],且肠球菌属在兽医临床标本中的分离率也不断增加。目前,对肠球菌的鉴定仍依靠传统表型特征的检测作为重要的鉴定方法。但由于许多肠球菌种间的表型特征非常接近,因此鉴定起来费时费力,以至错失治疗时机,使病情得不到及时的控制。不少的分子生物学技术已经被应用于粪肠球菌的流行病学分析[5],扩 增 片 段 长 度 多 态 性 (amplified fragment length polymorphism,AFLP)方法可以用来获得细菌基因型信息[6]。采用AFLP技术在链霉菌菌种鉴定上也是一种方便有效的工具[7]。本试验以从不同发病猪场分离出的42个分离株为研究对象,在表型鉴定符合粪肠球菌的基础上,进行了AFLP及系统进化分析研究,旨在对该菌进行分类分型,用以追溯传染源,了解湖南省猪场粪肠球菌引起感染的情况及主要基因类群的分布差异。
1 材料与方法
1.1 材料
1.1.1 菌株 试验所用菌株包括标准菌株ATCC29212,临床分离株42株,主要从病猪肺、脾、脑、淋巴结等处分离。其中长沙分离8株、永州7株、娄底5株、郴州4株、岳阳3株、益阳3株、邵阳3株、怀化2株、衡阳1株、株洲1株,还有5株不能确定到市级地区。
1.1.2 主要试剂EcoRⅠ、MseIw,购自加拿大Fermentas公司;T4DNA连接酶、TaqDNA聚合酶、dNTP、DL2000DNA 1adder、100bp DNA ladder,购自宝生物(大连)有限公司;琼脂糖为Sigma公司产品;其他常规试剂均为国产分析纯。
1.1.3 主要仪器 PCR扩增仪(ABI 2720型),美国ABI公司;DYY-6C型电泳仪、DYPZ-24E型电泳槽,北京六一仪器厂;5415D型台式高速离心机,德国eppendorf公司;AYJI-1002-U型超纯水仪,台湾艾科浦公司;SHA-B型水浴恒温震荡器,常州市华普达教学仪器有限公司。
1.1.4 培养基 TSB 培养基(Fluka)使用时按30g/L配制。
1.2 方法
1.2.1 引物及接头序列 接头序列根据基因组及酶切位点设计(EcoRⅠ接头,MseⅠ接头),引物参考文献[8],并由上海生工生物工程技术服务有限公司合成。EcoRⅠ 接头序列:5′-CTCGTAGACTGCGTACC-3′, 5′-CATCTGACGCATGGTTAA-3′;MseⅠ接头序列:5′-GACGATGAGTCCTGAG-3′,5′-TACTCAGGACTCAT-3′。EcoRⅠ PCR 扩增 引 物:5′-GACTGCGTACCAATTC-3′(核 心 序列),MseⅠ PCR 扩增引物:5′-GATGAGTCCTGAGTA-3′(核心序列)。引物组分别标记为EcoRⅠ+1/+2/+3,MseⅠ+1/+2/+3。
1.2.2 细菌基因组DNA的提取 取出保存菌种接种于TSB培养基中,37℃摇床培养12h,染色镜检后收集菌液备用。采用酚氯抽提法进行DNA提取[9]。
1.2.3 限制性酶切及连接 酶切使用双酶切法,EcoRⅠ(10U/μL),MseⅠ(10U/μL);37℃作用4h后转到65℃继续4h,95℃作用20min使酶失活,酶切后用10g/L琼脂糖凝胶电泳检验酶切效果。连接用T4DAN连接酶连接酶切产物与EcoRⅠ接头和MseⅠ接头;于16℃连接4h后再95℃作用10min失活T4 DNA连接酶,置-20℃保存备用。
1.2.4 PCR扩增及引物筛选 预扩增,使用引物组EcoRⅠ+1和MseⅠ+1预扩增,连接产物作模板,PCR体系50μL。置PCR仪中运行20个循环。选择性扩增,将预扩增产物稀释10倍作为模板,反应体系50μL,模板(稀释后预扩增产物),PCR采取梯度退火方法,即起始退火温度设为65℃,然后每2循环后退火温度降1℃,直到退火温度为55℃后不再降低退火温度。扩增产物用20g/L琼脂糖凝胶电泳检测,以确定是否扩增成功和聚丙烯酰胺电泳时上样的量。最佳引物的筛选,对所设计的引物进行组合,分别按照预扩增和选择性扩增进行筛选获得最佳的引物对。
1.2.5 聚丙烯酰胺凝胶电泳分析 凝胶电泳,80 g/L聚丙烯酰胺凝胶,每孔加8μL样品(6×上样缓冲液),同时每板加一孔5μL 100bp DNA标准;70 V电压电泳5h。硝酸银染色,显色清晰后加入纯净水终止;再漂洗1次,成像系统拍照。AFLP图谱的分析,在获得选择性扩增图谱后进行读带,有条带记录为“1”,无条带记录为“0”。然后用 NTSYSpc-2.10e中算术平均非加权对群法(UPGMA)进行类聚分析。
2 结果
2.1 预扩增
在经PCR扩增和琼脂糖凝胶电泳后出现AFLP应有的smear现象,即电泳出现拖尾、弥散现象(图1)。因此,可进行下一步选择性扩增。

图1 初步扩增结果Fig.1 The results of preliminary amplification(primer E+G/M+T)
2.2 选择性扩增
在琼脂糖凝胶电泳图谱(图2)中可见明亮的条带,说明选择性扩增成功,可以将样品进行80g/L聚丙烯酰胺凝胶电泳。
在80g/L聚丙烯酰胺凝胶电泳图谱(图3)中获取比琼脂糖更多条带的AFLP图谱。泳道8为100bp DNA标准分子量,其余各泳道对应不同的菌株(仅列部分,其余见图4)。
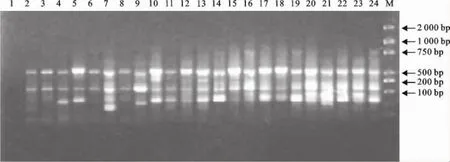
图2 选择性扩增电泳结果Fig.2 The results of selected amplification distinguished in agarose gel(primer E+GC M+TG)

图3 聚丙烯酰胺凝胶电泳结果Fig.3 The results of selected amplification distinguished in polyacrylamide gelatin(primer E+GC M+TG)
2.3 湖南粪肠球菌的AFLP聚类分析结果
由湖南猪源粪肠球菌AFLP聚类分析树状图上可知,所有42菌株可以分为3个大簇,定义为A、B、C。分离于关节的菌株主要分布于相似性约在63.0%的D、E两簇上,来自于长沙的CS64,CS81,CS86,CS76有着极相似的图谱,分布于D1簇。按照相似性为75.0%分类可以获得21种基因型,其中11种基因型且分离到一个菌株。
3 讨论
3.1 AFLP图谱的分型效果
本试验运用AFLP对湖南猪源粪肠球菌的DNA多态性进行分析。需要指出的是本试验在进行丙烯酰胺凝胶电泳时没有一块玻璃板能够同时装载42样个本,只能分装成3块同时配制相同规格的凝胶,使得3板间的DNA Marker条带略有偏差,因此在读带时做了相应的调整。试验结果表明湖南地区分离到的粪肠球菌在基因上存在多样性,从本次获得的AFLP图谱中相似性最小的仅有50%,可区分3个大簇A、B、C,按照相似性为75%分类可以获得21种基因型。根据以往的工作,应用16SrDNA序列描述菌株差异性,湖南分离株的同源性都大于98%,因此,来源于病猪的湖南分离株的16S rDNA变异并不大,在进化树上未能体现湖南分离株的地域性差异。由此可见,AFLP在描述种内菌株的差异,特别是检验地缘关系相近的分离株时有较大的优势。
3.2 关于引物的筛选及AFLP的条件
在进行多态性分析时,条带的数目要求适量,过多则会导致电泳时难分离开及统计分析困难,过少又不能反映菌株间的差异,因此调控图谱条带数目对于AFLP分析至关重要。假设基因组碱基A、G、T、C所占比例是一样的,那么在核心序列末端每多加上1个碱基,理论上扩增出的条带数目就会减少3/4。本试验设计的扩增引物的核心序列部分与接头序列互补配对,且分别设计了核心序列+1、+2、+3的多种引物组合,以寻求获取最佳AFLP图谱。试验结果表明在预扩增时使用引物E+G/M+T和选择性扩增时使用E+GC/M+TG获得的图谱最为理想,如果用核心序列+3则条带明显减少,不利分析。从这点看体现了AFLP技术在获得条带数目方面的灵活可调性。电泳电压对AFLP图谱也有很大影响,在试验中不同的电泳仪不同的样品都要求不同的电泳条件。本试验中电泳电压为70V时获得较理想的图谱。

图4 AFLP分析结果Fig.4 A dendrogram based on AFLP analysis generated by UPGMA
3.3 湖南分离株的AFLP图谱与表型的相关性
DNA指纹技术常被用于追踪传染源、调查传播途径、推断流行方式,其精确性是传统方法不能比拟的。有人通过母代家畜和后代家畜体内分离株的DNA指纹差异证明该微生物的传播方式为垂直传播[10]。本试验从AFLP图谱看,分离株CS64、CS81和CS86有着很近的亲缘关系,在此推断,这4株可能由同一菌株克隆而来,粪肠球菌可能在这一地区内猪场间相互传播。此外,还有YZ02、YZ03、CS85和CS13估计也是属于同一菌株克隆而来。毒力基因型asa1+EF0591-efaA+EF3314+esp+gelE+cylA+ace+的菌株比较集中于 A簇,而所获得AFLP图谱与耐药型菌株没有明显的相关性。研究表明AFLP图谱乃至其他DNA指纹技术都能很清晰描述菌株的图谱差异,但是否与表型相关则需要做表型试验进行对照,而且一种反应体系或条带图谱往往不能与多个表型表现相关。
本试验应用AFLP分析技术,再结合以往对该分离株初步鉴定,不但进一步确定了其分属不同的种,并了解不同地理种群已出现了分化。在此,我们的结果也很难与耐药性、毒力等相关表型信息同时关联,且要用AFLP图谱描述与各个表型的相关性还有待深入研究。
[1]Mundy L M,Sahm D F,Gilmore M.Relationships between enterococcal virulence and antimicrobial resistance[J].Clin Microbiol Rev,2000,3:513-522.
[2]Garbajosa P R,Bonten J M,Robinson D A,et al.Multilocus sequence typing scheme forEnterococcusfaecalisreveals hospital-adapted genetic complexes in a background of high rates of recombination[J].Clin Microbiol,2006,44:2220-2228.
[3]陈一资,蒋文灿,胡 滨.对鸭场爆发罕见粪肠球菌病的研究[J].中国兽医学报,2003,(23)4:324-325.
[4]王亚宾,胡清林,陈丽颖,等.一株致仔猪关节炎粪肠球菌的鉴定[J].动物医学进展,2010,31(S1):157-161.
[5]Waar K,Muscholl-Silberhorn A B,Willems R J,et al.Genogrouping and incidence of virulence factors ofEnterococcus faecalisin liver transplant patients differ from blood culture and fecal isolates[J].Infect Dis,2002,185:1121-1127.
[6]鲁辛辛,王 玫,周 宇.细菌核酸分类鉴定技术的研究进展[J].中华检验医学杂志,2004,27(6):394-397.
[7]Lanoota B,Vancanneyta M,Hostea B,et al.Phenotypic and genotypic characterization of mutants of the virginiamycin producing strain 899and its relatedness to the type strain ofStreptomycesvirginiae[J].Syst Appl Microbiol,2005,28:77-84.
[8]Woodford N,Adebiyi A M,Palepou M F,et al.Diversity of VanA glycopeptide resistance elements in enterococci from human and nonhuman sources[J].Antimicrob Agents Chemother,1998,42:502-508.
[9]李金钟.肠球菌分类与鉴定新进展[J].临床检验杂志,2006,24(3):228-230.
[10]Amass S F,Sanmiguels P,Clark L K.Demonstration of vertical transmission ofStreptococcussuisin swine by genomic fingerprinting[J].Clin Microbiol,1997,35(6):1595-1959.

