耐复方新诺明香港海鸥型菌中Ⅰ类整合子、磺胺类耐药基因(sul1、sul2)的检测*
陈 冰,冯嘉丽,李美霞,朱江峰,魏耀红,张 欧,康 燕,俞守义,陈 清,胡 静
耐复方新诺明香港海鸥型菌中Ⅰ类整合子、磺胺类耐药基因(sul1、sul2)的检测*
陈 冰1,冯嘉丽1,李美霞2,朱江峰1,魏耀红2,张 欧1,康 燕2,俞守义1,陈 清1,胡 静1
目的了解香港海鸥型菌淡水鱼与蛙来源分离株磺胺类耐药性以及Ⅰ类整合子介导耐药的情况。方法采用K-B纸片扩散法检测香港海鸥菌耐复方新诺明药物菌株,PCR方法扩增耐药菌株sul1、sul2和Ⅰ类整合子基因。结果128株香港海鸥菌中共有20株耐复方新诺明药物,耐药率15.6%;20株耐药菌株中sul1阳性率90%,sul2阳性率50%,Ⅰ类整合子携带率80%。结论淡水鱼与蛙来源香港海鸥型菌菌耐磺胺的机制可能与Ⅰ类整合子、sul1、sul2基因有关。
香港海鸥型菌;Ⅰ类整合子;磺胺类耐药基因
香港海鸥菌(Laribacter hongkongensis,LH)最早为2001年香港大学学者在一位肝硬化病人的胸腔积脓中分离得到,经鉴定为变形菌门、β-亚纲奈瑟氏菌科的新菌属[1]。2004年,香港学者 Woo等进行的一项多中心病例对照研究中表明[2],香港海鸥菌与旅行者腹泻和社区获得性胃肠炎有关。目前,香港海鸥菌在香港地区、中国大陆多省份、瑞士、日本、非洲等多地区均有检出[2-4],香港海鸥菌主要分布在某些种类的淡水产品中。在之前的研究中发现,香港海鸥菌存在对多种抗生素都存在耐药性[1,5-6]。鉴于复方新诺明是临床治疗消化道感染的抗菌药物之一,为了解淡水产品分离的香港海鸥菌对复方新诺明耐药及磺胺类耐药基因携带情况,我们对广州市部分淡水鱼与蛙分离香港海鸥菌复方新诺明耐药性及磺胺类耐药基因进行检测。
1 材料与方法
1.1 菌株 香港海鸥型菌株由本实验室冯嘉丽等[5]于2008年11月-2009年4月,自广州市淡水产品的肠道中分离获得香港海鸥菌128株,其中鱼源株39株(30.46%),蛙源株89株(69.53%)。
1.2 试剂来源 K-B药敏纸片购自Oxiod公司,DNA提取试剂盒购自天根生物有限公司,引物合成自英伟创津公司,PCR试剂盒购自大连宝生公司等。
1.3 药敏实验 采用K-B纸片扩散法,金黄色葡萄球菌(ATCC25923)、大肠杆菌(ATCC25922)作为质控菌,判定按照美国临床与实验室标准(CLSI)肠杆菌科判断标准(2008年版)执行。抗生素纸片包括:头孢菌素类类、青霉素类、氨基糖甙类、四环素、复方新诺明等。
1.4 基因检测 挑选出对复方新诺明耐药的菌株进行Ⅰ类整合子及磺胺类耐药基因sul1、sul2的PCR扩增,扩增体系、引物设计参考文献[7](见表1)。磺胺类耐药基因阳性对照株为本实验室PCR检测阳性且经测序验证,GenBank登录号分别为JN088221和JN088222,整合子阳性对照株为本实验室保存[5]。所得PCR扩增产物由华大基因公司测序。

表1 扩增引物序列及退火温度Tab.1 Oligonucleotides for PCR analysis of integrons and suls and annealing temperature
2 结 果
2.1 香港海鸥菌药敏实验结果 128株分离得到的海鸥菌对复方新诺明耐药共20株,耐药率15.6%,其中蛙源香港海鸥菌18株,鱼源2株,耐药率分别为20.22%和5.13%。
2.2 Ⅰ类整合子及磺胺类耐药基因检测 20株耐复方新诺明香港海鸥菌中,有16株Ⅰ类整合子检测阳性,阳性率80%,整合子扩增产物片段为可变区,大小在1 100~3 000 bp左右。
本次实验中共检出sul1基因阳性菌18株(90%),扩增片段大小约为417 bp,sul2基因10株(50%),扩增片段大小约为287 bp。16株Ⅰ类整合子阳性株中,全部sul1基因阳性(100%),10株sul2基因阳性(62.5%),检测到同时存在sul1和sul2两种基因的有10株。有2株Ⅰ类整合子阴性株,然而sul1基因检测阳性。详见表2。
实验中,另随机抽取30株对复方新诺明敏感的菌株分别进行3种基因的检测,结果均为阴性。
3 讨 论
本次实验中分离的128株香港海鸥菌中磺胺类药耐药率15.6%,表明磺胺类耐药存在一定的耐药性。革兰氏阴性肠道细菌对磺胺类药物的耐药性与存在磺胺类耐药基因(例如sul)有关,过去认为革兰氏阴性肠道细菌对磺胺类药物的耐药性很大程度上是由质粒介导,目前认为Ⅰ类整合子也是介导耐药性的重要元件[9-10]。整合子是存在于细菌基因组或质粒上的基因片段,具有位点特异性重组功能,可存在于质粒、转座子等可移动基因元件上,在不同种的细菌之间进行水平转移,是细菌多重耐药性形成和扩散的重要原因。
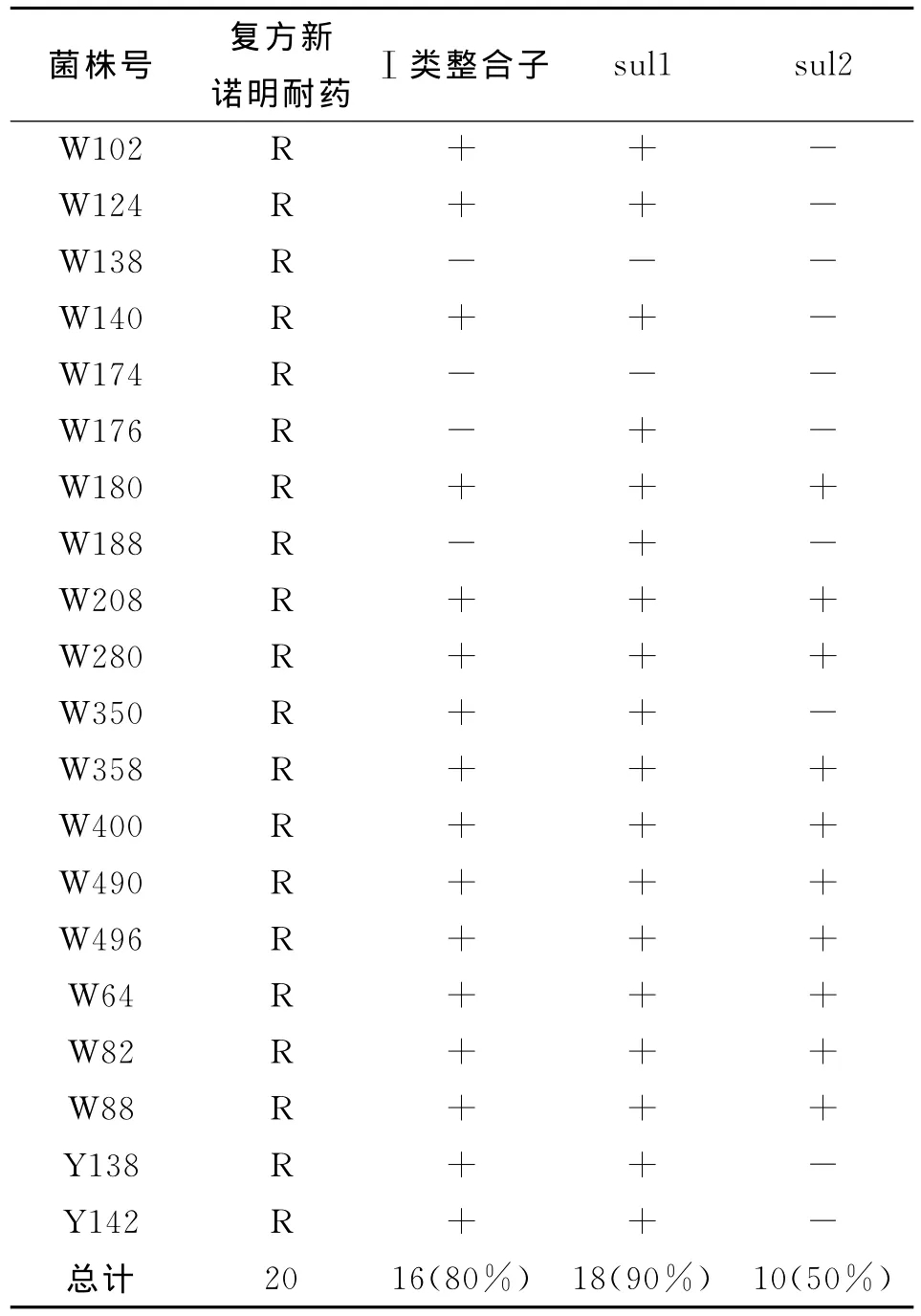
表2 香港海鸥菌耐药菌株及Ⅰ类整合子和磺胺类耐药基因携带情况Tab.2 Class I integrons and sul genes detected in the 20 SXT-resistant Laribacter hongkongensis isolates
本研究结果显示,在20株耐复方新诺明药物的香港海鸥菌中,磺胺类耐药基因sul1、sul2的检出率分别为90%和50%,而30株复方新诺明敏感菌株的这两种基因均为阴性,表明了香港海鸥菌对复方新诺明的耐药与sul基因,特别是sul1基因的存在密切相关。但本研究也发现有个别耐药菌株sul基因阴性(2/20,表2),表明对磺胺类药物的耐药不仅有sul基因参与。本课题组前期的研究表明[5],Ⅰ类整合子携带有5种类型基因盒:包括dfr A1-orf C,aad A1-dfr A,dfr A17-aad A5,dfr A14-arr2-cml A5,dfr A32-ere A-aad A2,其中dfr基因也是一种与磺胺类药物耐药有关的基因。
20株耐复方新诺明香港海鸥菌中,有16株Ⅰ类整合子检测阳性,阳性率80%。在这16株Ⅰ类整合子阳性的菌株中,sul1基因全部阳性(16/16),sul2基因62.5%(10/16)阳性。这个结果说明了Ⅰ类整合子可能是介导香港海鸥菌耐磺胺类药物的主要元件。
Partridge等研究表明[10],典型的整合子结构包括它的5′-CS保守序列包含整合酶基因及它的3′-CS端保守序列包含qacE△1+sul1基因分别编码季胺类消毒剂和磺胺类耐药基因,但是也存在3′端保守端缺失的现象。本研究中,在磺胺类耐药整合子阳性株中均检出了sul1基因。而2株整合子基因检测阴性,但是检测存在sul1基因,是否是存在整合子3′端缺失的现象,有待进一步深入的研究。
研究中还观察到,虽然部分菌株表现为对复方新诺明耐药,但是未检测到Ⅰ类整合子、sul1和sul2基因,可能是其存在其他类型耐药基因或存在其他的耐药机制,例如生物膜的形成、外膜通透性改变和外排泵的过度表达都可能导致细菌的耐药[11]。因此,对于香港海鸥菌耐磺胺类药物的机制的了解,还需要更多的研究。
随着对香港海鸥菌研究的不断深入,陆续自淡水鱼、食用虎纹蛙、田螺、白鹭甚至水库水体中发现香港海鸥菌的存在。Macros等学者[12]提出将香港海鸥菌与产肠毒素脆弱类杆菌、产酸克雷伯杆菌列入2004-2007年间新发现的3种引起人类急性细菌性腹泻的病原体。香港海鸥菌作为一种新的细菌性腹泻的病原体已受到广泛关注。实验中首次对香港海鸥菌中磺胺类耐药基因进行检测,对香港海鸥菌携带磺胺类耐药基因进行初步的流行病学调查,丰富了耐药基因的流行病学资料。
[1]Yuen K Y,Woo P C,Teng J L,et al.Laribacter hongkongensis gen.nov.,sp.nov.,a novel gram-negative bacterium isolated from a cirrhotic patient with bacteremia and empyema[J].J Clin Microbiol,2001,39(12):4227-4232.
[2]Woo P C,Lau S K,Teng J L,et al.Association of Laribacter hongkongensis in community-acquired gastroenteritis with travel and eating fish:a multicentre case-control study[J].Lancet,2004,363(9425):1941-1947.
[3]Ni X P,Ren S H,Sun J R,et al.Laribacter hongkongensis isolated from a patient with community-acquired gastroenteritis in Hangzhou City[J].J Clin Microbiol,2007,45(1):255-256.
[4]谭海芳,谭翰清,丁丽娜,等.筛选与确诊试验在检测香港海鸥菌中的建立与应用[J].热带医学杂志,2010(6):675-677.
[5]Feng J L,Yan H,Chowdhury N,et al.Identification and characterization of integron-associated antibiotic resistant Laribacter hongkongensis isolated from aquatic products in China[J].Int J Food Microbiol,2011,144(3):337-341.
[6]Woo P C,Kuhnert P,Burnens A P,et al.Laribacter hongkongensis:a potential cause of infectious diarrhea[J].Diagn Microbiol Infect Dis,2003,47(4):551-556.
[7]Aarestrup F M,Lertworapreecha M,Evans M C,et al.Antimicrobial susceptibility and occurrence of resistance genes among Salmonella enterica serovar Weltevreden from different countries[J].J Antimicrob Chemother,2003,52(4):715-718.
[8]Levesque C,Piche L,Larose C,et al.PCR mapping of integrons reveals several novel combinations of resistance genes[J].Antimicrob Agents Chemother,1995,39(1):185-191.
[9]Vinue L,Saenz Y,Rojo-Bezares B,et al.Genetic environment of sul genes and characterisation of integrons in Escherichia coli isolates of blood origin in a Spanish hospital[J].Int J Antimicrob Agents,2010,35(5):492-496.
[10]Partridge S R,Tsafnat G,Coiera E,et al.Gene cassettes and cassette arrays in mobile resistance integrons[J].FEMS Microbiol Rev,2009,33(4):757-784.
[11]Toleman M A,Bennett P M,Walsh T R.ISCR elements:novel gene-capturing systems of the 21st century[J].Microbiol Mol Biol Rev,2006,70(2):296-316.
[12]Marcos L A,Dupont H L.Advances in defining etiology and new therapeutic approaches in acute diarrhea[J].Journal of Infection,2007,55(5):385-393.
Detection of the classⅠintegrons,sul1 and sul2 genes in trimethoprim-sulfamethoxazole resistant Laribacter hongkongensis isolates
CHEN Bing1,FENG Jia-li1,LI Mei-xia2,ZHU Jiang-feng1,WEI Yao-hong2,ZHANG Ou1,KANG Yan2,YU Shou-yi1,CHEN Qing1,HU Jing1
(School of Public Health and Tropical Medicine,Southern Medical University,Guangzhou 510515,China)
The objective was to study the occurrence of sulphonamide resistance genes and classⅠintegrons in Laribacter hongkongensis(LH)isolates.The isolates were obtained from freshwater fishes and frogs.The trimethoprim-sulfamethoxazole(SXT)agent susceptibility was carried out by the K-B disk diffusion method,and all sulphonamide-resistant isolates were investigated for the presence of sul1,sul2 and class I integron genes by PCR.The 15.6% (20/128)of the Laribacter hongkongensis isolates were resistant to SXT.Among the SXT resistant isolates,sul1,sul2 and class I integron genes were detected out 90% (18/20),50% (10/20)and 80% (16/20),respectively.Results indicate that the mechanism of SXT-resistant LH isolates from freshwater fishes and frogs are likely to associate with sul1,sul2 and class I integron genes.
Laribacter hongkongensis;class I integron;sul gene
R378
A
1002-2694(2012)03-0223-03
*国家自然科学基金项目(No.30700686)和广州市医药卫生科技项目(2009-2di-19)
胡静,Email:hjalzh@fimmu.com;陈清,Email:qch.2009@163.com
1.南方医科大学公共卫生与热带医学学院,广州510515;2.广州市疾病预防控制中心,广州 510030
2011-09-26;
2011-12-20

