舟山近海条石鲷野生群体与人工放流群体遗传多样性的AFLP分析
李三磊, 徐冬冬 楼 宝 王伟定 辛 俭 毛国民 詹 炜
(1. 浙江省海洋水产研究所, 浙江省海水增养殖重点实验室, 浙江 舟山 316100; 2. 浙江海洋学院, 浙江 舟山316004)
舟山近海条石鲷野生群体与人工放流群体遗传多样性的AFLP分析
李三磊1,2, 徐冬冬1, 楼 宝1, 王伟定1, 辛 俭1, 毛国民1, 詹 炜1
(1. 浙江省海洋水产研究所, 浙江省海水增养殖重点实验室, 浙江 舟山 316100; 2. 浙江海洋学院, 浙江 舟山316004)
利用 AFLP技术对舟山近海条石鲷(Oplegnathus fasciatus)野生和放流群体的遗传多样性进行比较分析, 旨在为条石鲷的人工增殖及其种质资源的保护和利用提供遗传学的基础资料。采用 8对引物组合在2个群体中共扩增得到位点316个, 多态性位点为162个, 多态性比例为51.67%。野生群体和放流群体的多态性位点比例(P), Nei’s基因多样性指数(H)和Shannon’s多样性指数(I)分别为46.75%和44.67%, 0.097和 0.089, 0.16和 0.15。野生群体的遗传多样性水平略高于放流群体, 但差异并不显著(P>0.05)。两群体间的遗传相似系数(S)和遗传距离(D)分别为0.99和0.0073; 基因分化系数(Gst)为0.036,群体间无显著遗传分化(P>0.05)。分子方差(AMOVA)分析表明96.82%的遗传变异来源于群体内的个体间, 群体间无显著的遗传差异(P>0.05)。以上研究结果表明条石鲷的放流群体与野生群体间无明显的遗传分化, 放流群体的遗传多样性水平尚处于一个合理状态。但为了避免放流群体对野生群体产生负面的遗传效应, 应当增加放流群体繁育亲本的数量, 并对放流群体的遗传变异水平进行持续监测。
条石鲷(Oplegnathus fasciatus); 野生群体; 人工放流群体; 遗传多样性; AFLP
针对目前海洋渔业资源衰退现状, 世界各国政府均实施积极的资源养护和渔业管理措施, 优化渔业资源结构, 改善海域生态环境, 恢复日趋衰退的海洋渔业资源。其中, 人工增殖放流是恢复渔业资源最为直接有效的措施。但是, 增殖放流的苗种来自于人工繁育, 从保护生态学角度来看, 由于放养鱼类与野生鱼类之间存在遗传差异, 可能对自然群体产生负面影响, 使鱼类基因库的遗传变异降低, 从而直接影响到野生群体的遗传多样性水平[1-2]。
一直以来, 养殖群体对野生群体的遗传效应备受研究人员关注, 因放养而导致的负面遗传影响的证据日益增多。在大西洋鲑中, 多代连续的人工养殖造成养殖群体 42%的等位基因丢失, 当这些苗种放流或逃逸到自然界时, 对野生群体的遗传结构产生影响, 导致野生群体的数量降低[3-4]。日本学者研究发现条斑星鲽(Verasper moseri)放流回捕群体的遗传变异水平和有效群体大小较低, 具有引起近交衰退的风险[5]。中国学者吴旭等[6]研究发现, 肖四海湖野生和放流的鳜群体的遗传结构存在显著分化。Fritzner等[7]研究认为, 为避免人工增殖群体与野生群体的基因库混杂, 在实施增殖时, 应对人工放流群体进行遗传多样性评估, 从而确保野生群体的遗传结构稳定。因此, 采用适当、适时监控, 研究放流群体与野生群体的遗传异质性, 了解放流物种资源的遗传结构及其遗传多样性状况, 对评估增殖放流效果、丰富海洋生物的遗传学背景资料具有着重要的作用。
条石鲷(Oplegnathus fasciatus), 隶属于鲈形目(Perciformes)、石鲷科(Oplegnathidae)、石鲷属(Oplegnathus), 俗称日本鹦鹉鱼, 自然分布于太平洋和印度洋沿岸, 中国产于黄海、东海和台湾海峡, 是一种暖温性海洋中下层的岛礁性鱼类[8-9]。近年来,由于环境污染、气候条件变化及过渡捕捞等原因, 造成包括条石鲷在内的野生经济鱼类资源急剧衰退。条石鲷被农业部确定为重要的人工增殖放流品种,自 2006年起连续多年在舟山海域进行人工增殖放流。目前, 有关条石鲷的研究主要涉及早期发育[10-11]、人工繁育[12]及养殖[13-14]等方面。关于其群体遗传学的研究报道仅见于孙鹏等[15-16]利用线粒体控制区域序列分析技术研究了条石鲷单一性群体的遗传多样性水平和遗传结构。有关条石鲷野生群体和人工放流群体的遗传多样性的比较分析尚未见报道。本研究利用 AFLP技术对舟山近海的条石鲷野生群体和放流群体的遗传多样性进行比较分析, 探讨野生群体和放流群体的遗传差异, 为条石鲷人工增殖提供科学指导, 并为其种质资源的保护和利用提供遗传学的基础资料。
1 材料与方法
1.1 材料
实验所用的一个条石鲷群体于2005年采自舟山附近海区, 取样前未曾进行条石鲷的人工放流活动,故所取样本可以认定为野生群体, 共 30尾, 平均体长为18.35 cm±1.32 cm。另一个群体为人工放流群体,由舟山市水产研究所于2010年人工繁育所得, 共30尾, 平均体长为5.53 cm±0.52 cm。人工放流群体的亲鱼为舟山嵊泗附近海域放流前捕获的野生鱼驯养而成, 共32尾, 雌性与雄性的比例约为2 :1。对采集样本取肌肉或尾鳍, –20℃冰箱保存备用。
1.2 基因组DNA提取
基因组DNA采用海洋动物组织基因组DNA提取试剂盒提取(TIANGEN, 北京), 然后用 0.8%琼脂糖凝胶电泳检测, 并利用紫外分光光度计测定 DNA浓度和纯度, 用双蒸水稀释至50 mg/L备用。
1.3 AFLP分析
AFLP分析参照Vos等[17]的方法。根据预实验结果, 选取选择性引物组合 E-AAC/ M-CTC、E-AAC/M-CTG、E-ACA/ M-CAT、E-ACA/ M-CTT、E-ACC/M-CAT、E-ACC/ M-CTT、E-AGA/ M-CAT 和 E-AGA/M-CTT用于AFLP分析。选择性扩增产物经6%的变性聚丙烯酰胺凝胶电泳后银染显色, 将获得清晰条带的胶板进行拍照并记录结果。
1.4 数据处理和分析
根据电泳图谱中扩增条带的有无分别记录为“1”和“0”, 所有数据统计后形成矩阵, 统计多态性位点数, 计算显性基因型频率等参数。利用Popgen1.32[18]软件计算多态性位点比例 (P)、Shannon’s多样性指数 (I)[19]、Nei’s[20]基因多样性指数 (H)、Nei’s[21]遗传距离 (D) 及遗传相似系数 (S)、Nei’s[22]基因分化系数 (Gst) 等遗传参数, 利用 Arlequin3.1[23]软件进行 AMOVA分析。基于个体间的遗传距离, 利用Mega3.1[24]软件构建UPGMA系统树。
2 结果与分析
2.1 AFLP扩增结果
利用筛选出的 8对选择性引物组合, 在两个群体的60个个体中共得到316个清晰位点, 其中多态性位点为162个, 多态性比例为51.67%(图1)。不同引物的扩增位点数从33到52不等, 扩增出的多态性位点数为 14~28不等, 检出的多态性比例范围为38.89%~68.29% (表1)。野生和放流群体中分别扩增出多态性位点数为144个和134个, 多态性比例分别为46.75%和44.67%, 野生群体中扩增的多态性比例略高于人工放流群体, 但差异并不显著 (df =1,χ2=0.23,P>0.05)。

图1 引物组合E-AGA/M-CTT扩增得到的条石鲷AFLP指纹图谱Fig. 1 AFLP patterns of Oplegnathus fasciatus using primer set E-AGA/M-CTT
2.2 野生群体与放流群体的遗传变异分析
条石鲷野生群体与放流群体的 Nei’s基因多样性指数(H)和 Shannon’s多样性指数(I)分别为 0.097和 0.089, 0.16和0.15(表2)。野生群体的 Nei’s基因多样性指数(H)和 Shannon’s多样性指数(I)略高于放流群体。两群体间的遗传相似系数和遗传距离分别为0.99和0.0073, 基因分化系数Gst为0.036, 两群体间无显著遗传分化(P>0.05); AMOVA分析结果表明96.82%的遗传变异来源于群体内的个体间, 仅有3.16%的遗传变异来自群体间, 两群体间无显著遗传差异 (P>0.05, 表3)。
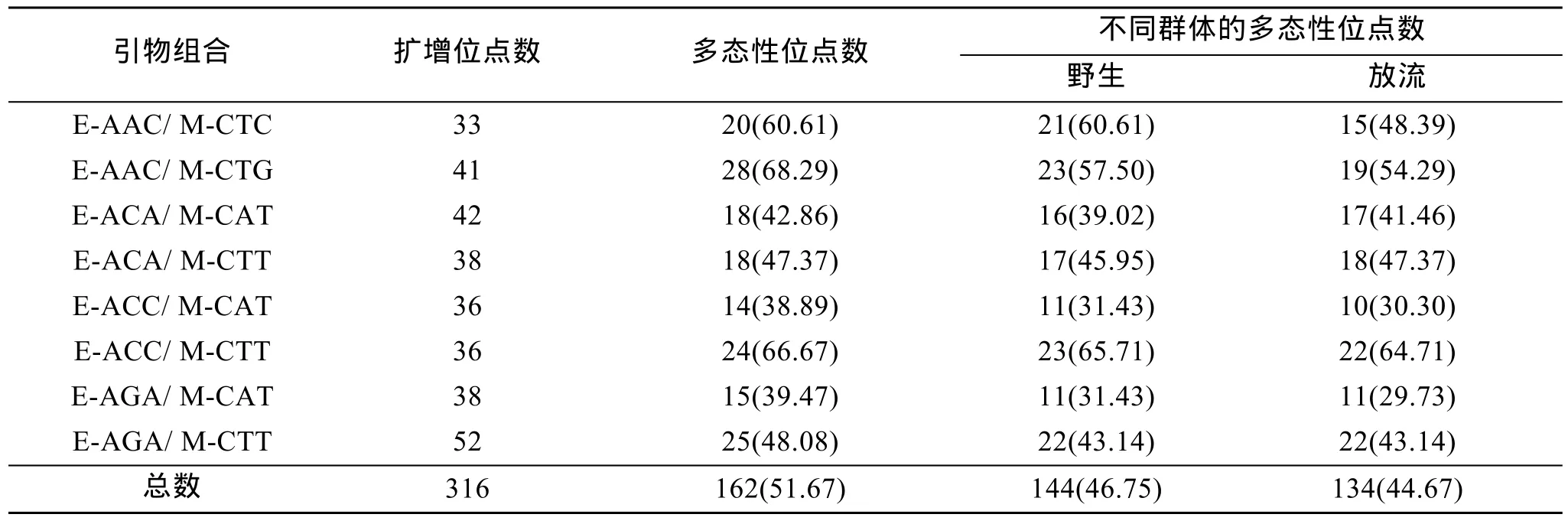
表1 不同引物组合的扩增结果Tab. 1 Amplification results of different primer combinations

表2 条石鲷两个群体的遗传多样性参数Tab. 2 Parameters of genetic diversity in two populations of Oplegnathus fasciatus

表3 条石鲷两个群体遗传变异的AMOVA分析Tab. 3 Analysis of molecular variance (AMOVA) in two populations of Oplegnathus fasciatus
将 316个扩增位点的显性基因型频率以 10%为单位划分区间, 0和100%分别设为一个单独的区间,共划分12个区间, 统计显性基因型频率位于各区间内的位点数。结果显示, 两个群体扩增位点数在不同显性基因型频率区间的分布呈现一定的规律, 反映了两个群体的遗传结构特点 (图2)。野生群体在1%~9%区间的扩增位点数呈现峰值, 放流群体在20%~29%区间的扩增位点数呈现峰值, 两个群体在30%~100%区间的扩增位点数分布变化趋势基本一致。从扩增位点数在不同显性基因频率区间的分布来看, 放流群体的低频率显性位点数比野生群体的少, 表明野生群体的显性基因频率分布相对放流群
2.3 野生群体与放流群体的聚类分析
利用MEGA3.1软件基于条石鲷60个个体间的遗传距离, 构建了UPGMA系统树如图3。系统树结果显示, 60个个体分为两支, 两个群体的个体分散于两个分支, 没有出现群体聚类的现象, 表明两个群体没有出现明显的遗传分化, 具有较近的亲缘关系。
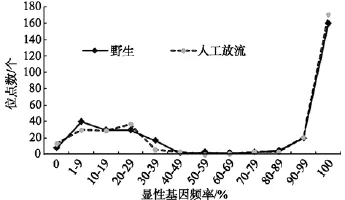
图2 扩增位点数在不同显性基因频率区间内的分布Fig. 2 Distributions of amplified loci in different frequency of intervals
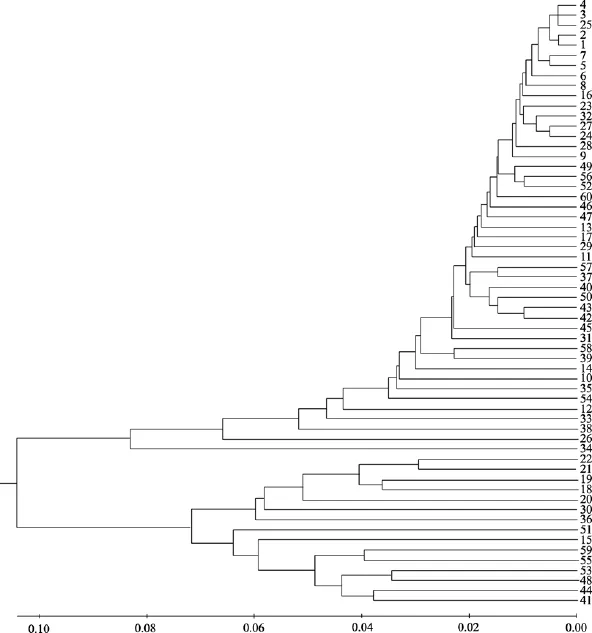
图3 条石鲷60个个体的UPGMA聚类树Fig. 3 UPGMA dendrogram of 60 individuals of Oplegnathus fasciatus
3 讨论
本实验利用 8对 AFLP选择性引物组合对舟山近海条石鲷野生群体与人工放流群体的遗传多样性进行分析, 在 60个个体中共得到扩增位点 362个,不同引物组合扩增位点数从33到52不等, 多态性位点数为14~28不等, 每对引物平均得到扩增位点数和多态性位点数分别约为40和20, AFLP标记表现出了丰富的多态性和较高的灵敏度。
多态性位点比例(P)、Nei’s基因多样性指数(H)和 Shannon’s多样性指数 (I) 是衡量群体遗传多样性的重要参数, 反映了群体在数个基因座位上的遗传变异, 其值大小可以反映群体遗传变异的高低。本研究中, 条石鲷野生群体的多态性位点比例为46.75%, Nei’s遗传多样性指数(H) 和 Shannon’s多样性指数(I)分别为0.097和0.16。与中国近海鱼类现有的 AFLP分析数据相比, 条石鲷的遗传多样性水平较真鲷(Pagrus major) (P=58.4%~64.0%)[25]、小黄鱼(Larimichthys polyactis) (P=55.34~60.09%,H=0.12~0.14)[26]、银鲳 (Pampus argenteus) (P=62.9%~67.0%,I=0.23~0.25)[27]低, 与黄姑鱼 (Nibea albiflora)(P=51.70%,H=0.10)[28]相当, 但较褐牙鲆(Paralich-thys olivaceus) (P=40.29~44.31%)[29]和 马 鲛 鱼(Scomberomorus niphonius) (P=38.54%~45.70%,H=0.081~0.098)[30]高。这表明条石鲷的野生群体的遗传变异水平相对略高。孙鹏等[15]利用线粒体COⅠ基因和Cytb基因对舟山海域的条石鲷野生群体进行群体遗传学分析也表明其野生群体具有较高的遗传多样性水平。这可能与海洋鱼类的生境面积广大且种群分布分散有关[26-28]。
物种的遗传多样性高低与其适应能力、生存能力和进化潜力密切相关。野生种群遗传多样性水平的减少可能导致鱼类成活率、生长与繁殖效率变低,降低种群个体对变化的环境的适应能力[31]。由于放流的苗种来自人工养殖, 许多研究表明养殖群体的遗传变异水平低于野生群体, 如大西洋鲑(Salmo salarL.)[3]、褐牙鲆[29,32]等, 因此, 放流苗种可能对野生群体产生负面的遗传影响。本研究中, 比较条石鲷两个群体的扩增多态位点数、多态性位点比例(P)、Nei’s基因多样性指数(H)和 Shannon’s多样性指数(I)等参数发现野生群体的遗传变异水平略高于放流群体, 但差异不显著, 群体间无明显的遗传分化,这可能是由于人工放流群体的繁育亲鱼与野生群体来自同一海域有关; 然而, 放流群体中1%~9%低频位点数略低于野生群体, 这可视为隐性纯合位点过剩, 稀有位点的丢失, 致使放流群体的遗传多样性水平会略低于野生群体, 这可能由于放流群体培育过程中亲鱼数量限制及繁殖过程中的一些不合理因素(如亲鱼大小、亲鱼雌雄比例、遗传漂变等)所致[31-33]。条石鲷野生群体稀有位点对整个群体的遗传变异水平影响不算大, 但对于整个基因库却很重要。因此, 对该条石鲷人工放流群体的遗传多样性水平处于一个合理状态的现状也不能过于乐观。
人工增殖放流是快速恢复种群数量的有效方法,然而, 人工放流的遗传效应一直存在争议。Gonzalez[34]研究发现:虽然近 30年的人工放流使日本海域黑鲷(Acanthopagrus schlegelii)种群数量增加,但也导致其自然群体的遗传变异水平有所下降。Kitada[35]对真鲷和太平洋鲱(Clupea pallasi)增殖放流的遗传效应进行跟踪研究, 结果发现放流群体并未影响野生群体的遗传多样性。保证人工放流群体的遗传多样性水平可以避免其对野生群体遗传危害。从种质资源保护的角度出发, 进行人工放流时用于人工繁育的亲本应当直接从自然群体中选择, 不要采用养殖的子代作为亲本, 且亲本数量不宜过少,一般推荐的亲本数量在50~350尾[31,36]。本研究中,条石鲷放流群体的亲本与野生群体来自同一海域,且放流群体的遗传多样性水平尚处于较为合理的状态, 可能不会对野生群体的遗传结构造成影响。但为了避免放流群体对自然群体遗传多样性的负面影响,应当增加繁育亲本的数量; 而且, 在今后的放流过程中要对放流群体的遗传变异水平进行持续监测。
[1]尤锋, 张培军, 相建海, 等. 海水养殖鱼类遗传多样性的保护[J]. 海洋科学, 2003, 27(12):10-13.
[2]Schwartz M K, Luikart G, Waples R S. Genetic monitoring as a promising tool for conservation and management [J]. Trends Ecol Evol, 2007, 22(1):25-33.
[3]Skaala O B, Hoyheim K, Glover K, et al. Microsatellite analysis in domesticated and wild Atlantic salmon(Salmo salarL.):allelic diversity and identification of individuals [J]. Aquaculture, 2004, 240(1-4):131-143.
[4]Castillo A G F, Ayllon F, Moran P, et al. Interspecific hybridization and introgression are associated with stock transfers in salmonids[J]. Aquaculture, 2008,278(1-4):31-36.
[5]Ortega-Villaizá N R. Monitoring of the genetic variability of the hatchery and recaptured fish in the stock enhancement program of the rare species barfin flounderVerasper moseri[J]. Fisheries Sci, 2005, 71(5):1120-1130.
[6]吴旭, 严美姣, 李钟杰. 肖四海湖野生和人工放流鳜群体遗传结构分析[J]. 水生生物学报, 2010, 34(3):562-568.
[7]Fritzner N G, Hansen M M, Madsen S S, et al. Use of microsatellite markers for identification of indigenous brown trout in a geographical region heavily influenced by stocked domesticated trout[J]. J Fish Biol, 2001,58(5):1197-1210.
[8]朱元鼎, 张春霖, 成庆泰. 东海鱼类志[M]. 北京:科学出版社, 1963:254-255.
[9]张春霖, 成庆泰, 郑葆珊, 等. 黄渤海鱼类调查报告[M].北京:科学出版社, 1995:153-154.
[10]常抗美, 毛建平, 吴剑锋, 等. 条石鲷胚胎及仔稚鱼的发育[J]. 上海水产大学学报, 2005, 14(4):401-405.
[11]柳学周, 徐永江, 王妍妍, 等. 条石鲷的早期生长发育特征[J]. 动物学报, 2008, 54(2):332-341.
[12]倪梦麟, 范卫明, 毛志增, 等. 舟山沿海条石鲷人工育苗技术研究[J]. 浙江海洋学院学报(自然科学版),2007, 26(2):143-146.
[13]Tachibana K, Yagi M, Hara K, et al. Effects of feeding of β-carotene supplemented rotifers on survival and lymphocytes proliferation reaction of fish larvae(Japanese parrotfish (Oplegnathus fasciatus) and spotted parrotfish (O. punctatus)) preliminary trials [J].Hydrobiologia, 1997, 358:313-316.
[14]徐永江, 柳学周, 王妍妍, 等. 温度、盐度对条石鲷胚胎发育影响及初孵仔鱼饥饿耐受力[J]. 渔业科学进展, 2009, 30(3):25-30.
[15]孙鹏, 尹飞, 彭士明, 等. 条石鲷线粒体 COⅠ和Cytb序列的遗传变异[J]. 水产学报, 2011, 35(3):327-333.
[16]孙鹏, 李杰, 尹飞, 等. 条石鲷养殖群体线粒体控制区序列遗传变异分析[J]. 海洋渔业, 2011, 33(1):9-14.
[17]Vos P, Hogers R, Bleeker M, et al. AFLP:a new technique for DNA fingerprinting [J]. Nucleic Acids Res,1995, 23:4407-4414.
[18]Yeh F C, Yang R C, Boyle T. Population genetic analysis of co-dominant and dominant markers and quantitative traits [J]. Belg J Bot, 1997, 129:157.
[19]Lewontin R C. The apportionment of human diversity[J].Evol, 1972, 6:381-398.
[20]Nei M. Analysis of gene diversity in subdivided populations[J]. Proc Natl Acad Sci USA, 1973, 70:3321-3323.
[21]Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals[J].Genet, 1978, 89:583-590.
[22]Nei M. Molecular Evolutionary Genetics[M]. New York:Columbia University Press, 1987:512.
[23]Excoffier L, Laval G, Schneider S. ARLEQUIN, version 3.0:an integrated software package for population genetics data analysis[J]. Evol Bioinform, 2005, 1:47-50.
[24]Kumar S, Tamura K, Nei M. MEGA 3:Integrated software for molecular evolutionary genetics analysis and sequence alignment[J]. Brief Bioinform, 2004, 5:150-163.
[25]王志勇, 王艺磊, 林利民, 等. 利用 AFLP指纹技术研究中国沿海真鲷群体的遗传变异和趋异[J]. 水产学报, 2001, 25(4):289-293.
[26]Lin L S, Ying Y P, Han Z Q, et al. AFLP analysis on genetic diversity and population structure of small yellow croakerLarimichthys polyactis[J]. Afr J Biotechnol,2009, 8(12):2700-2706.
[27]Zhao F, Dong Y H, Zhuang P, et al. Genetic diversity of silver pomfret (Pampus argenteus) in the Southern Yellow and East China Seas[J]. Biochem Syst Ecol,2011, 39(2):145-150.
[28]韩志强, 高天翔, 王志勇, 等. 黄姑鱼群体遗传多样性的AFLP分析[J]. 水产学报, 2006, 30(5):641-646.
[29]Xu X F, Zhang Q Q, Wang Z G, et al. Assessing genetic diversity of wild populations of Japanese flounder using AFLP markers[J]. Acta Oceanol Sin, 2006, 25(3):82-89.
[30]Shui B N, Han Z Q, Gao T X, et al. Genetic structure of Japanese Spanish mackerel (Scomberomorus niphonius)in the East China Sea and Yellow Sea inferred from AFLP data[J]. Afr J Biotechnol, 2008, 7(21):3860-3865.
[31]Lorenzen K. Population dynamics and potential of fisheries stock enhancement:practical theory for assessment and policy analysis[J]. Philos T R Soc B,2005, 360(1453):171.
[32]张全启, 徐晓斐, 齐洁, 等. 牙鲆野生群体与养殖群体的遗传多样性分析[J]. 中国海洋大学学报, 2004,34(5):816-820.
[33]Araki H, Schmid C. Is hatchery stocking a help or harm?Evidence, limitations and future directions in ecological and genetic surveys[J]. Aquaculture, 2010, 308:S2-S11.
[34]Gonzalez E B, Nagasawa K, Umino T. Stock enhancement program for black sea bream (Acanthopagrus schlegelii) in Hiroshima Bay:monitoring the genetic effects[J]. Aquaculture, 2008, 276(1-4):36-43.
[35]Kitada S, Shishidou H, Sugaya T, et al. Genetic effects of long-term stock enhancement programs[J]. Aquaculture, 2009, 290(1-2):69-79.
[36]Gold J R, Ma L, Saillant E, et al. Genetic effective size in populations of hatchery-raised red drum released for stock enhancement[J]. T Am Fish Soc, 2008, 137(5):1327-1334.
The genetic diversity of wild and hatchery-releasedOplegnathus fasciatusfrom inshore water of Zhoushan revealed by AFLP
LI San-lei1,2, XU Dong-dong1, LOU Bao1, WANG Wei-ding1, XIN Jian1,MAO Guo-min1, ZHAN Wei1
(1. Marine Fishery Institute of Zhejiang Province, Zhejiang Province Key Lab of Mariculture and Enhancement,Zhoushan 316100, China; 2. Zhejiang Ocean University, Zhoushan 316004, China)
Apr.,18, 2011
Oplegnathus fasciatus; wild population; hatchery-released population; genetic diversity; AFLP
The genetic variations of wild and hatchery-releasedOplegnathus fasciatusfrom inshore water of Zhoushan were analyzed using amplified fragment length polymorphism (AFLP) to provide basic genetic information for the population enhancement and genetic resource protection and utilization in this fish species. A total of 316 loci were generated in the two populations using 8 primer combinations, among which 162 (51.67%) were polymorphic.The percentages of polymorphic loci in the wild or hatchery-released populations were 46.75% or 44.67%, respectively. The genetic diversities in terms of Nei’s gene diversity and Shannon’s diversity index in the wild population were 0.097 and 0.16, respectively. The corresponding values in hatchery-released population were 0.089 and 0.15,respectively. These results showed that the genetic diversity in the wild population was slightly higher than that in the hatchery-released population, but the difference in genetic diversity of the two populations was not significant(P>0.05). The genetic similarity coefficient and genetic distance between the two populations were 0.99 and 0.0073,respectively. The value ofGst coefficient was 0.036, which indicated no significant genetic differentiation between the two populations (P>0.05). The analysis of molecular variance (AMOVA) showed that 96.82% of the genetic variation occurred among individuals within populations, which also revealed no significant genetic variation between the two populations (P>0.05). The genetic diversity of hatchery-released population was at a reasonable level of state and genetic differentiation was not significant between the wild and hatchery-released populations. It was noticeable to take approaches to avoid the potential negative genetic effects of hatchery-released on wild populations. The number of parental fish should increase and the genetic variation level of hatchery-released population ought to be continuously monitored in the future.
Q953 文献标识码:A 文章编号:1000-3096(2012)08-0021-07
2011-04-18;
2011-10-20
国家科技支撑计划资助项目(2011BAD13B08); 国家海洋公益性行业科研专项(201005013); 国家自然科学基金资助项目(41106114); 浙江省科技计划资助项目(2010F20006); 浙江省海水养殖重点科技创新团队项目(2010R50025)
李三磊(1985-), 男, 山东泰安人, 硕士, 主要从事鱼类遗传育种研究, 电话:0580-3050985, E-mail:sanjun198563@126.com; 楼宝,通信作者,电话:0580-3050985, E-mail:loubao6577@163.com
谭雪静)

