fSSR分析技术的建立及在大黄鱼(Larimichthys crocea)亲子鉴定中的应用
武祥伟,刘贤德,王志勇
(集美大学 水产学院 农业部东海海水健康养殖重点实验室,福建 厦门 361021)
fSSR分析技术的建立及在大黄鱼(Larimichthys crocea)亲子鉴定中的应用
武祥伟,刘贤德,王志勇
(集美大学 水产学院 农业部东海海水健康养殖重点实验室,福建 厦门 361021)
以大黄鱼(Larimichthys crocea)为材料,对荧光标记微卫星(fSSR)分析体系进行了优化,建立了适合大黄鱼fSSR分析的最佳条件。与常规的银染法相比,该方法成本略高,但其分辨率高,可重复性强,检测效率为银染法的6~10倍。利用优化的fSSR技术进行了大黄鱼亲子鉴定的研究,结果显示在使用6对引物的情况下,大黄鱼两个群的亲子鉴定率超过99%。
fSSR;大黄鱼;亲子鉴定;Gel-Scan 3000
微卫星(microsatellite),又称简单重复序列(simple sequence repeats,SSR),是当前应用较为广泛的 DNA分子标记之一[1]。微卫星检测的方法有多种,如同位素法[2]、银染法[3]、测序法[4]、荧光标记法[5]。目前大部分实验室都采用银染法,此方法具有操作简便、无同位素污染等优点,但需时较长,慢速银染法需要2~3 h,快速银染法也在半小时以上。荧光标记微卫星(fSSR)检测技术是近年来发展的一项新技术,其基本原理是在一条或两条引物的5’端标记一种荧光素,如HEX,FAM等,用此种引物进行PCR扩增,产物在聚丙烯酰胺凝胶上分离,在电泳过程中荧光素经激光照射激发出荧光被捕捉到而成像。此种方法无需银染显色,分析效率较高。但目前尚未见使用 fSSR方法对大黄鱼进行分析的报道。
本研究使用Gel-Scan 3000激光扫描电泳系统,对 SSR反应中多个因子进行优化,建立了大黄鱼fSSR分析体系,并使用优化的fSSR技术对2个群的大黄鱼进行了亲子鉴定,以验证该体系的有效性。
1 材料与方法
1.1 材料
1.1.1 fSSR体系建立材料 2009年采自福建省宁德市官井洋海区的养殖大黄鱼,剪取部分鳍条,保存于75 %乙醇,用于DNA抽提。大黄鱼SSR引物[6,7],由上海英骏生物工程公司合成,上游引物5’端标记荧光素HEX(表1)。
1.1.2 亲子鉴定材料 所用材料取自福建省宁德市大黄鱼养殖基地,由集美大学大黄鱼品种选育课题组提供。2007年3月,从经过室内升温强化培育的候选亲本群体中挑选7雌8雄共15尾性状优良、体质健壮、成熟度较好的个体,采用大黄鱼常规育苗生产中所用的“人工催产、随机交配”繁育措施建立了2个窝选群体,分别包含4♀×4♂(1#群),3♀×4♂(3#群)。亲鱼产卵结束后收集受精卵,室内孵化并于1个月后转移至海区网箱养殖。亲本剪取部分背鳍保存,备用。2008年10月分别从2群子代(20月龄)中分别随机选取279尾和315尾,剪取部分背鳍。以上样本固定于75%的酒精中,用于下一步的DNA提取和亲子鉴定分析。

表1 大黄鱼SSR引物信息Tab. 1 SSR primers information of larger yellow croaker
1.2 方法
1.2.1 DNA抽提 采用传统酚-氯仿法提取基因组DNA,1%琼脂糖凝胶电泳检测DNA质量,并将浓度调整至30 µg/mL,置于-20 ℃保存备用。
1.2.2 SSR分析 对影响 PCR反应的主要因子设置梯度进行单因素优化分析,优化时每梯度使用2尾大黄鱼样品。PCR在ABI thermal cycler(美国ABI公司)上进行,初始反应体系参考任鹏等[7]的报道。
1.2.3 变性聚丙烯酰胺凝胶电泳 Gelscan 3000电泳程序:PCR产物中加入1/6体积的6×Loading Buffer(98%去离子甲酰胺,1.75% 10 mmol/L EDTA,0.25%溴酚蓝),95℃变性5 min后迅速置于冰上,取1 µL于5%变性聚丙烯酰胺凝胶(19:1)上电泳。以 GenScanTM-500 TAMRATM Size Standard作为分子量标准,电泳完毕后保存扫描图像,并使用ONE DScan software分析统计数据。
Bio-Rad电泳程序:聚丙烯酰胺凝胶电泳及银染程序参照王志勇等使用的方法[8]进行。
1.2.4 亲子鉴定分析 使用Cervus 3.0软件中鉴定效率最高的NE-PP模型作为分析模式[9,10],首先根据亲本基因频率进行模拟分析,之后依据子代基因型进行亲子鉴定分析。
2 结 果
2.1 影响大黄鱼fSSR体系的因素
2.1.1 模板浓度 7个DNA浓度:5 ng、10 ng、15 ng、20 ng、30 ng、50 ng、60 ng,电泳结果(图1)显示各浓度均能扩增出清晰目的带,从体系稳定性与经济角度考虑,本文选择10 ng的DNA浓度。
2.1.2 引物浓度 6个引物浓度梯度:0.08 µmol/L、0.1 µmol/L、0.15 µmol/L、0.2 µmol/L、0.3 µmol/L、0.35 µmol/L的电泳图谱(图2)显示后2个浓度扩增较好。基于降低成本考虑,引物浓度为0.3 µmol/L较合适。
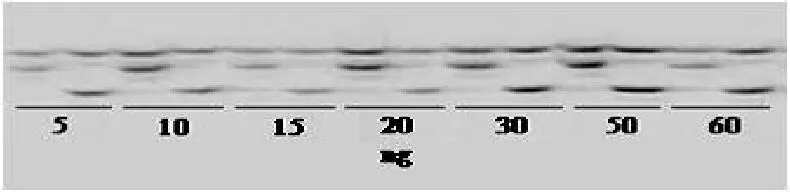
图1 DNA浓度对fSSR扩增的影响Fig.1 Effect of template concentration on fSSR reaction
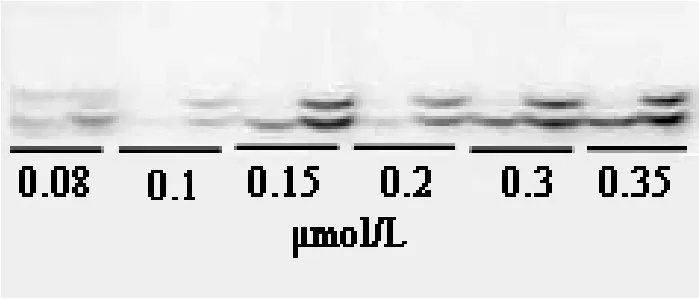
图2 引物浓度对fSSR扩增的影响Fig. 2 Effect of primer concentration on fSSR reaction
2.1.3 Mg2+浓度 7个 Mg2+浓度 1 mmol/L、1.2 mmol/L、1.5 mmol/L、1.8 mmol/L、2.0 mmol/L、2.3 mmol/L、2.5 mmol/L电泳图谱(图3)显示浓度过高出现非特异扩增,Mg2+为1.2 mmol/L目的带亮度合适。
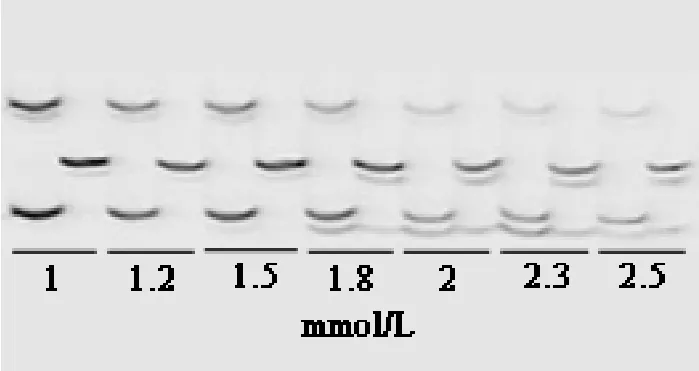
图3 Mg2+浓度对fSSR扩增的影响Fig. 3 Effect of Mg2+ concentration on fSSR reaction
2.1.4 Taq DNA聚合酶浓度 7个Taq DNA聚合酶浓度 0.2 U、0.5 U、0.7 U、1 U、1.2 U、1.5 U、2 U电泳图谱(图4)表明 Taq DNA聚合酶量过高易引起非特异性扩增,0.2 U的Taq DNA聚合酶量是经济的选择。
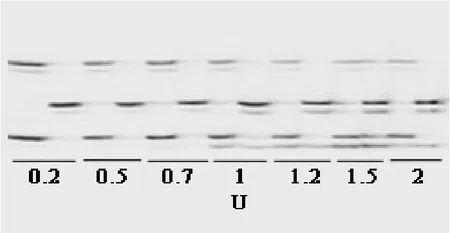
图4 Taq DNA聚合酶浓度对fSSR扩增的影响Fig. 4 Effect of Taq DNA polymerase concentration on fSSR reaction
2.1.5 PCR循环次数 本文设置20循环、25循环、30循环、35循环、40循环5个循环次数梯度。电泳显示,循环数少,检测不到目的带,从扩增条带的清晰度、特异性方面考虑,适合的循环扩增次数应不少于30循环(图5)。
2.1.6 PCR体系体积 本文设置3个反应体系体积:5 µL、10 µL、15 µL。电泳图谱(图 6)显示 3组间无明显差异,均能扩增出稳定清晰的电泳条带,基于稳定性和成本考虑,本研究选择10 µl体系。?

图5 PCR循环次数对fSSR扩增的影响Fig. 5 Effect of the number of PCR cyclers on fSSR reaction
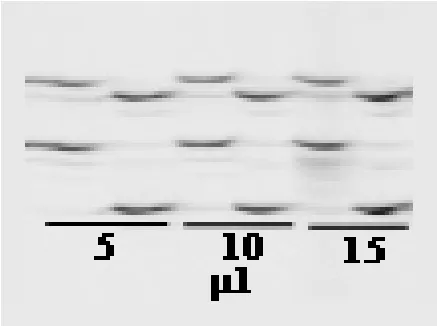
图6 PCR体系体积对fSRR扩增的影响Fig. 6 Effect of PCR total volume on fSSR reaction
2.1.7 fSRR体系的建立 依据上述确立的条件,本文建立了一套基于Gel-Scan 3000实时激光扫描电泳系统的大黄鱼fSSR分析体系(表2),并结合实际应用进行了微调。
PCR程序:95 ℃ 5 min;33个循环包括94 ℃30 s,退火温度 30 s,72 ℃ 50 s;保温 72 ℃ 15 min。
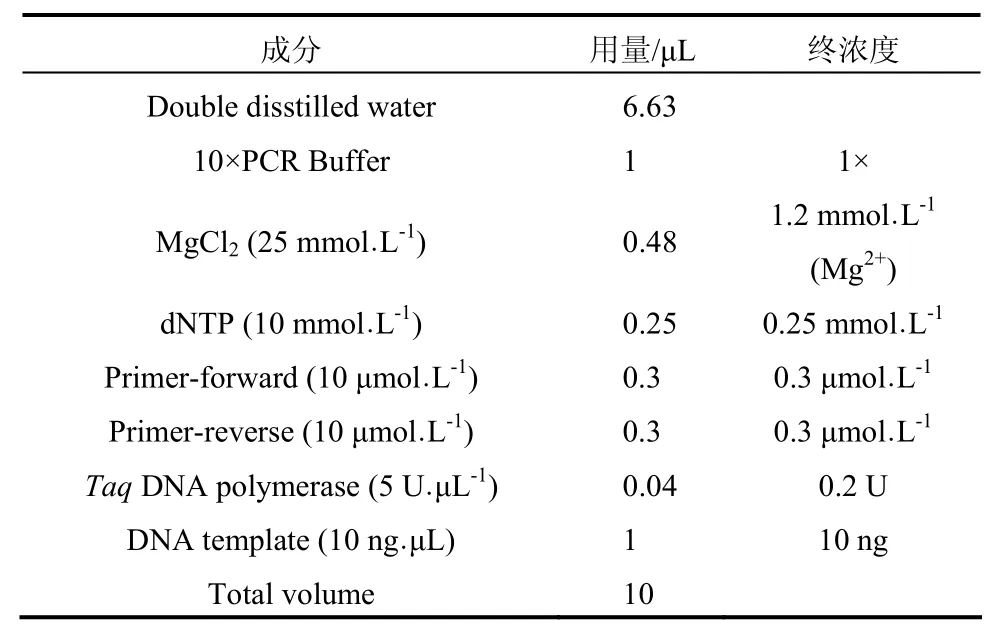
表2 大黄鱼fSSR反应体系Tab. 2 fSSR reaction system for large yellow croaker
2.1.8 fSSR体系稳定性检测 使用12尾养殖大黄鱼样品与3个大黄鱼SSR位点(表1)检测fSSR体系的稳定性。电泳图谱显示目的条带清晰整齐,重复性强(图7),3个位点分别检测到24、23、22个目的条带。
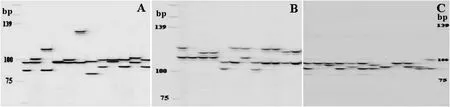
图7 基于Gel-Scan 3000的3个SSR位点电泳图谱Fig. 7 Electrophoresis pattern of three microsatellite markers based on Gel-Scan 3000(A: LYC0002, B: LYC0004, C: LYC0066)
2.2 普通聚丙烯酰胺凝胶电泳
使用普通PCR扩增体系[7]与银染方法,对上述样品与微卫星位点进行聚丙烯酰胺凝胶电泳分析,3个SSR位点(未标记HEX)在12尾大黄鱼中分别检测到23、21、20个目的条带(图8)。

图8 3个SSR位点的聚丙烯酰胺凝胶电泳图谱Fig. 8 Polyacylamide gel electrophoresis profile of three microsatellite loci(A: LYC0002, B: LYC0004, C: LYC0066)
2.3 fSSR在大黄鱼亲子鉴定中的应用
本文6对SSR引物(表1),使用本文所建立的方法,对 1#和 3#大黄鱼群体进行了亲子鉴定研究,结果在95%置信度下,1#和3#群的鉴定率均在99%(表 3)以上,显示该方法具有较好的鉴定效果。

表3 2个群的大黄鱼鉴定结果Tab. 3 Parentage results for two batches of large yellow croaker
3 讨 论
3.1 fSSR体系的建立
fSSR体系中,模板的用量是首要影响因素,其用量的多少直接影响到 PCR扩增结果[11],因此本研究首先设置了范围较广的模板浓度梯度进行优化试验。其次,Mg2+及Taq聚合酶浓度对荧光标记微卫星分析体系的影响较大,浓度过高将产生非特异性扩增。引物用量或循环次数过低将导致扩增产物量少,电泳条带暗淡;一般认为引物与模板的比例至少为108︰1才能保证PCR过程中引物与模板退火,而不是模板自身退火[12];由于不同微卫星引物的扩增效率及扩增特异性有差别,故较难准确把握PCR循环次数,通常30次循环比较合适;但由于荧光标记微卫星引物的价格高于普通引物,所以可采用小范围提高循环次数,减少引物量来降低成本,因此本文使用33个PCR循环。此外减小PCR体系体积也是降低成本的一种策略,但体积过小,一方面可能造成样本间试剂加入量不均等,扩增效果不均一,另一方面由于水分蒸发,体系中干扰物的影响变得明显,扩增效果不理想,本实验室在进行SSR分析时也证明了此点,因此本文采用10 µL体系。
3.2 荧光法与银染法检测效率与成本的比较
早期的DNA聚丙烯酰胺凝胶电泳是应用放射性核素标记完成的,但该方法操作不便,而且可使用范围局限于少数具有放射性核素设施的单位;其后主要使用银染法,该方法克服了放射性核素标记法的缺点,但银染法不仅耗时较长,显色效果有时较难把握,而且,由于正、反单链以及未解离的双链都能被染色,常导致一个等位基因产生多个银染条带,当等位基因之间差异较小时,常常与之相混杂,难以区分是来自同一个等位基因的重复条带,或者来自不同等位基因的条带。而且银染法需从胶板人工读取数据,工作繁琐,受人的主观性与误判影响导致的误差大。荧光法不仅可以使用计算机软件统计数据,自动读出等位基因大小,而且它仅标记PCR产物的单链、不带标记的反向单链不会被检出,1个等位基因电泳分析结果通常只显示唯一一条清亮的条带,弥补了银染法等存在的上述不足,是微卫星检测技术未来的发展方向之一[13]。正因为如此,本研究使用12尾大黄鱼样品与3个微卫星位点,荧光法较银染法分别多检测到1、2、2个目的条带。
本研究中银染法与荧光法完成70个SSR反应分别需费用为18.7元和19.5元,荧光法成本稍高,但银染法需多一步银染过程,银染之后需人工读取数据,效率较低。因此,考虑到时间成本和人工成本,本文建立的荧光法检测效率是银染法的 6~10倍,是一种更经济的分析方法。此外,荧光法费用较高的原因在于荧光素标记的引物是普通引物价格的 10倍左右,且不宜长期保存。因此,当需分析大量的微卫星位点时,可以考虑通过采用标记通用引物法降低成本[14]。
3.3 fSSR方法的应用
微卫星标记具有在基因组中数量多、分布较均匀、共显性遗传、多态性高以及PCR扩增稳定可靠等优点,已应用在水产生物众多研究领域。微卫星标记应用的前提是开发出多态性高的微卫星位点。在大黄鱼微卫星位点的开发研究方面,林能锋等[15]使用 PCR法筛选大黄鱼部分基因组文库,得到 2对稳定扩增的大黄鱼引物;Guo等[6]、 An等[16]与Chang等[17]则使用FIASCO法分别筛选出11对、6对与 11对多态性较高的大黄鱼微卫星位点;郝君等[18]采用生物素-磁珠富集方法筛选出30对大黄鱼微卫星位点;丁少雄等[19]使用6对眼斑拟石首鱼的微卫星引物成功在大黄鱼中进行了跨种扩增。多态性较高的微卫星标记已应用在水产生物的遗传结构及遗传多样性分析[20]、遗传连锁图谱构建[21]、分子标记辅助育种[22]以及预测有效群体大小及群体波动[23]等研究领域。
微卫星标记在水产动物亲子鉴定研究中也有较多的应用。在斑节对虾(Penaeus monodon)中,Jerry等[24]使用 7个多态性较高的微卫星位点成功鉴别出来自 13个家系的个体的亲缘关系;Neff[25]使用11个微卫星位点对美国Opinicon湖中几个蓝腮太阳鱼(Lepomis macrochirus)群体后代的系谱关系进行了鉴定;Bentzen等[26]使用多重微卫星PCR方法对 14个亲本的 135尾大鳞大马哈鱼(Oncorhynchus tshawytscha)成功进行了系谱关系分析与追踪;Rodzen等[27]使用微卫星标记分析了157尾高首鲟(Acipenser transmontanus)的亲缘关系,鉴定率超过99%。本文使用6对SSR引物,利用本文建立的fSSR技术对2个大黄鱼混合交配群进行亲子鉴定分析,鉴定率均超过99%,不仅证明了该技术在大黄鱼系谱追踪上的可行性,而且为将来更准确进行大黄鱼遗传连锁图谱构建、分子标记辅助育种、种质资源保护等研究奠定了基础。
[1] Chistiakov D A, Hellemans B, Volckaert F A M. Microsatellites and their genomic distribution, evolution, function and applications: a review with special reference to fish genetics [J]. Aquaculture, 2006,255: 1-29.
[2] 刘爱平, 胡立宪. 微卫星多态性检测时银染法和同位素技术比较 [J]. 湖南医科大学学报, 1996, 21(2):169-171.
[3] Bassam B J, Caetano-Anolles G, Gresshoff P M. Fast and sensitive silver staining of DNA in polyacrylamide gels [J]. Analytical Biochemistry, 1991, 196: 80-83.
[4] Miller K M, Laberee K, Kaukinen K H, et al. Development of microsatellite loci in pinto abalone (Haliotis kamtschatkana) [J].Molecular Ecology Notes, 2001, 1: 315-317.
[5] 朱泽远, 王亚菊, 施用晖, 等. 荧光标记微卫星分析人工饲养中华绒螯蟹的遗传多样性 [J]. 中国海洋大学学报, 2007, 37(4):591-596.
[6] Guo W, Wang Z Y, Wang Y L, et al. Isolation and characterization of six microsatellite markers in the large yellow croaker(Pseudosciaena cracea Richardson) [J]. Molecular Ecology Notes,2005, 5: 369-71.
[7] 任鹏.杂色鲍和大黄鱼微卫星标记的筛选 [D]. 厦门: 集美大学,2008.
[8] Wang Z Y, Jayasanka P, Khoo S K, et al. AFLP fingerprinting reveals genetic variability in common carp stocks from Indonesia [J]. Asian Fisheries Science, 2000, 13: 139-147.
[9] Kalinowski S, Taper M, Marshall T. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment [J]. Molecular Ecology, 2007, 16:1099-1106.
[10] 赵广泰. 大黄鱼“闽优 1号”选育群体遗传结构分析及生长相关性状的遗传参数估计 [D]. 厦门: 集美大学, 2010.
[11] 刘海云, 王敏, 王继亮, 等. 大豆 SSR技术反应体系的优化 [J].华北农学报, 2007, 22(5): 36-39.
[12] 余艳, 陈海山, 葛学军. 简单重复序列区间(ISSR)引物反应条件优化与筛选 [J]. 热带亚热带植物学报, 2003, 11(1): 19-19.
[13] Barnett B, Wike C, Dennis A, et al. High-precision genotyping by denaturing capillary electrophoresis [J]. Genome Research, 1998, 8:69-80.
[14] Schuelke M. An economic method for the fluorescent labeling of PCR fragments [J]. Nature Biotechnology, 2000, 18: 233-234.
[15] 林能锋, 许斌福, 曾红. PCR法筛选大黄鱼微卫星DNA [J]. 福建畜牧兽医, 2005, 27(2): 7-8.
[16] An H S, Cho, K C, Park J Y, et al. Eleven new highly polymorphic microsatellite loci in the yellow croaker, Pseudosciaena crocea [J].Molecular Ecology Notes, 2005, 5: 866-868.
[17] Chang Y M, Ding L, Wang W W, et al. Isolation and characterizati--on of 11 microsatellite markers for the large yellow croaker,Pseudosciaena crocea [J]. Conservation Genetics, 2009, 10:1405-1408.
[18] 郝君, 孙效文, 梁利群, 等. 大黄鱼微卫星标记的富集与筛选[J].中国水产科学, 2006, 13: 762-766.
[19] 丁少雄. 分子标记在大黄鱼遗传育种及石首鱼科分子系统进化研究中的应用 [D]. 厦门: 厦门大学, 2001.
[20] Was A, Wenne R. Genetic differentiation in hatchery and wild sea trout (Salmo trutta) in the Southern Baltic at microsatellite loci [J].Aquaculture, 2002, 204: 493-506.
[21] Ren G, Stphane M, Kamila T C, et al. A TypeⅠand TypeⅡmicrosatellite linkage map of rainbow trout (Oncorhynchus mykiss)with presump tive coverage of all chromosome arms [J].Genomics, 2006, 7: 12-13.
[22] Fuji K, Kobayashi K, Hasegawa O, et al. Identification of a single major genetic locus controlling the resistance to lymphocystis disease in Japanese flounder (Paralichthys olivaceus) [J].Aquaculture, 2006, 254: 203-210.
[23] Hitoshi A, Robin S W, William R A, et al. Effective population size of steelhead trout: influence of variance in reproductive success,hatchery programs, and genetic compensation between life-history forms [J]. Molecular Ecology, 2007, 16: 953-966.
[24] Jerry D, Evans B, Kenway M, et al. Development of a microsatel--lite DNA parentage marker suite for black tiger shrimp Penaeus monodon [J]. Aquaculture, 2006, 255: 542-547.
[25] Neff B. Genetic paternity analysis and breeding success in bluegill sunfish (Lepomis macrochirus) [J]. Journal of Heredity, 2001, 92:111-119.
[26] Bentzen P, Olsen J, McLean J, et al. Kinship analysis of Pacific salmon: insights into mating, homing, and timing of reproduction[J]. Journal of Heredity, 2001, 92: 127-131.
[27] Rodzen J, Famula T, May B. Estimation of parentage and relatedness in the polyploid white sturgeon (Acipenser transmontanus) using a dominant marker approach for duplicated microsatellite loci [J]. Aquaculture, 2004, 232: 165-182.
Condition exploration and optimization for fSSR system and its application in large yellow croaker Larimichthys crocea for parentage assignment
WU Xiang-wei, LIU Xian-de, WANG Zhi-yong
(Key Laboratory of Healthy Mariculture for East China Sea, Ministry of Agriculture of China, Fisheries College, Jimei University, Xiamen 361021, China)
In this study, the fluorescent labeling microsatellite (fSSR) analysis system was constructed and optimized in larger yellow croaker Larimichthys crocea. Although the cost of the fluorescent method was a little higher than that of the silver-staining method, it was 6~10 times efficient compared with the latter with high resolution and repetition .Furthermore, the parentage assignment rates for two groups of large yellow croaker were both more than 99% using optimized fSSR with six microsatellite loci, which means this technology is very usefull.
fSSR; Larimichthys crocea; paretage assignment; Gel-Scan 3000
Q178.53
A
1001-6932(2011)04-0419-06
2010-11-25;
2011-03-12
公益性行业(农业)科研专项(200903029-4);国家自然科学基金项目(30771663);集美大学优秀青年骨干教师基金(2009C002)。
武祥伟 ( 1984- ),男,硕士研究生,主要从事水产动物遗传育种研究。电子邮箱:wxw9559@gmail.com。
王志勇,教授。电子邮箱:zywang@jmu.edu.cn。

