表面增强拉曼散射技术鉴别大肠杆菌和志贺氏菌的研究
吕 璞,龚继来*,王喜洋,曾光明,崔 卉,胡家文 (.湖南大学环境科学与工程学院,环境生物与控制教育部重点实验室,湖南 长沙 4008;.湖南大学化学化工学院,湖南 长沙 4008)
表面增强拉曼散射技术鉴别大肠杆菌和志贺氏菌的研究
吕 璞1,龚继来1*,王喜洋1,曾光明1,崔 卉2,胡家文2(1.湖南大学环境科学与工程学院,环境生物与控制教育部重点实验室,湖南 长沙 410082;2.湖南大学化学化工学院,湖南 长沙 410082)
以整个大肠杆菌和志贺氏菌为研究对象,通过和纳米银颗粒混合制片,利用表面增强拉曼效应检测并区分大肠杆菌和志贺氏菌.结果表明,未与纳米银颗粒混合的大肠杆菌和志贺氏菌没有明显的拉曼信号,而与纳米银颗粒混合后有明显的拉曼信号,二者有明显的区别,且重现性良好,可为鉴别大肠杆菌和志贺氏菌病原体研究及实际应用提供依据.
表面增强拉曼散射;纳米银颗粒;大肠杆菌;志贺氏菌
近年来,微生物污染已经渗透到水和食物方面,而且特定微生物引发的疾病也越来越多,因此快速的微生物检测方法受到广泛关注.目前,傅里叶变换红外光谱[1],利用表面等离子共振的免疫传感器[2],用特定探针的光纤DNA芯片技术[3]及荧光光谱[4-5]均应用于微生物的快速检测.
从1974年发现至今,由于表面增强拉曼散射技术与其他方法相比,具有灵敏度高,选择性强,特异性好等优点,已被广泛用于临床医学、生物化学、生物技术、环境、考古等多个领域.随着生物技术的发展及微生物污染的严重化,利用SERS技术检测微生物的研究也越来越多.
2002年,Cao等[6]采用核酸分子杂交技术同时检测了A型肝炎、B型肝炎、艾滋病、埃博拉病毒、天花病毒和Bacillus anthracis抗原等6种病原微生物.2005年,Zhang等[7]利用SERS传感器快速检测炭疽热生物标志物. 2010年,Knauer等[8]利用SERS直接检测水环境中的嗜肺军团菌和沙门氏菌.Knauer等的研究和其他的研究有所不同,是以整个微生物为研究对象,这就是所谓的“whole-organism fingerprinting”技术[9].然而这些研究所报道的方法样品前处理比较麻烦,花费时间较长.为此,本研究在“whole-organism fingerprinting”技术基础上,以整个大肠杆菌和志贺氏菌为研究对象,把纳米银颗粒和细菌简单地混合在硅片上,利用细菌与纳米银接触时产生的 SERS快速检测与辨别大肠杆菌和志贺氏菌.大肠杆菌是环境水质监测和食品安全中重要的指示微生物[10].志贺氏菌是最常见的肠杆菌科的病原菌,可通过水和食品传播腹泻及痢疾等疾病[11].因此对这2种菌的检测具有重要意义.
1 材料与方法
1.1 试剂与仪器
分析纯试剂:无水乙醇,丙酮,氯仿,氯化钠,氢氧化钠,硝酸银,二水合柠檬酸钠;生化纯试剂:蛋白胨,琼脂粉,牛肉膏粉;pH值 7.2的磷酸盐缓冲液,硅片.
TGL-16A台式高速冷冻离心机(长沙平凡),纯水自动装置(上海申生),B220S-T型超洗器(上海必能),1-10RIBA 激光共聚焦拉曼光谱仪(JOBIN YVON,法国),微量移夜器(上海求精),恒温恒湿培养箱 LRH-250-SⅡ(广东医疗),振荡培养箱LRH-250-SⅡ(广东医疗).
1.2 细菌的购买及培养
志贺氏菌(CCTCC AB200058)从中国典型培养物保藏中心购买,大肠杆菌(DH-5α)是本实验室准备的.
大肠杆菌培养基的配制:牛肉膏粉,3g/L;蛋白胨,10g/L;琼脂粉,12g/L;氯化钠,5g/L;pH,7.2~7.4.志贺氏菌培养基的配制:牛肉膏粉,3g/L;蛋白胨,5g/L;琼脂粉,20g/L;氯化钠,5g/L;pH,7.0. 2种菌分别按照各自的培养基在生化培养箱中37℃培养 24h,驯化 3次后,然后用液体培养基(不加琼脂粉)在生化振荡培养箱中 37℃培养48h.
1.3 纳米银颗粒的合成
纳米银颗粒采用文献[12]合成:将250mL的5.29×10-4mol/L的硝酸银水溶液加热至沸腾,在剧烈搅拌下迅速加入7mL质量分数为1%的柠檬酸钠水溶液,保持沸腾状态反应 1h后,自然冷却至室温,溶液呈黄绿色.将制好的溶液放于冰箱内4℃保存.
1.4 样品的制备及测定
1.4.1 硅片的洗涤 将切割好的小硅片放在小烧杯中,把小烧杯置于超声波清洗机中,依次用超纯水—乙醇—丙酮—氯仿—丙酮—乙醇—超纯水清洗,每种溶液都清洗8min.
1.4.2 细菌的处理 液体培养基培养后的大肠杆菌和志贺氏菌,取适量培养液 4000r/min离心10min,弃上清液,用pH值7.2的磷酸盐缓冲液清洗两次,然后用无菌水清洗若干次后加入无菌水稀释到1mL.
1.4.3 纳米银颗粒的处理 取上述制备的纳米银溶液30mL加入到50mL的离心管里,加入少量的表面活性剂,4000r/min离心 5min,弃上清液至剩余液体为1mL.
1.4.4 测定样品的制备 取上述处理过的细菌液0.5mL和离心过的纳米银溶液0.5mL加入到1.5mL的离心管,充分混合后,4000r/min离心5min,弃上清液至剩余溶液约为 50μL.然后把这个混合液体取5μL滴加到清洗过的小硅片上.晾干后测定拉曼信号.
1.4.5 拉曼信号的测定 用镊子将小硅片放到载物台的玻璃片,直接用50倍长聚焦物镜进行聚焦,测量时波长扫描范围为:400~2000cm-1,扫描 1次.扫描时间为 1s,滤光片密度为 1D,进行mapping扫描,激发波长为632nm.
1.4.6 空白实验 按照上述测量过程分别测定(1)干净的硅片的拉曼信号;(2)取0.5mL离心过的纳米银再在相同条件下离心一次的拉曼信号;(3)离心处理过的不加纳米银的 2种菌的拉曼信号.
2 结果与分析
2.1 纳米银颗粒的TEM表征

图1 纳米银颗粒的TEM图谱Fig.1 TEM image of silver colloid
纳米银颗粒的制备采用柠檬酸钠还原法制备,尺寸为 40~100nm,平均尺寸约为 55nm,其透射电子显微镜如图1所示.纳米银的合成受还原剂加入量的影响,其粒径随着柠檬酸钠加入量的增加而变大,形貌也逐渐不规则[13].因此可以从图中观测到纳米银的形状多样化,有圆形,棒状,三角形.
2.2 细菌的SEM表征
由图2可以看出,大肠杆菌和志贺氏菌都是杆状的,大肠杆菌平均长度为 2.83μm,志贺氏菌平均长度为3.34μm.
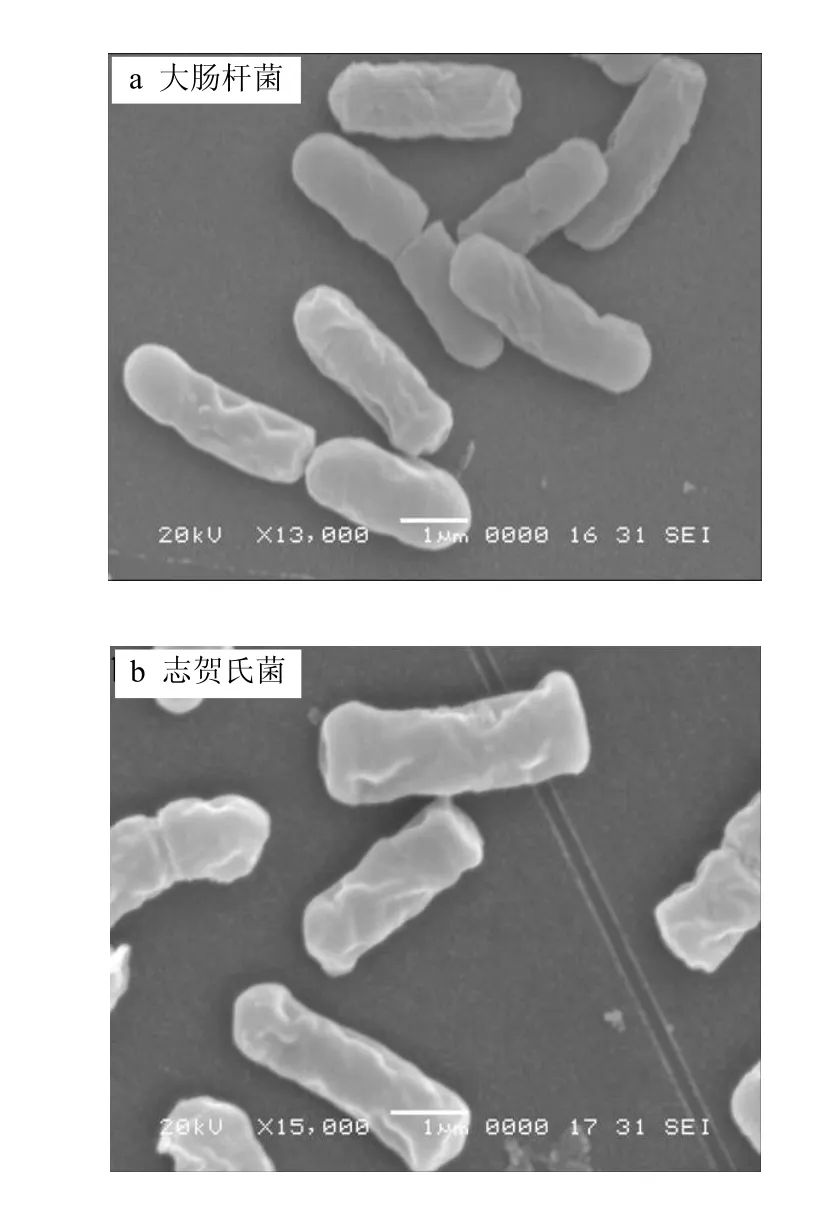
图2 大肠杆菌和志贺氏菌的SEM图谱Fig.2 SEM image of Escherichia coli and Shigella spp.
2.3 细菌和纳米银颗粒混合的TEM表征
图 3是大肠杆菌和志贺氏菌与纳米银颗粒混合液稀释若干倍的情况下测得,从图3可以看出,细菌被纳米银颗粒包围,可以充分发挥纳米银颗粒的增强效果.

图3 大肠杆菌和志贺氏菌与纳米银颗粒混合的TEM图谱Fig.3 TEM image of the mixture of Escherichia coli and Shigella spp. and silver colloid
2.4 两种细菌不同SERS信号分析
由图 4可见,纳米银颗粒都几乎没有拉曼信号,而干净的硅片在 520cm-1处有特定的硅信号.虽然未与纳米银颗粒混合的大肠杆菌和志贺氏菌的光谱图有很大区别(图 4),但是单独的大肠杆菌和志贺氏菌的拉曼谱峰很少且信号很弱,和纳米银颗粒混合后纳米银颗粒围绕在细菌周围,拉曼谱峰增加,且信号得到增强.与纳米银混合后的两种菌的拉曼信号有明显的区别,除了520cm-1处的硅信号,大肠杆菌分别在 658cm-1,728cm-1, 960cm-1左右,1000cm-1左右,1330cm-1左右, 1460cm-1左右,1570cm-1左右有明显的信号, 728cm-1的峰说明了大肠杆菌中含有 N-乙酰-D-葡萄糖胺和N-乙酰胞壁酸[14].在658cm-1的峰和Jarvis等[14]的研究结果相符合.而 1460cm-1和1570cm-1峰和Jarvis等[15]的另一项研究结果相符,且 Jarvis等在文献[15]中报道大肠杆菌并没有出现658cm-1这个峰,这可能是由于拉曼测定条件的不同,正如Jarvis等[16]叙述:一样的大肠杆菌,就因为一个在波长 785nm测定拉曼信号,一个在532nm测定拉曼信号,两者的信号峰位置大有不同.实验拉曼信号的测定是在波长632nm,而Jarvis等是分别在波长785nm,532nm测定.志贺氏菌分别在 670cm-1左右,820cm-1左右,1330cm-1左右,1570cm-1左右有明显的信号.两种菌最明显的区别在600~700cm-1之间.通过分析可以看出实验可以测定并鉴别大肠杆菌和志贺氏菌2种菌.

图4 纳米银和2种菌混合后及空白实验的拉曼光谱Fig.4 Raman mapping spectra of bacteria and blank
2.5 硅片上的重现性实验
分别对 2种菌的同一样品和不同时间的样品做mapping扫描分析,同一样品做平行实验,每个样品测25个点,两个平行样50个点,有如图5(a)和图6(a)所示的拉曼信号的有32个点左右,不同时间的样品间隔大概半个月左右测量拉曼信号,如图5(b)和6(b)所示.通过对比可以看出2种菌在硅片上的拉曼信号有良好的重现性.
实验中细菌是在水环境条件下,并且大肠杆菌和志贺氏菌都是水环境中常见的菌株,因此实验条件与实际相接近,能够为大肠杆菌和志贺氏菌的检测方法的实际应用提供依据.
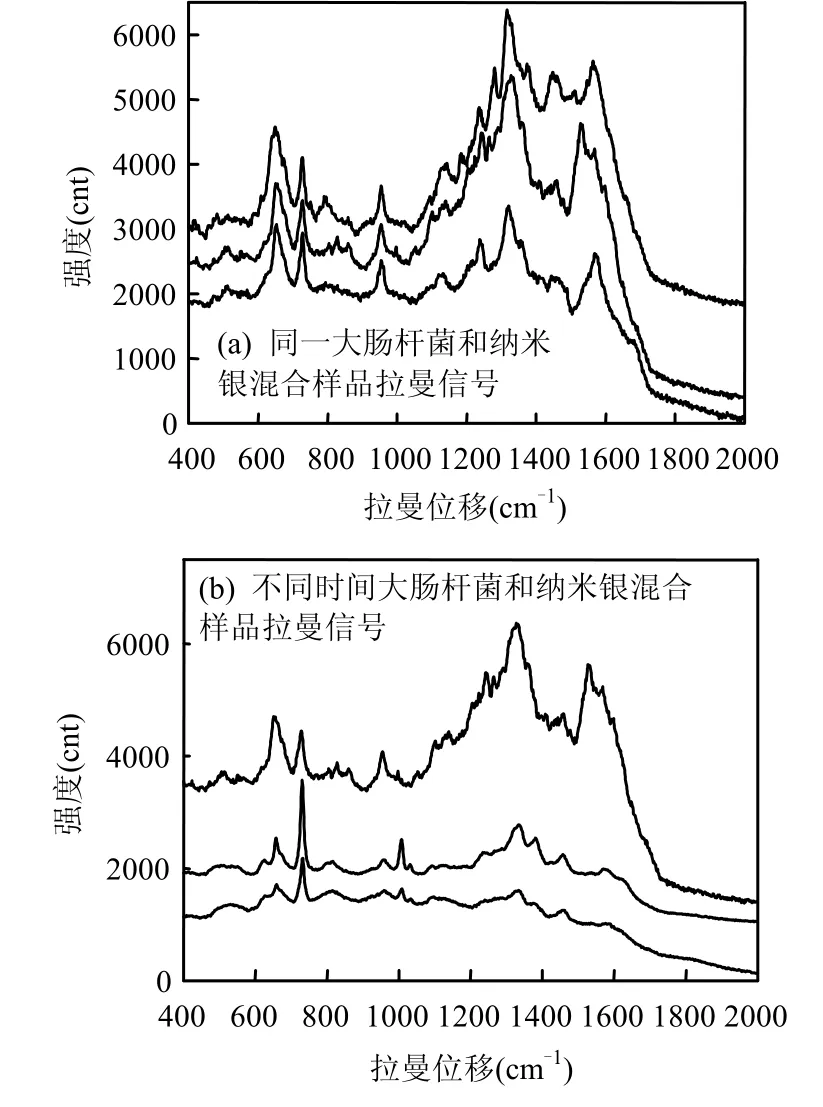
图5 大肠杆菌重现性拉曼光谱Fig.5 Raman mapping spectra of Escherichia coli

图6 志贺氏菌重现性拉曼光谱Fig.6 Raman mapping spectra of Shigella spp.
3 结论
3.1 单独的大肠杆菌和志贺氏菌的拉曼谱峰很少且信号很弱,和纳米银颗粒混合后纳米银颗粒围绕在细菌周围,拉曼谱峰增加,且信号得到增强.
3.2 大肠杆菌和志贺氏菌与纳米银颗粒混合后测得的拉曼信号有明显区别,说明这种方法能够鉴别两种菌.600~700cm-1之间区别最明显,大肠杆菌在658cm-1处的拉曼信号峰和以前的研究相符合,由于测定条件不同,大肠杆菌在 728cm-1也有峰,这个峰说明大肠杆菌中含有N-乙酰-D-葡萄糖胺和N-乙酰胞壁酸.
3.3 两种菌的同一样品和不同时间的样品的拉曼信号有良好的重现性.
[1] Irudayaraj J, Yang H, Sakhamuri S. Differentiation and detection of microorganisms using Fourier transform infrared photoacoustic spectroscopy [J]. J. Mol. Struct., 2002,606(1-3):181-188.
[2] Oh B K, Kim Y K, Lee W, et al. Immunosensor for detection of Legionella pneumophila using suface plasmon resonance [J]. Biosens. Bioelectron., 2003,18(5/6):605-611.
[3] Ahn S, Walt D R. Detection of Salmonella spp.using microsphere-based, fiber-optic DNA microarrays [J]. Anal.Chem., 2005, 77(15):5041-5047.
[4] Estes C, Duncan A, Wade B, et al. Reagentless detection of microorganisms by intrinsic fluorescence [J]. Biosens. Bioelectron., 2003,18(5/6):511-519.
[5] Podrazky O, Kuncova G. Determination of concentration of living immobilized yeast cells by fuorescence spectroscopy [J]. Sens. Actuators B, 2005,107(1):126-134.
[6] Cao C Y W, Jin R, Mirkin C A. Nanoparticles with Raman spectroscopic fingerprints for DNA and RNA detection [J]. Science, 2002,297(30):1536-1540.
[7] Zhang X,Young M A, Lyandres O, et al. Rapid detection of an Anthrax Biomarker by surface-enhanced Raman [J]. J. Am. Chem. Soc., 2005,127(12):4484-4489.
[8] Knauer M, Ivleva N P, Liu X, et al. Surface-enhanced Raman scattering-based label-free microarray readout for the detection of microorganisms [J]. Anal.Chem., 2010,82(7):2766-2772.
[9] Goodacre R,Timmins E M, Burton R, et al. Rapid identification of urinary tract infection bacteria using hyperspectral wholeorganism fingerprinting and artificial neural networks [J]. J. Microbiol., 1998,144(5):1157-1170.
[10] 王 娜,何 苗,施汉昌.环境中大肠杆菌快速生物检测技术研究进展 [J]. 安全与环境学报, 2006,6(3):127-131.
[11] 黄宝华,陈庆森,庞广昌.志贺氏菌研究及其快速检测技术发展现状 [J]. 食品科学, 2004,25(11):333-336.
[12] Lee P C, Meise D. Adsorption and surface-enhanced Raman of dyes on silver and gold sols [J]. J. Phys. Chem., 1982, 86(17): 3391-3395.
[13] 张玲艳.Ag溶胶和ZnO基底的表面增强拉曼光谱研究 [D]. 南京:南京理工大学, 2010,1-70.
[14] Jarvis R M, Goodacre R. Discrimination of bacteria using surface-enhanced Raman spectroscopy [J]. Anal. Chem., 2004, 76(1):40-47.
[15] Jarvis R M, Brooker A, Goodacre R. Surface-enhanced Raman spectroscopy for bacterial discrimination utilizing a scanning electron microscope with a Raman spectroscopy interface [J]. Anal. Chem., 2004,76(17):5198-5202.
[16] Jarvis R M, Goodacre R. Characterisation and identification of bacteria using SERS [J]. Chem. Soc. Rev., 2008,37(5):931-936.
Detection and discrimination of Escherichia coli and Shigella spp. using surface-enhanced Raman spectroscopy.
LÜ Pu1, GONG Ji-lai1*, WANG Xi-yang1, ZENG Guang-ming1, CUI Hui2, HU Jia-wen2(1.Key Laboratory of Environmental Biology and Pollution Control,Ministry of Education, College of Environmental Science and Engineering, Hunan University, Changsha 410082, China;2.College of Chemistry and Chemical Engineering, Hunan University, Changsha 410082, China). China Environmental Science, 2011,31(9):1523~1527
E. coli and Shigella spp. were detected and differentiated using surface-enhanced Raman scattering (SERS) by preparing bacteria samples with silver colloids on a silicon surface. Results show that Raman signals of E. coli and Shigella spp. were negligible in the absence of silver nanoparticles, but observable and distinguishable spectra were collected while in the presence of silver nanoparticles. Thus, E. coli and Shigella spp. could easily be discriminated based on their different Raman spectra. The method in this work exhibited high reproducibility and showed great promise for the detection of E. coli and Shigella spp. in environmental samples.
surface-enhanced Raman scattering; silver colloid; Escherichia coli; Shigella spp.
X172
A
1000-6923(2011)09-1523-05
2011-01-20
教育部新世纪优秀人才支持计划项目(NCET-09-0328)
* 责任作者, 副教授, jilaigong@hnu.cn
吕 璞(1986-),女,河南许昌人,湖南大学环境科学与工程学院硕士研究生,主要从事水质监测新技术研究.发表论文1篇.

