白地霉分批发酵产脂肪酶动力学模型的建立
王 挥 袁 强,2 张 敏 黎继烈 李忠海
白地霉分批发酵产脂肪酶动力学模型的建立
王 挥1袁 强1,2张 敏1黎继烈1李忠海1
(中南林业科技大学生命科学与技术学院1,长沙 41004)
(湖南未名创林生物能源有限公司2,长沙 410004)
对脂肪酶产生菌白地霉PP1315在5 L自控发酵罐中的分批发酵进行了动力学分析。探讨了PP1315在5 L罐分批发酵过程中菌体生长、脂肪酶合成及基质消耗的变化规律,结果表明,菌体生长呈典型S型曲线,酶的合成与菌体生长趋势呈现部分相关性,属于部分偶联型。基于这些过程曲线的变化规律,结合Logistic方程和Luedeking-Piret方程,建立了脂肪酶产生菌PP1315分批发酵过程的动力学模型,并通过数学推导和非线性拟合获得了模型中各参数的最佳取值,脂肪酶合成动力学模型的相关系数为R2=0.925,确定了能够较好表征PP1315分批发酵过程中菌体生长、产物脂肪酶合成以及基质消耗的动力学方程。
脂肪酶 分批发酵 动力学模型 非线性拟合
脂肪酶(Lipase,EC3.1.1.3)是一类重要的甘油酯键水解酶,可以在油水界面上催化三酰甘油水解生成脂肪酸和甘油,以及中间产物甘油一酯和甘油二酯[1]。脂肪酶的产业化主要来源于微生物发酵[2],目前工业上采用微生物发酵制备和提取脂肪酶应用于食品、医药、洗涤、皮革、造纸等行业[3]。近年来因其可催化各类化学选择性、区域选择性、立体结构选择性转化反应等特点,促使脂肪酶在有机合成中得到广泛应用[4-5]。随着矿化石油资源日益枯竭和人们生态环境保护意识的提高,使得脂肪酶催化合成生物柴油作为可再生替代环保燃料为世人瞩目[6-8]。
采用微生物发酵产脂肪酶的过程具有多样性、耦合性、时变性和非线性等特点,既包括了细胞内各种各样的生化反应,也涉及细胞间各种物质交换的复杂体系[9],由此可见对微生物发酵规模放大过程规律的研究,是发酵产品能否实现工业化生产的关键[10]。根据张敏[11]对白地霉 PP1315 摇瓶发酵的研究可知,白地霉PP1315发酵是一个比较复杂的过程,因此深入探讨白地霉PP1315在发酵罐中的分批发酵条件和动力学特征,对于促进脂肪酶的规模化生产很有必要。本研究通过建立白地霉PP1315分批发酵动力学模型,旨在实现白地霉PP1315产脂肪酶的发酵过程优化控制,降低生产成本,为进一步放大试验和连续发酵提供理论基础。
1 材料与方法
1.1 材料、试剂与仪器
白地霉(Geotrichum candidum)PP1315:由本课题组经多次诱变选育并保藏于发酵工程实验室。
斜面培养基:麦芽糖40 g/L,蛋白胨10 g/L,琼脂粉15 g/L。
种子培养基:葡萄糖2.0 g/L,蛋白胨 2.5 g/L,K2HPO40.1 g/L,橄榄油1.0 mL/L。
发酵培养基:玉米粉15 g/L,豆饼粉10 g/L,橄榄油 10 ml/L,K2HPO41 g/L,MgSO4·7H2O 1.5 g/L。
B型SL 5 L全自动控制发酵罐:上海保兴生物设备工程有限公司;Avanti J-E高速冷冻离心机:美国贝克曼公司。
1.2 试验方法
1.2.1 种子制备
500 mL摇瓶装种子培养基100 mL,接斜面活化好的菌种两环,在28℃、摇床转速180 r/min条件下培养24 h,备用。
1.2.2 分批发酵
在5 L全自动发酵罐中装发酵培养基3 L,并加入0.05%的泡敌,在0.1 MPa、121℃的的条件下灭菌20 min,冷却后接种 5%(体积比),发酵温度28 ℃,pH 6.0,直到发酵结束。
用复膜氧电极和pH电极在线测定发酵过程中的溶氧和pH变化,发酵过程中流加6 mol/L NaOH以控制pH,通过调节通气量约1.0 L/min,搅拌转速200~500 r/min,控制溶氧水平在20%以上,并在有必要时及时流加泡敌。每6 h取一次样,测定总糖、菌体生物量和脂肪酶酶活,每次做3个平行试验,取其平均值(相对误差小于5%)。
1.2.3 分析方法
总糖的测定采用3,5-二硝基水杨酸(DNS)法[12]。
脂肪酶的活力测定采用碱滴定法[13]。
菌体生物量的测定采用干重法,取5 mL发酵液离心后,用蒸馏水洗2次,再离心,60℃烘箱内烘干至恒重后称重。
1.2.4 数据处理
模型参数的优化及动力学参数的获取均运用Origin 7.5对动力学方程非线性拟合获得。
2 结果与分析
2.1 脂肪酶发酵过程代谢变化特征
在分批发酵过程中,白地霉PP1315脂肪酶发酵过程代谢变化如图1所示。在0~18 h,菌体生长延迟期
,
该段时间内菌体碳源消耗少,菌体生物量增加较慢,脂肪酶产量为0,18 h后菌体进入对数期,菌体耗糖量明显上升,脂肪酶产量随着菌体的生长迅速增加,60 h进入稳定期后,菌体生物量几乎不增加,耗糖量减少,但是脂肪酶产量继续上升,96 h是活力达到最高240.15 U/mL,因此该脂肪酶的合成属于生长部分耦联型。96 h后脂肪酶合成速率也开始下降,108 h停止发酵。
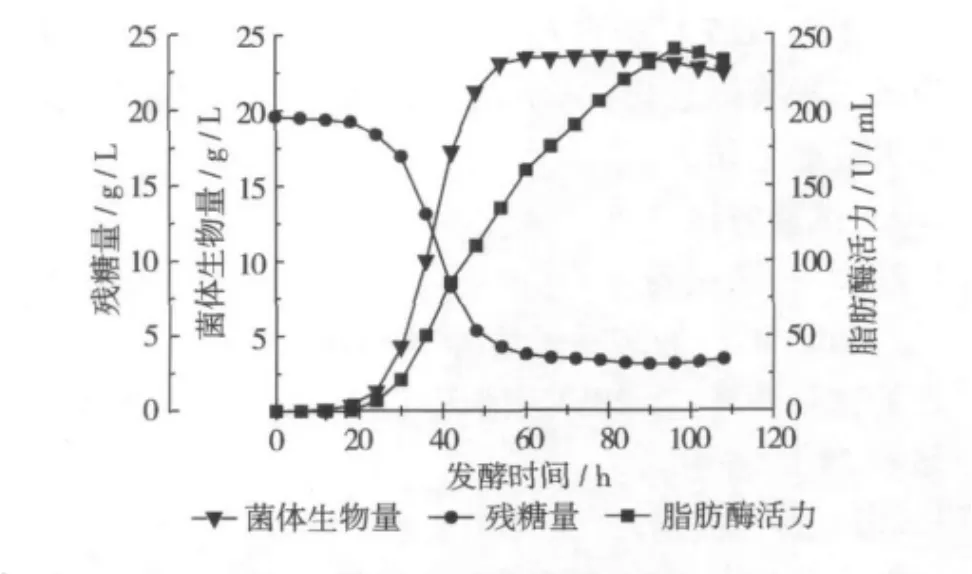
图1 白地霉PP1315脂肪酶发酵过程曲线
2.2 菌体生长动力学模型
对于细胞生长的动力学模型,由于在分批发酵过程中底物及菌体浓度对生长的限制作用不容忽略,故不宜采用Monod方程来描叙分批发酵菌体模型,目前多采用非结构模型来描述,Logistic方程能很好地反映分批发酵过程中菌体生长的一般规律,是目前应用较为普遍的方程之一,常用于细胞生长动力学的描述。同时,由于白地霉PP1315细胞生长曲线为S形,因此,将其应用于白地霉PP1315液体发酵中,可得白地霉PP1315生长的Logistic方程,其可积分为代数方程:

Logistic方程可积分为代数方程:

式(2)中:μm为菌体最大生长速率;X为菌体生物量/g/L;X0为初始菌体生物/g/L;Xm为最大菌体生物量/g/L;t为时间/h。
运用Origin 7.5对菌体生长试验值按照公式(2)进行非线性拟合,结果如图2。

图2 菌体生长的试验值与模型计算值的比较
菌体干质量(X)随发酵时间变化的函数为:

式中:X0=0.008 96 g/L,Xm=23.587 g/L,μm=0.210 83。从图2可知,式(3)的计算值可以较好的与试验数据相吻合R2=0.988,该方程能够作为白地霉PP1315脂肪酶合成优化控制的参考依据,发酵结束前期菌体自溶,导致试验值与拟合值在发酵后期有较大的偏差。
2.3 脂肪酶合成动力学
在微生物的分批发酵过程中,细胞内的生物合成途径十分复杂,其代谢调节机制特点各异。Gaden[14]根据产物生成速率与细胞生长速率之间的关系,将产物的形成与微生物细胞生长关系的动力学模式分为三类:Ⅰ产物形成与细胞生长偶联型;Ⅱ产物形成与细胞生长部分偶联型;Ⅲ产物形成与细胞生长非偶联型。Luedeking和 Piret[14]对此进行了总结,提出了著名的Luedeking-Piret方程用来描述产物形成与细胞生长的关系。
Luedeking-Piret方程:

式中:α≠0、β=0表示第Ⅰ类发酵;α≠0、β≠0表示第Ⅱ类发酵;α=0、β≠0表示第Ⅲ类发酵。从白地霉PP1315分批发酵试验结果可以判断,白地霉PP1315产脂肪酶的发酵过程属于产物形成与细胞生长呈部分偶联型,结合式(2)和式(4),Luedeking-Piret方程可积分为代数方程:

式中:P为脂肪酶酶活/U/mL;P0为初始脂肪酶酶活/U/mL;α为生长耦联系数;β为非生长耦联系数。
由于0 h时,P0=0,X0对产物的影响可以忽略不计,方程5可以简化为:

运用Origin 7.5对白地霉PP1315脂肪酶合成发酵试验值按照式(6)进行非线性拟合,结果如图3所示,得到脂肪酶合成量(P)随发酵时间变化的函数:
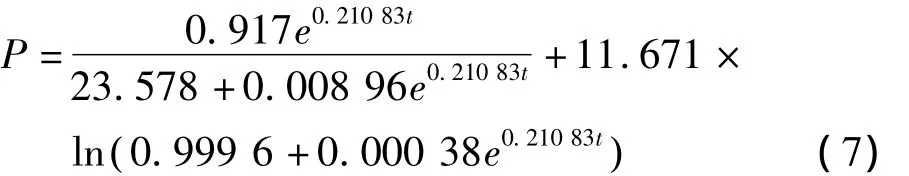
其中 α =4.346 36,β =0.104 36,在图 3 可知式(7)能够较好的拟合所得试验数据(R2=0.925),较好的描叙了发酵过程中脂肪酶的合成情况,脂肪酶在合成后期,发酵液中蛋白酶以及其他限制性因素的积累,脂肪酶缓慢变性导致失活,故在发酵后期试验值与拟合值有所偏差。

图3 脂肪酶活的试验值与模型计算值的比较
2.4 基质消耗动力学
在白地霉PP1315脂肪酶发酵过程中,碳源的消耗主要有3个方面:一是用于细胞生长,二是细胞维持基本生命活动消耗,三是用于合成产物。基质消耗可以用与Luedeking-Piret相似的方程表示。
在分批发酵过程中,碳源的消耗可用如下动力学模型表示:

结合方程式(4)、(5)和(8)积分可得:
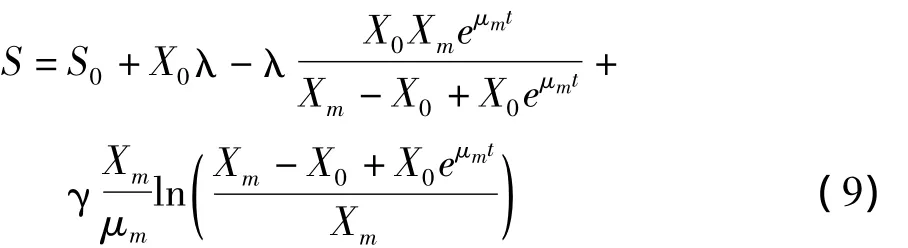
式中:S为总糖浓度/g/L;YX/S为碳源用于菌体生长的得率常数,λ=1/YX/S;γ为碳源用于产物积累的得率常数/h-1;
按照式(9)对发酵过程中总糖值进行非线性拟合,结果如图4所示,得到基质消耗量随发酵时间变化的方程:

式中:S0=19.584 g/L,λ =0.664 07,γ =-0.000 54。式(10)能够较好的描叙脂肪酶合成过程中底物消耗的情况(R2=0.975),由图4可以看出发酵后期由于少量菌体自溶导致发酵后期试验值与拟合值有一定偏差。
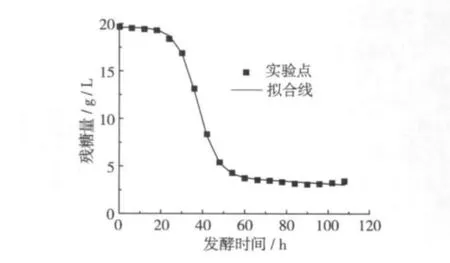
图4 基质消耗的试验值与模型计算值的比较
2.5 模型的验证
为了检验模型的误差,在相同试验条件下进行3次发酵罐放大培养,所得试验数据取平均值,实测值与模型预测计算值进行比较,结果见表1。模型预测计算值与实测值误差在发酵开始期及其发酵后期较大,在发酵过程中误差较小,总体来说绝大部分误差低于10%,模型计算值能够较好的反映实际发酵过程中的试验值,因此该发酵动力学模型能够较好地描叙白地霉PP1315产脂肪酶发酵过程中菌体生长、基质消耗和产物合成的变化规律。
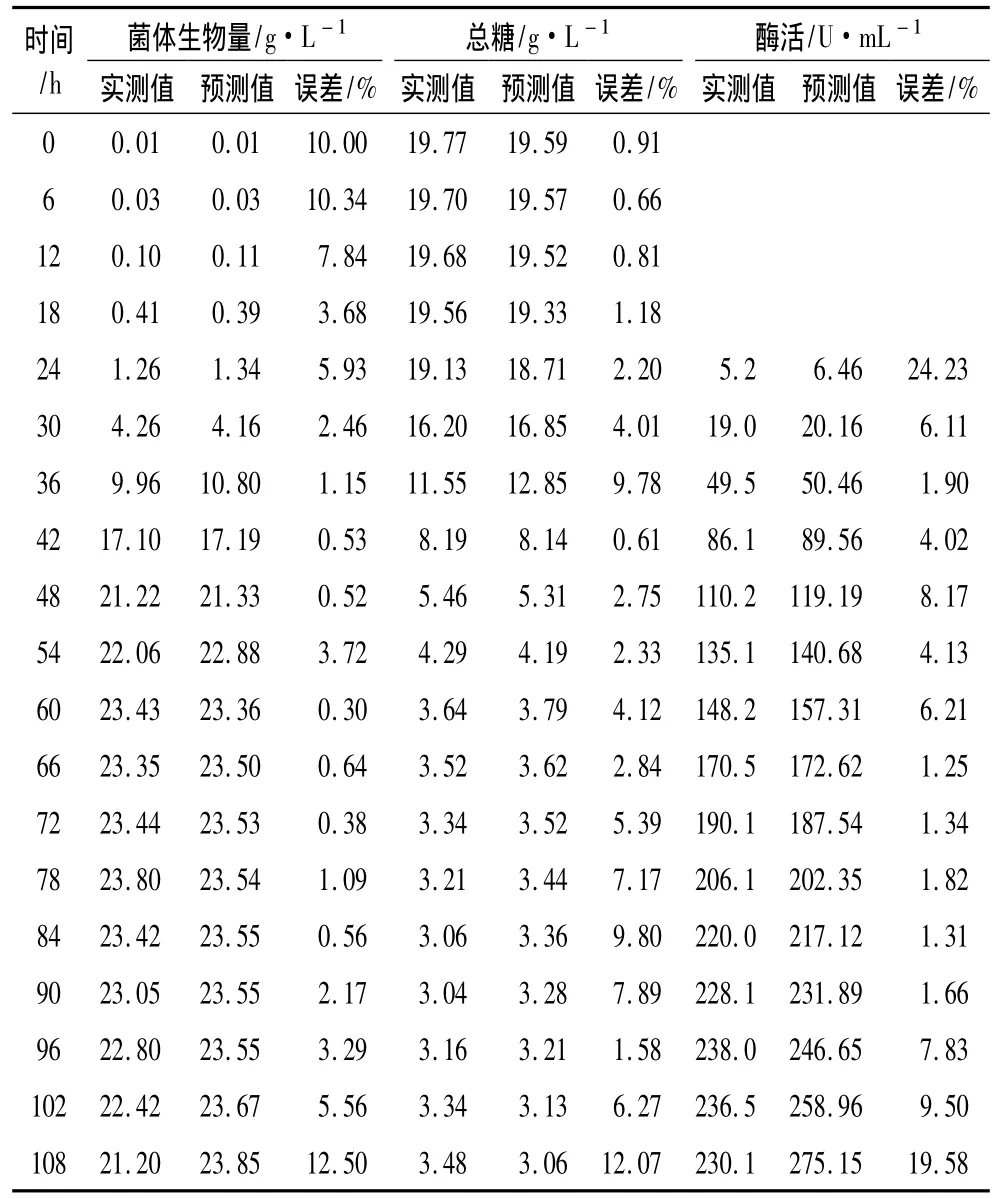
表1 试验值与预测计算值的比较
3 结论
本研究分别利用 Logistic方程和 Luedekingpiret方程描述了白地霉PP1315产脂肪酶的分批发酵过程,建立了菌体生长、脂肪酶合成和基质消耗的动力学模型。分别为:

(1)菌体生长动力学模型(2)脂肪酶合成动力学模型

(3)基质消耗动力学模型

试验结果显示,试验值能够与模型预测值较好拟合,菌体生长动力学模型的相关系数为 R2=0.988,基质消耗动力学模型的相关系数为 R =0.975,由于在发酵后期脂肪酶的缓慢失活,脂肪酶实际活力与模型理论值存在一定差异,脂肪酶合成动力学模型的相关系数为R2=0.925,但是总体上而言该模型的建立可以解释白地霉PP1315脂肪酶动力学规律,可为实际发酵过程中菌体生长、产物合成及基质消耗的预测与控制提供参考。
[1]Norin M,Olsen,Svendsen A.Theoretical studies of Rhizomucor miehei lipase activation[J].Protein Engineering,1993,6(8):855-863
[2]Elibol M,Ozer D.Influence of oxygen transfer on lipase production by Rhizopus arrhizus[J].Process Biochemistry,2000,36:325 -329
[3]Rohit S,Uttam C B.Production,purification,characterization,and applications of lipases[J].Biotechnol Ogy Advances,2001,19:627 -662
[4]Jaeger K E,Eggert T.Lipases for bioTech-nology[J].Current Opinion in BioTech-nology,2002,3(4):390 -397
[5]Kazlauskas R J,Bornscheuer U T.Biotransformations with lipases[J].BioTech-nology,1998,8:37 -50
[6]Glória P M,Mudge SM.Cleaning oiled shores:laboratory experiments testing thepotential use of vegetable oil biodiesels[J].Chemosphere,2004,54:297 -304
[7]Wardle D A.Global sale of green air travel supported using biodiesel[J].Renewable and Sustainable Energy Reviews,2003,7:1 -64
[8]Iso M,Chen B,Eguchi M.Production of biodiesel fuel from triglyceride andalcohol using i- mmobilized lipase[J].Journal of Molecular Catalysis B:Enzymatic,2001,16:53 -58
[9]刘朝辉,陈云,齐崴,等.中性-甘露聚糖酶分批发酵动力学研究[J].化学工程,2008,36(10):66 -71
[10]张建国,陈晓明,贺新生.灵芝胞外多糖分批发酵动力学模型[J].生物工程学报,2007,23(6):1065 -1070
[11]张敏.产脂肪酶菌株的选育、发酵条件优化及其酶学性质研究[D].长沙:中南林业科技大学,2010
[12]Huang JX,Yang JS,Wei Y.Dynamic models for polyhydroxybutyrate fermentation process[J].Chemical engineering,2005,33(1):44 -47
[13]江慧芳,王雅琴,刘春国.三种脂肪酶活力测定方法的比较及改进[J].化学与生物工程.2007,8(24):72-74
[14]戚以政,汪叔雄.生化反应动力学与反应器[M].北京:化学工业出版社,1999:140-147.
Establishment of Batch Fermentation Kinetics of Lipase Producing Strain Geotrichum Candidum
Wang Hui1Yuan Qiang1,2Zhang Ming1Li Jilie1Li Zhonghai1
(College of Life Science and Tech-nology,Central South University of Forestry and Tech-nology1,Changsha 410004)
(Hunan Weiming Chuanglin Bio - energy Co,.Ltd2,Changsha 410004)
The kinetics of lipase batch fermentation process was studied by Geotrichum candidum(PP1315)in a 5L stirred bioreactor.Firstly cell growth,product formation and substrate consumption throughout the lipase fermentation were investigated.The result showed that cell growth of PP1315 was characterized as an S - type curve,which presented a partial correlation with lipase synthesis.So,the partial coupling model was concluded.Based on variations of these process curves,the kinetics models for cell growth,lipase synthesis and substrate consumption were established according to the Logistic equation and Luedeking-Piret equation.Through the nonlinear regression and mathematical derivation,the optimal parameters in the above models were obtained.Finally,the kinetics equations of cell growth,lipase synthesis and substrate consumption,which was characterized in the batch fermentation process of PP1315,were obtained.
lipase,batch fermentation,kinetics model,nonlinear regression
Q814
A
1003-0174(2011)10-0061-05
国家林业局948计划(2010-4-22),国家科技支撑计划(2009BADB1B10)
2010-12-23
王挥,男,1974年出生,博士,食品生物技术
黎继烈,女,1959年出生,教授,食品生物技术

