正链RNA病毒复制酶结构与功能研究进展
王 超 ,吴 润,刘光清
(1.中国农业科学院上海兽医研究所,上海 200241;2.甘肃农业大学动物医学院,兰州 730070)
正链RNA病毒是一类基因组为单股正链RNA分子的病毒,约占病毒总分类的1/3以上,种类数目非常庞大,例如常见的小RNA病毒科(Piconaviridae)、嵌杯病毒科(Caliciviridae)、星状病毒科(Astroviridae)、野田村病毒科(Nodaviridae)、黄病毒科(Flaviviridae)、披膜病毒科(Togaviridae)以及一些重要的植物病毒等,感染宿主谱非常广泛。除逆转录病毒外,所有正链RNA病毒都编码有RNA依赖性RNA聚合酶(RNA-dependent RNA polymerase, RdRp)[1],它与病毒编码的一些蛋白酶(如解旋酶)以及宿主细胞因子一起构成病毒的复制酶系统[2,3],负责病毒基因组的复制。其中,RdRp是复制酶系统中的核心成分。因此,研究RdRp的结构与功能对于阐明正链RNA病毒的复制和调控机理等具有重要意义。近年来,关于RdRp的结构与功能研究已取得了许多令人瞩目的进展,本文即对相关研究进展进行综述。
1 RdRp概述
根据RdRp氨基酸序列的相似性和种系发生分析,大致可以将其划分为三个大的超群(Ⅰ、Ⅱ和Ⅲ)。其中Ⅰ群的代表病毒有小RNA病毒、野田村病毒、豇豆花叶病毒、线虫多面体病毒等;Ⅱ群的代表病毒有香石竹斑驳病毒、瘟病毒、丙型肝炎病毒、单链RNA噬菌体等;Ⅲ群的代表病毒有烟草花叶病毒、甲病毒、风疹病毒、戊型肝炎病毒等[1]。三个超群的RdRp都缠绕折叠形成一个准确的包围着八个保守基序(Ⅰ-Ⅷ)的结构,其中四个基序存在于各类的聚合酶中,包括DNA依赖的RNA聚合酶(DdRp)、DNA依赖的DNA聚合酶(DdDp)和RNA依赖的DNA聚合酶(RdDp)[4-5]。在这八个保守基序中,只有五个氨基酸残基是所有聚合酶中完全保守的,它们分别是基序Ⅰ中的Lys3、基序Ⅳ中的Asp118和Asp124及基序Ⅵ中的Asp268和Asp269。当然,这三个超群的聚合酶在序列上也是有相互关系的,虽然有些氨基酸残基较为保守,比如,一个超群中的一些保守性残基也存在于其他的超群中。RdRp与病毒蛋白和一些宿主细胞因子一起组成复制复合物,共同执行复制酶功能。例如,脊髓灰质炎病毒的复制复合物包含宿主的Poly(C)和Poly(A)结合蛋白(分别为PCBP和PABP)、多聚酶前体3CD[6]和EF-1α[7]等。
2 RdRp的氨基酸序列与高级结构
2.1 RdRp的氨基酸序列特征目前,一些重要RNA病毒的RdRp的结构与功能研究已经取得重大进展。如脊髓灰质炎病毒(Poliovirus,PV)[8]、丙型肝炎病毒(hepatitis C virus,HCV)[9]、兔出血症病毒(Rabbit hemorrhagic disease virus,RHDV)[10]、口蹄疫病毒(Foot-and-mouth disease virus,FMDV)[11]等的RdRp晶体结构已基本阐明。RdRp结构域的N端残基包含了部分多肽链,在位置和构型上与RdRp对应的残基不同,剪切了的N端插入到一个结合袋中与许多氢键结合。
前文提到的三个超群中,每个超群都有其标志性的保守氨基酸残基,包括:超群Ⅰ中的Lys/Arg12(基序Ⅰ)、Arg27(基序Ⅱ)、Gly72(基序Ⅲ)、Phe/Tyr339(基序Ⅶ);超群Ⅱ中的Arg32(基序Ⅱ)、Ser/Thr128(基序Ⅳ)、Cys340(基序Ⅶ)、Arg/Lys365(基序Ⅷ);超群Ⅲ中的Ser/Thr190、Thr/Ser200(基序Ⅴ)。值得注意的是,这些标志性的氨基酸残基在各个超群的次要成员中也是保守的。另外,虽然有些病毒的聚合酶的亲缘关系很远,在一些保守基序中却表现出很高的相似性。例如,马铃薯Y属病毒和冠状病毒聚合酶的基序Ⅲ和小RNA病毒聚合酶的基序Ⅶ[1]。
正链RNA病毒聚合酶多肽链的保守区域规则地分布在C-末端附近(除了豇豆花叶病毒组和线虫传多面体病毒),小RNA病毒的聚合酶是所有正链RNA病毒中最小的,整个多肽链中60%都是保守性的区域,说明这些保守性基序可能包含了RNA延伸中的大部分氨基酸残基。
2.2 RdRp的高级结构特征脊髓灰质炎病毒的RdRp的晶体结构是第一个被确定下来的很典型的聚合酶结构,同其他种类的聚合酶相似,呈右手型,具有三个亚结构域:掌型亚结构域(palm subdomain)、拇指型亚结构域(thumb subdomain)和手指型亚结构域(finger subdomain)(图1(ii))[12]。掌型亚结构域中的氨基酸序列呈高度保守性,在PV中,4段核苷酸基序(A-D,图1(iii))相互折叠形成了掌型的中心区域,包括上面的2个α-螺旋和下面的4个反相平行的β-折叠片;C段中高度保守的GDD活性位点存在于所有的RdRp中[1,13];E段位于掌型亚结构域和拇指型亚结构域中间。拇指型亚结构域大部分是由α-螺旋化的C-氨基酸残基组成,还包括N-端的一个β-折叠片[14]。手指型亚结构域包括四个不同的紧密的缠绕在一起的氨基酸序列,分别是:“食指”、“中指”、“无名指”、“小指”(图1(iii))[15]。拇指型亚结构域和手指型亚结构域之间的相互作用形成了一个完全包围着GDD活性位点的构象,而且,在聚合酶的后面形成了一个NTP的进入通道。PV的RdRp的这种拓扑结构和其他的小RNA病毒的非常相似(图1(iv)),包括人鼻病毒(Human rhinovirus,HRV)[16]和口蹄疫病毒(FMDV)[11]的RdRp,尽管他们的一级结构有很低的同源性。此外,RdRp还有一个很重要的特点是有很多聚合酶之间相互作用的区域。
3 RdRp的生物学功能与作用机制

图1 脊髓灰质炎病毒3Dpol结构
RdRp起始RNA合成的机制主要有两种:即引物依赖的RNA合成(primer-dependent RNA synthesis)和非引物依赖的RNA合成即从头合成(De novo RNA sythesis)两种方式[17]。其中,前者根据所依赖引物的不同又可分为3种:蛋白引物,如小RNA病毒科的PV等;前导引物,如冠状病毒科的小鼠肝炎病毒等;模板引物,如嵌杯病毒科的兔出血症病毒等。后者根据起始位点的不同又可分为2种:末端起始,基因组不分节段的正链RNA病毒大多采用这种方式,如噬菌体Qβ采用3'末端从头合成机制起始RNA合成[18];内部起始,基因组分节段的正链RNA病毒大多采用这种方式起始亚基因组RNA的合成,如HCV能通过内部从头合成的方式起始RNA的合成[19]。有些病毒只采用一种方式起始RNA合成,有些则采用两种或两种以上的方式。所以,RdRp的生物学功能与作用机制也有很多种,下面就目前研究的比较透彻的小RNA病毒科的PV来详细地阐述RdRp的生物学功能。
在小RNA病毒中,病毒产生的多聚蛋白需要病毒编码的蛋白酶切割成成熟的蛋白,包括一些前体蛋白,在病毒复制的多个过程中起到了重要作用,PV的3CDPro是病毒蛋白酶3CPro和3Dpol(RdRp)的前体蛋白,具有多种功能[20]。它具有与3CPro不同的蛋白酶切活性,但却没有聚合酶活性[21]。它酶切产生病毒结构蛋白VP0、VP1、VP3和非结构蛋白3AB、3CD、3CPro、3Dpol。RdRp催化尿苷酸(uridine monophosphate,UMP)共价连接到小蛋白VPg的酪氨酸的N-端上,这个尿嘧啶化的VPg之后就作为RNA合成起始的蛋白引物[22,23]。这个过程也被称为VPg的尿苷酰化作用。正链的合成被认为是利用PV基因组上的一个RNA发夹结构作为其引物[24,25]。一种模型(图2)[26]认为在PV基因组上的2C非编码区上的顺式作用复制信号cre(2C)结合3CD与结合了VPg的3Dpol形成一个多组分的复合物[24]。尿嘧啶化的VPg接着转移到病毒RNA的3'端,通过聚合酶开始合成VPg连接的RNA。在负链合成中,Poly(A)可能作为尿嘧啶化的引物,虽然Poly(A)模板没有特异性也不需要3CD来帮助VPg的尿嘧啶化[27]。小RNA病毒复制起始于两个UMP连续地共价连接到引物蛋白VPg的Tyr3的羟基上(VPg-pUpU),由3DPol利用一个内部基因组茎环cre作为模板采用一个回滑机制发挥催化功能(图3)[28]。
以保守的A1A2ACA序列茎环中的A1作为模板,由3Dpol来催化UMP和VPg-Tyr的羟基之间形成磷酸二酯键。然后,VPg-pU回滑至A2并与A2形成氢键,之后第二个UMP就添加到了A1模板上,最后,就形成了VPg-pUpU。

图2 3CD参与的VPg尿嘧啶化模型
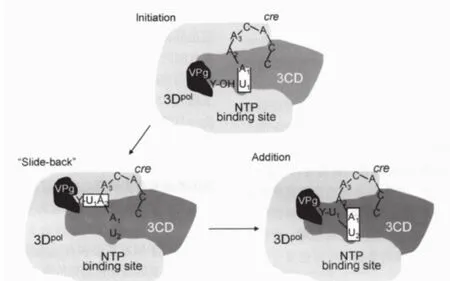
图3 PV 3Dpol催化形成VPg-pUpU回滑机制模型
4 RdRp的研究意义
4.1 有助于研发抗病毒药物正链RNA病毒不仅对人类的健康构成了威胁而且严重危害着经济动植物的生长,为了有效的预防和抵抗这类病毒的侵袭,就必须深入了解病毒的整个生命周期,掌握病毒RNA基因组的复制过程, 明确RdRp在整个复制过程中的作用机制。在此基础上,可以考虑将RdRp作为抗病毒治疗的靶标,研究抗病毒药物,在基因组复制环节,阻断病毒的生命过程。近年来,RNA沉默技术为研发抗RNA病毒的药物提供了一个崭新的途径,因此,可以根据RdRp的基因序列,利用RNA干扰方法,减弱或阻断病毒的复制过程,进而达到抗病毒治疗的目的。例如,Ashfaq等[29]研究认为针对于非结构蛋白NS2-NS5B(RdRp)的siRNAs可以有效地阻止HCV在Huh-7细胞内的复制。NS4Bsi-166和NS5Bsi-1064可以引起HCV RNA基因组明显的减少,并且在20μmol/L浓度时可以抑制病毒滴度达90%以上。 RNA沉默药物将有望成为一种更为高效快捷的抗病毒治疗途径。
4.2 有助于研发疫苗同其他的RNA病毒一样,RdRp的复制缺乏校准机制,其错配率一般在10-3~10-4之间。正是因为这个原因,RNA病毒种群是以准种的形式存在的[30]。由于准种多样性的原因,RNA病毒在面对选择压力时可以很快的适应,例如暴露在宿主的免疫防御机制下的时候[31]。RNA病毒准种的多样性使得RNA病毒的种群的量已经接近“误差灾变论”的阈值,因此,适当增加错配率的话就会导致大部分病毒基因组失去活性以及病毒种群的灭亡。比如核苷酸类似物病毒唑的突变则增加了PV基因组的核酸错配率,就会导致病毒种群的灭亡[32],所以,PV对于病毒唑的抵抗依赖于RdRp中的一个氨基酸位点的突变即:Gly64→Ser(G64S)[33]。有趣的是,这个突变增加了RdRp复制的保真性,所以,在病毒复制的时候错配率就相应的减少了。RNA病毒种群的生存依赖于其复制的错误性和基因组灵活性之间的一种平衡。复制错配率太高会导致种群的灭亡,过低则会因不能适应宿主的环境而灭亡。所以,RdRp复制的保真性则使得临床诊断的意义更加重大[12]。
病毒复制的错误性直接决定了病毒种群躲避免疫中和作用和抵抗抗病毒药物的能力,相应的,RdRp保真性微小的增加都会使病毒的毒力减弱。据此,可以考虑研制RNA病毒的减毒疫苗。RdRp还与病毒的感染相关,最初将其称为病毒感染相关抗原,有研究表明该蛋白可能含有一些特异性的抗原位点,通过对其潜在B细胞表位的预测、分析和研究,进一步研发抗病毒的多表位疫苗也是一条良好的思路[34]。
4.3 有助于研究RNA病毒的分子演化对于正链RNA病毒RdRp结构和功能的研究也许和分子演化的过程也是密切相关的。地球上早期的生命形式很可能是由RNAs占据着主导地位,它不仅是遗传物质而且也是催化单位[35,36]。RNA是很多细胞生命过程中重要的组成部分,在翻译、核酸剪接和DNA复制等过程中都发挥了重要的作用,RNA的这种广泛的存在性是和它在生命进化过程中的原始功能是一致的。DNA和蛋白质是在随后的细胞生命过程中出现的,病毒复制酶很有可能是首批出现的蛋白质中的一员,具有特异性识别DNA的能力。RdRp在RNA合成过程中的一些高度有序的作用步骤和DdRps是相一致的[37,38]。Siegel等[39]研究认为雀麦草花叶病毒(Brome mosaic virus,BMV)的RdRp可以精确地以单股DNA为模板起始RNA的合成,而且,模板竞争实验结果显示,RdRp在与RNA和DNA的相互作用过程中具有相似的亲和力。这些结果表明了RdRp可以很容易地参与到DNA的转录过程中。因此,研究不同种RNA病毒复制酶基因之间的亲缘关系,对于揭示病毒之间的遗传和进化关系具有重要意义,对于一些新发现的RNA病毒,可以根据其复制酶基因的编码序列,判定它在RNA病毒家族中的地位,也可以据此探讨其来源及演化规律。
5 结语
研究RNA病毒的复制过程与机理对于阐述该类病毒的生命周期与规律,甚至研发抗病毒药物或疫苗等都具有重要意义。而RdRp则是催化RNA病毒复制的关键蛋白酶,阐明了RdRp的生物学特性、结构与功能以及发挥催化功能的机制等,也就拿到了研究RNA病毒生命规律的钥匙。近年来,随着生物信息学及多种生命科学研究技术的不断发展,一些重要RNA病毒的复制酶结构和功能陆续得到了比较透彻的研究,其研究成果也被逐渐应用于RNA病毒的基础和应用研究之中。可以相信,随着对RdRp结构与功能的不断深入探索和研究,我们对单股正链RNA病毒的生命规律将会有更清晰的了解和认识、这对于研发新型和有效的抗病毒药物和疫苗等将具有重要的理论和指导价值。
[1] Koonin E V.The phylogeny of RNA-dependent RNA polymerases of positive-strand RNA viruses[J].J Gen Virol, 1991, 72(Ptq): 2197-2206.
[2] Buck K W.Comparison of the replication of positivestranded RNA viruses of plants and animals[J].Adv Virus Res, 1996, 47: 159-251.
[3] Lai M M.Cellular factors in the transcription and replication of viral RNA genomes: A parallel to DNA-dependent RNA transcription[J].J Virol, 1998, 244(1):1-12.
[4] O’Reilly E K, Kao C C.Analysis of RNA-dependent RNA Polymerase structure and function as guided by known polymerase structures and computer predictions of secondary structure[J].J Virol, 1998, 252(2): 287-303.
[5] Ahlquist P.RNA-dependent RNA Polylmerases, viruses and RNA sileneing.Science, 2002, 296(5571): 1270-1273.
[6] Ahlquist P, Noueiry A O, Lee W M,et al.Host factors in positive-strand RNA virus genome replication[J].J Virol,2003, 77(15): 8181-8186.
[7] Harris K S, Xiang W, Alexander L,et al.Interaction of poliovirus polypeptide 3CDprowith the 5’ and 3’termini of the poliovirus genome: Identification of viral and cellular cofactors needed for efficient binding[J].J Biol Chem, 1994, 269: 27004-27014.
[8] Hansen J L, Long A M, Schultz S C.Structure of the RNA-dependent RNA polymerase of poliovirus[J].Structure,1997, 5(8): 1109-1122.
[9] Lesburg C A, Cable M B, Ferrari E,et al.Crystal structure of the RNA-dependent RNA polymerase from hepatitis C virus reveals a fully encircled active site [J].Nat Struct Biol, 1999, 6(10): 937-943.
[10] Ng K K, Cherney M M, Vazquez A L,et al.Crystal structures of active and inactive conformation of a caliciviral RNA-dependent RNA polymerase[J].J Biol Chem, 2002, 277: 1381-1387.
[11] Ferrer-Orta C, Arias A, Perez-Luque,et al.Structure of foot-and-mouth disease virus RNA-dependent RNA polymerase and its complex with a template-primer RNA[J].J Biol Chem, 2004, 279(45): 47212-47221.
[12] Kok C C, McMinn P C.Picornavirus RNA-dependent RNA polymerase[J].Int J Biochem Cell Biol, 2009, 41(3):498-502.
[13] Kamer G, Argos P.Primary structural comparison of RNA-dependent RNA polymerase from plant, animal and bacterial viruses[J].Nucleic Acid Res, 1984, 12(18):7269-7282.
[14] Hansen J L, Long A M, Schultz S C.Structure of the RNA-dependent RNA polymerase of poliovirus[J].Structure, 1997,5(8): 1109-1122.
[15] Thompson A A, Peersen O B.Structural basis for proteolysis-dependent activation of the poliovirus RNA-dependent RNA polymerase[J].EMBO J, 2004, 23: 3462-3471.
[16] Love R A, Maegley K A, Yu X,et al.The crystal structure of the RNA-dependent RNA polymerase from human rhinovirus: a dual function target for common cold antiviral therapy[J].Structure, 2004, 12(8): 1533-1544.
[17] 伊光辉, 张楚瑜.正链RNA病毒起始RNA合成分子机制的研究进展[J].病毒学报, 2005, 21(2):150-154.
[18] Biebricher C K, Eigen M, Luce R.Product analysis of RNA generatedde novoby Qβ replicase[J].J Mol Biol,1981, 148: 369-390.
[19] Pellerin C, Lefebvre S, Little M,et al.Internal initiation sites ofde novoRNA synthesis within the hepatitis C virus polypyrimidine tract[J].Biochem Biophys Res Commun,2002, 295(3): 682-688.
[20] Marcotte L L, Wass A B, Gohara D W,et al.Crystal Structure of Poliovirus 3CD Protein: Virally Encoded Protease and Precursor to the RNA-Dependent RNA Polymerase[J].J Virol, 2007, 81(7): 3583-3596.
[21] Harris K S, Reddigari S R, Nieklin M J,et al.Purification and characterization of Poliovirus polypeptide 3CD, a Proteinase and a Precursor for RNA Polymerase[J].J Virol,1992, 66(12): 7481-7489.
[22] Ferrer-Orta C, Agudo R, Domingo E,et al.Structural insights into replication initiation and elongation processes by the FMDV RNA-dependent RNA polymerase[J].Curr Opin Struct Biol, 2009, 19(6): 752-758.
[23] Gu C, Zeng T, Li Y,et al.Structure-function analysis of mutant RNA-dependent RNA polymerase complexes with VPg[J].Biochemistry(Mosc), 2009, 74(10): 1132-1141.
[24] Paul A V, Rieder E, Kim D W,et al.Identification of an RNA hairpin in Poliovirus RNA that serves as the Primary template in the in vitro uridylylation of VPg[J].J Virol,2000, 74(22): 10359-10370.
[25] Rieder E, Paul A V, Kim D W,et al.Genetic and biochemical studies of Poliovirus cis-acting replication element cre in relation to VPg uridylylation[J].J Virol,2000, 74(22): 10371-10380.
[26] Pathak H B, Oh H S, Goodfellow I G,et al.Picornavirus genome replication: roles of precursor proteins and ratelimiting steps in oril-dependent VPg uridylylation[J].J Biol Chem, 2008, 283(45): 30677-30688.
[27] Paul A V, van Boom J H, Filippov D,et al.Proteinprimer RNA synthesis by purified poliovirus RNA polymerase[J].Nature, 1998, 393: 280-284.
[28] Paul A V.Possible unifying mechanism of picornavirus genome replication[J].Mol biol picornavir-uses, 2002,227-246.
[29] Ashfaq U A, Ansar M, Sarwar M T,et al.Posttranscriptional inhibition of hepatitis C virus replication through small interference RNA[J].J Virol, 2011, 8: 112.
[30] Domingo E, Holland J J.RNA virus mutations and fitness for survival[J].Annu Rev Microbiol, 1997, 51: 151-178.
[31] Vignuzzi M, Stone J K, Arnold J J,et al.Quasispecies diversity determines pathogenesis through cooperative interactions in a viral population[J].Nature, 2006,439(7074): 344-348.
[32] Pfeiffer J K, Kirkegaard K.Increased fidelity reduces poliovirus fitness and virulence under selective pressure in mice[J].PLoS Pathog, 2005, 1(2): ell.
[33] Diamond S E, Kirkegaard K.Clustered charged-toalanine mutagenesis of poliovirus RNA-dependent RNA polymerase yields multiple temperature-sensitive mutants defective in RNA synthesis[J].J Virol, 1994, 68(2): 863-876.
[34] 张昱, 王永录, 张永光, 等.口蹄疫病毒3D聚合酶B细胞表位的筛选鉴定[J].华北农学报, 2009, 24(3): 69-73.
[35] Gilbert W.Origin of life: The RNA world[J].Nature,1986, 319: 618.
[36] Joyce G F.RNA and the origins of life[J].Nature, 1989,338: 217-224.
[37] Adkins S, Siegel R W, Sun J.Minimal templates directing accurate initiation of subgenomic RNA synthesis in vitro by the brome mosaic virus RNA-dependent RNA polymerase[J].RNA, 1997, 3(6): 634-647.
[38] Sun J, Kao C C.RNA synthesis by the brome mosaic virus RNA-dependent RNA polymerase: Transition from initiation to elongation[J].J Virol, 1997, 233(1): 63-73.
[39] Siegel R, Bellon L, Beigelman.Identification of the base moieties in an RNA promoter specifically recognized by a viral RNA-dependent RNA polymerase[J].Proc Natl Acad Sci, 1998, 95(20): 11613-11618.

