瘦素基因nt+19A→G多态性与阻塞性睡眠呼吸暂停低通气综合征的关系
高春艳,刘运秋,刘晓宇,李宏芬
(河北联合大学附属开滦医院,河北唐山063000)
临床及流行病学资料证实,肥胖是导致阻塞性睡眠呼吸暂停低通气综合征(OSAHS)的重要危险因子,遗传因素在肥胖发生中起重要作用;OSAHS存在明显的家族聚集现象及种族差异[1],提示遗传因素参与OSAHS发病。人类瘦素基因位于染色体7q31.3,近年研究发现该基因区域与人类极度肥胖相关[2]。目前,有关瘦素基因与OSAHS的相关研究较少,我们对瘦素基因外显子5’非翻译区第19位核苷酸nt+19A→G的变异进行了研究,旨在探讨其基因多态性与中国北方成人OSAHS易感性之间的关系。
1 资料与方法
1.1 临床资料 选择2010年1月~2011年6月在我院住院的OSAHS患者180例(OSAHS组),男141例、女39例,年龄(46.7 ±10.3)岁,BMI(26.18 ±3.22)kg/m2;均符合《阻塞性睡眠呼吸暂停低通气综合征诊断及治疗专家共识(2007年草案)》中的诊断标准。另选同期查体健康者180例作为对照组,其性别、年龄、BMI与 OSAHS组有可比性。根据BMI分别将两组又分为体质量正常组(BMI<25 kg/m2)及肥胖组(BMI≥25 kg/m2)。排除年龄<18周岁,严重颌面畸形,患影响血氧饱和度的疾病,正在服用影响睡眠的药物,己接受针对OSAHS的治疗,多导睡眠仪监测睡眠时间少于6 h,合并高血压、心脑肾肝疾病、糖尿病及精神疾病者。两组均为我国北方汉族居民,无血缘关系。
1.2 方法 两组均于夜间在SRM-9601多导睡眠监测(PSG)系统下进行至少6 h的睡眠监测,并进行颈围、腰围、腹围、上气道检查等。
1.2.1 基因型检测 抽取晨起空腹肘静脉血5 ml,采用EDTA抗凝,低渗法分离白细胞,氯仿/异戊醇法提取DNA,PCR-RFLP法检测nt+19A→G基因型。DNA扩增总体系25 μl,上游引物:5'-CCCGCGAGGTGC ACACTG-3',下 游 引 物:5'-A GGAGGAAGGAGCGCGCC-3',引物链由上海生工生物公司提供。反应条件:94℃预变性5 min,94℃变性30 s,65℃退火30 s,进行30个循环,扩增产物221 bp。经2.5%琼脂糖电泳确认扩增结果后,在20 μl酶切反应体系中,加入 MspA1I内切酶0.5 μl(NEB 公司),混匀后置37℃水浴中过夜,酶切产物置入2.5%琼脂糖凝胶(EB染色)电泳,与Marker对照,在紫外灯下观察结果。AA纯合子无酶切位点,为221 bp;GG纯合子为183、38 bp;GA 杂合子为 183、221、38 bp,因 38 bp 太小,在电泳过程中丢失。
1.2.1 统计学方法 采用SPSS13.0统计软件,计量资料用±s表示,组间比较用独立样本t检验或方差分析,计数资料比较用χ2检验;遗传平衡检验采用Hardy-Weinberg平衡定律。P≤0.05为差异有统计学意义。
2 结果
2.1 两组基线资料比较 见表1。
表1 两组基线资料比较(±s)

表1 两组基线资料比较(±s)
基线资料 OSAHS组(n=180) 对照组(n=180)P颈围(cm)41.57 ±2.36 39.42 ±2.53 <0.01腹围(cm) 93.01 ±9.58 89.34 ±8.99 <0.01腰臀比 0.930 ±0.051 0.902 ±0.046 <0.01体脂含量(%)26.00 ±4.03 25.12 ±3.56 <0.05
2.2 两组nt+19A→G基因型与基因频率比较见表 2、3。

表2 两组nt+19A→G位点不同基因型频率的比较[例(%)]

表3 两组nt+19A→G等位基因频率的比较[例(%)]
2.3 不同基因型OSAHS患者的临床指标比较OSAHS患者中,nt+19A→G各基因型个体比较,BMI、颈围、腹围、腰臀比均无统计学差异(P均>0.05)
2.4 不同基因型OSAHS患者的各睡眠指标比较见表4。
3 讨论
OSAHS是具有潜在风险的临床综合征,许多研究显示,肥胖是其重要的危险因子,遗传参与肥胖及OSAHS的发病。Palmer[3]通过对 OSAHS及肥胖患者的基因组扫描认为,OSAHS及肥胖的易患性可能通过一些共同的基因起作用,而且参与影响AHI、BMI的染色体定位十分相近,提示肥胖与OSAHS可能通过共同的基因调控导致内分泌异常。瘦素基因是近年发现的与肥胖相关的最主要基因。Hager等[4]等研究认为,nt+19A→G基因变异与白种人肥胖者的血清瘦素水平相关。Rossana等[5]研究发现,在意大利严重肥胖患者的nt+19A→G基因中,A、G等位基因的分布频率在肥胖组与对照组间无统计学差异,其基因多态性与瘦素水平无关,与胆固醇、甘油三酯、BMI、脂肪含量等均无相关性,唯一有差异的是GG基因型肥胖女性的腰臀比明显小于AA或GA基因型者;该学者认为,虽然Hager发现G等位基因携带者的瘦素较低,但其未考虑腰臀比的影响,故不能排除其结果因脂肪分布不同所致。张蓉等[6]认为,瘦素基因nt+19A→G变异可影响中国人2型糖尿病的局部体脂(主要是腹部皮下脂肪)分布。
表4 不同基因型OSAHS患者的各睡眠指标比较(±s)
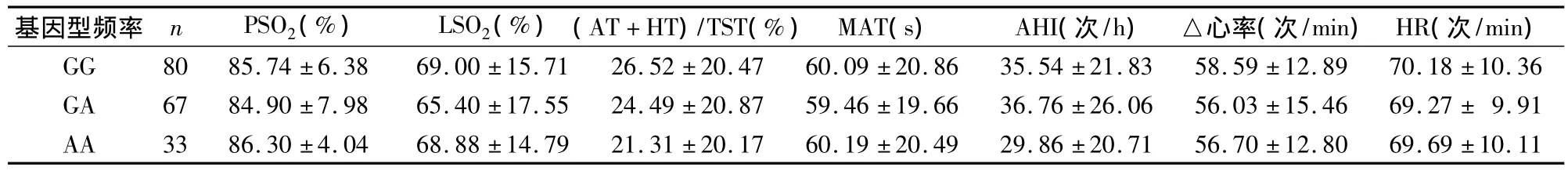
表4 不同基因型OSAHS患者的各睡眠指标比较(±s)
注:PSO2:夜间平均氧饱和度;LSO2:最低氧饱和度;AHI:呼吸暂停低通气指数;(AT+HT)/TST:(总呼吸暂停时间+总低通气时间)/总睡眠时间;MAT:最长呼吸暂停时间;△心率:夜间最高心率-夜间最低心率;HR:夜间平均心率。不同基因型OSAHS患者的各睡眠指标比较,P均 >0.05
基因型频率 n PSO2(%) LSO2(%)(AT+HT)/TST(%)MAT(s) AHI(次/h) △心率(次/min)HR(次/min)GG 80 85.74 ±6.38 69.00 ±15.71 26.52 ±20.47 60.09 ±20.86 35.54 ±21.83 58.59 ±12.89 70.18 ±10.36 GA 67 84.90 ±7.98 65.40 ±17.55 24.49 ±20.87 59.46 ±19.66 36.76 ±26.06 56.03 ±15.46 69.27 ± 9.91 AA 33 86.30 ±4.04 68.88 ±14.79 21.31 ±20.17 60.19 ±20.49 29.86 ±20.71 56.70 ±12.80 69.69 ±10.11
瘦素基因nt+19A→G变异存在三种基因型,本研究显示,OSAHS组 GG型占45.0%,GA型占36.7%,AA型占18.3%,其 G、A 的等位基因频率分别为63.3%、36.7%;对照组的 GG、GA、AA 基因型频率分别为37.8%、46.7%、15.5%,其 G、A 等位基因频率分别为61.1%、38.9%。两组基因型分布及等位基因频率分布均无统计学差异。OSAHS组中的GG、GA、AA基因型频率个体比较,其颈围、腹围、腰臀比、BMI等均无统计学差异,提示其变异对局部体脂分布无明显影响,结果与Rossana等[5]报道一致,但与张蓉等[6]结果不同,可能与相关疾病的选择、样本量大小等有关。三种基因型个体PSG各项指标比较均无明显统计学差异,考虑其单一基因变异在OSAHS发病中可能无重要作用,其原因可能为肥胖及OSAHS发生有多基因参与,且受生活方式、饮食习惯等因素影响;其发病有遗传异质性,不同个体发病有不同的基因参与,同一个体可能有多致病基因,单一基因可能仅以不同的风险率参与疾病的发生。由于瘦素基因结构复杂,存在多种变异,故对其与肥胖、OSAHS的关系有待于深入研究。
综上所述,本研究认为瘦素基因nt+19A→G单一基因变异在我国北方汉族居民OSAHS发生中可能无明显作用。
[1]Taheri S,Miqnot E.The genetics of sleep disorders[J].Lancet Neurol,2002,1(4):242-250.
[2]Clement K,Garner C,Hager J,et al.Indication for linkage of the human OB gene region with extreme obesity[J].Diabetes,1996,45(5):687-690.
[3]Palmer LJ.Genomic approaches to understanding obstructive sleep apnea[J].RespirPhysiol Neurobiol,2003,135(2-3):187-205.
[4]Hager J,Clement K,Francke S,et al.A polymorphism in the 5’untranslated region of the human ob gene is associated with low leptin levels[J].Int J Obes,1998,22(3):200-205.
[5]Rossana L,Emanuela P,Maria EB,et al.The A19G polymorphism in the 5'untranslated region of the human obese gene does not affect leptin levels in severely obese patients[J].J Clin Endocrinol Metab,2000,85(10):3589-3591.
[6]张蓉,郑以漫,项坤三,等.瘦素基因nt+19A→G变异与中国人2型糖尿病脂代谢、体脂含量及分布的关系[J].中华内分泌代谢杂志,2000,16(1):6-9.

