真空包装冷却猪肉冷藏过程中菌相变化
江 芸,高 峰,徐幸莲,叶可萍,周光宏,*
真空包装冷却猪肉冷藏过程中菌相变化
江 芸1,2,高 峰1,徐幸莲1,叶可萍1,周光宏1,*
(1. 南京农业大学 国家肉品质量安全控制工程技术研究中心,江苏 南京 210095;2. 南京师范大学金陵女子学院,江苏 南京 210097)
应用传统微生物培养和PCR-DGGE方法研究真空包装冷却猪肉4℃贮藏过程中的菌相变化。细菌培养计数结果表明,乳酸菌生长迅速,在贮藏后期即超过了细菌总数值。DGGE结合16S rRNA基因序列分析结果表明,贮藏初期肉中初始菌相较复杂,贮藏末期主要是漫游球菌、肉食杆菌、乳杆菌、乳球菌和热死环丝菌成为优势腐败菌。
冷却猪肉;真空包装;菌相组成;聚合酶链式反应-变性梯度凝胶电脉法(PCR-DGGE)
冷却肉因微生物污染、繁殖极易导致腐败变质,进而引发肉品安全问题。研究表明,真空包装结合低温贮藏可有效延长肉品货架期[1]。冷却肉真空包装条件下贮藏过程中菌相的变化,基于传统微生物培养计数的研究已有较多报道,许多研究均表明,真空包装肉贮藏末期主要的腐败菌是乳酸菌,如乳杆菌(Lactobacillus)、明串珠菌(Leuconostoc)、肉食杆菌(Carnobacterium)以及热死环丝菌(Brochothrix thermosphacta)[1-3]。近年来,随着分子生物学的发展,基于DNA指纹技术的分子生物学研究手段越来越多地被引入到微生物学包括食品微生物学研究中,例如变性梯度凝胶电泳法(denaturing gradient gel eletrophoresis,DGGE)、限制性片段长度多态性分析(restriction fragment length polymorphism,RFLP)、末端限制性片段长度多态性分析(terminal restriction fragment length polymorphism,TRFLP)、随机扩增多态性DNA(random amplified polymorphic DNA,RAPD)、单链构象多态性分析(single strand conformational polymorphism,SSCP)等。其中DGGE具有可靠性强、重现性高、方便快捷等优点而越来越受到重视,是近年来微生物学研究中应用较广泛的分子技术之一。
DGGE技术是由Fischer和Lerman于1979年最先提出的用于检测DNA突变的一种电泳技术[4]。Muzyer等[5]首次将DGGE技术应用于微生物生态学研究,并证实了这种技术在研究自然界微生物群落的遗传多样性和种群差异方面具有明显的优越性。与传统微生物学方法相比,DGGE可不采用培养方法,而从食物样品中直接提取总DNA,这样能检测到难以培养或不能培养的微生物,能够同时检测多种微生物,而且检测速度快。目前,DGGE技术在食品微生物研究方面有许多报道,如奶酪、香肠、酸面团、泡菜、葡萄酒等食品均有报道。近年来,应用DGGE技术研究不同包装牛肉贮藏过程中的菌相变化亦有报道[6-8],Li等[9]应用DGGE技术研究了真空包装猪肉,但贮藏期仅6d。
本实验将冷却猪肉真空包装低温贮藏,在不同贮藏时期不经培养直接提取肉中细菌DNA,应用PCR-DGGE技术,研究菌相变化,揭示贮藏末期主要优势腐败菌,为进一步研究其保鲜措施提供科学依据。
1 材料与方法
1.1 材料及处理
由南京市某一冷却猪肉专卖店购买两条猪后腰肉(A组和B组),由专卖店工作人员按正常操作程序分别切分成5块,用洁净包装袋按组分别包装,置于冰盒中于30min内送置实验室,立即将每块肉样各放入一真空包装袋中,包装膜的氧气通透率(O2transmission rate, OTR)为30cm3/(m2·24h·atm),进行真空包装(真空度0.1MPa,抽空时间30s,热封温度90℃,热封时间5s)。分别标上A组和B组。4℃冰箱放置,分别于第0、3、10、17天和第24天每组各取1个包装,取肉样进行分析。
1.2 试剂与仪器
AxyPrep细菌基因组DNA小量试剂盒 美国Axygen公司;Wizard SV Gel and PCR Clean-up System纯化试剂盒、GoTaq Green Master Mix (2×) 美国Promega公司;丙烯酰胺、甲叉双丙烯酰胺、尿素、Tris、EDTA、TEMED、甘油、过硫酸胺、琼脂糖 美国Sunshine公司;甲酰胺(分析纯);DNA Marker DL2000大连TaKaRa公司;Goldview染料 北京赛百盛公司;溴化乙锭(ethidium bromide,EB)应用液 美国Sigma公司;平板计数琼脂(plate count agar,PCA)培养基、MRSA(de man rogosa sharpe agar)培养基 北京陆桥有限公司;所用引物均由上海Invitrogen公司合成。
BioRad DCode apparatus DGGE电泳仪、GelDoc 2000 system凝胶成像仪 美国BioRad公司;Mastercycler ep personalPCR仪 德国Enperndorf公司;水平电泳仪 北京百晶生物技术有限公司;DP12 Olympus显微镜 日本Olympus公司;DHZ-C恒温振荡器 江苏太仓市实验设备厂;64R台式高速冷冻离心机 美国Beckman公司;Anke TLG-16G台式离心机 上海安亭科学仪器厂;LX-100手掌型离心机 江苏海门市其林贝尔公司;DZQ500/ 2SB真空充气包装机 浙江葆春包装机械总厂;SW-CFIF超净工作台 苏州安泰充气技术有限公司。
1.3 方法
1.3.1 微生物选择性培养计数
无菌操作称取冷却猪肉25g,无菌剪刀剪碎,放入225mL灭菌生理盐水中,充分振荡15min,取1mL上清液进行10倍梯度稀释,选择合适的稀释度,细菌总数采用PCA培养基37℃培养48h,乳酸菌采用MRSA培养基30℃培养48h[10]。培养结束后计数,结果以lg(CFU/g)表示。
1.3.2 直接提取冷却肉细菌DNA
无菌剪取一定肉样,用灭菌剪刀剪碎,精确称取10g碎肉样,放入灭菌锥形瓶(内有10g玻璃珠、100mL灭菌生理盐水),摇床280r/min、4℃条件振摇15min[9,11],静置5min,取30mL上清液于一灭菌离心管中,200×g离心5min后,取15mL上清液于另一灭菌离心管中,12000×g离心5min。离心所得沉淀,采用AxyPrep细菌基因组DNA提取试剂盒,方法稍作调整。沉淀加入150μL Buffer S悬浮,加入20μL溶菌酶(50mg/mL),混匀,37℃孵育1h,加入30μL蛋白酶K(20mg/mL),混匀,55℃孵育2~3h,然后继续按试剂盒操作说明进行。提取的总DNA溶于80μLTE缓冲液,1.2%琼脂糖凝胶电泳检测后,-20℃冻存。
1.3.3 巢式聚合酶链式反应(nested PCR)
采用巢式PCR扩增冷却肉细菌16S rRNA基因V3可变区。第一轮PCR采用引物8f (5'-GGA GAG TTT GAT CA/CT GGC T-3')和798r (5'-CCA GGG TAT CTA ATC CTG TT-3')扩增约800bp的16S rRNA基因序列。PCR反应体系(25μL):GoTaq Green Master Mix(2×)12.5μL,引物各1μL(0.4μmol/L),DNA模板1μL,ddH2O 9.5μL。PCR扩增程序:94℃预变性2min;94℃变性1min,58℃退火1min,72℃延伸2min,25个循环;最后72℃再延伸2min[12]。PCR产物经1.2%琼脂糖凝胶电泳检测后,采用纯化试剂盒进行割胶纯化,纯化程序依据试剂盒使用说明进行。纯化产物再次经琼脂糖凝胶电泳检测后,-20℃冻存。
以上述纯化产物为模板进行第二轮PCR反应,采用引物GC338f (5'-CGC CCG CCG CGC GCG GCG GGC GGG GCG GGG GCA CGG GGG GAC TCC TAC GGG AGG CAG CAG-3')和518r (5'-ATT ACC GCG GCT GCT GG-3'),对细菌16S rRNA基因V3区片段进行“降落”PCR (touchdown PCR)扩增。PCR反应体系(50μL):GoTaq Green Master Mix (2×) 25μL,引物各1μL(0.2μmol/L),DNA模板1μL,ddH2O 22μL。PCR扩增程序:94℃预变性5min,然后采用touchdown PCR程序,即20个循环(94℃变性1min,退火温度从65℃到55℃,退火30s,72℃延伸3min),再于恒定的退火温度下进行10个循环(94℃变性1min,55℃退火30s,72℃延伸3min),最后72℃再延伸10min[5,13]。PCR产物经1.2%琼脂糖凝胶电泳检测后,-20℃冻存。
1.3.4 变性梯度凝胶电泳(DGGE)
参照Muyzer等[5]方法,对细菌16S rDNA的V3区扩增产物进行DGGE分析。采用DGGE电泳仪,聚丙烯酰胺凝胶浓度为8%(丙烯酰胺与甲叉双丙烯酰胺的质量比为37.5:1),变性梯度为35%~55%(100%变性剂含有7mol/L尿素和40%甲酰胺),在0.5×TAE缓冲液中,60℃恒温条件下,先200V电压预电泳10min,随后再85V电压电泳16h。
电泳结束后,将DGGE胶片用1×TAE(含0.5mg/L EB)染色15min。弃去染色液,再用ddH2O漂洗20min。染色后用凝胶成像系统照相。
1.3.5 DNA回收、纯化和测序
将EB染色的V3区的DGGE胶片放置于紫外灯下,切下不同位置的条带,分别放入1.5mL灭菌离心管中,加入20μL无菌水4℃冰箱过夜。取2μL为模板DNA以GC 338f/518r为引物进行16S rDNA V3区扩增,扩增方法同1.3.3节,PCR产物经DGGE后证明与所割条带位于相同的迁移位置,再割胶回收,用不含GC夹子的引物再次扩增16S rDNA V3区序列,纯化后送至上海Invitrogen生命技术有限公司测序。登录NCBI(www. ncbi.nlm.nih,gov/blast/),将所得序列与数据库中的已知序列进行相似性比对[14]。
2 结果与分析
2.1 微生物培养结果
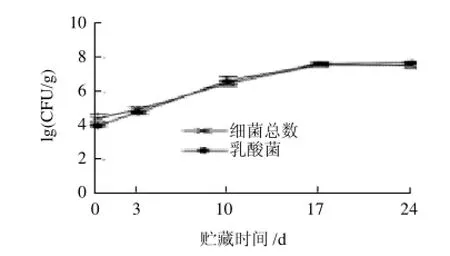
图1 真空包装冷却猪肉贮藏过程中细菌总数和乳酸菌培养计数结果Fig.1 Counts of total bacteria and lactic acid bacteria in vacuumpackaged chilled pork during storage
由图1可知,贮藏初期细菌总数对数值的平均值超过了4,乳酸菌低于细菌总数约0.5 lg(CFU/g)。随着贮藏时间的延长,细菌总数和乳酸菌均逐渐增多,贮藏末期均达到了7 lgCFU/g以上。贮藏过程中,乳酸菌增长迅速,至第3天时已接近细菌总数值,至第10天时则超过了细菌总数值。
2.2 PCR-DGGE结果
采用巢式PCR,第一轮PCR采用引物8f/798r扩增出约800bp左右的预期条带(图2),PCR产物采用试剂盒进行纯化,纯化产物采用引物GC338f/518r对细菌16S rDNA的V3区片段进行第二轮PCR扩增,得到约230bp的扩增产物。在变性梯度35%~55%进行DGGE,结果见图3。
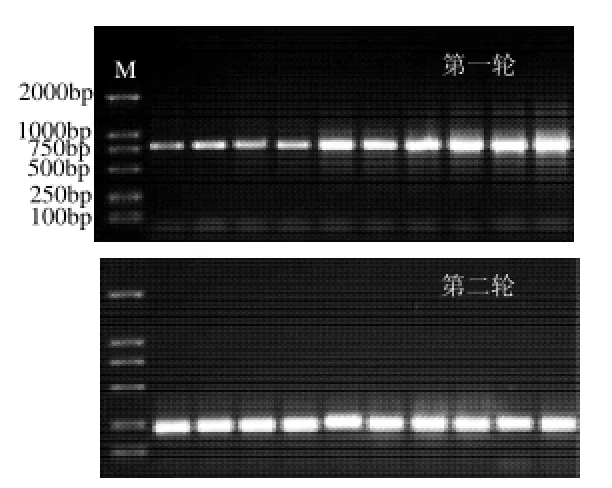
图2 巢式PCR电泳结果Fig.2 Electrophoresis profile of nested PCR products

图3 真空包装冷却猪肉直接提取细菌DNA的16S rRNA基因V3区DGGE图谱Fig.3 DGGE profile of PCR products for the V3 region of 16S rRNA gene extracted from vacuum-packaged chilled pork
由图3可知,贮藏初期产生了较多条带,表明冷却肉中初期污染有多种细菌;随着贮藏时间的延长,条带分布发生明显变化,一些条带逐渐变弱或消失,一些条带由弱变强,还出现了一些新条带;至贮藏末期,亮条带主要位于图谱上半部,表明贮藏过程中菌相组成发生了显著变化。
2.3 割胶条带测序及结果分析
对图3 DGGE图谱被分离的条带1~15,经过割胶、扩增、测序,登录NCBI用BLAST在GenBank数据库中与参考序列进行相似性分析,结果如表1所示。各条带相似性在98%以上。由图3和表1可知,第0天时,肉中存在的初始污染菌有多种,有Serratia/Pseudomonas (沙雷氏菌属/假单胞菌属,条带1和2)、Achromobacter (无色杆菌属,条带3)、Psychrobacter(条带4)、Acinetobacter(不动杆菌属,条带5和6)、Lactococcus (乳球菌属,条带7),表明贮藏初期肉中菌相较复杂。有些条带未被测定,有些条带测定失败,图中未标出。
随着贮藏时间的延长,上述许多菌所对应的条带逐渐变弱甚至消失,Proteobacterium对应的条带10出现,Enterobacter/Hafnia(肠细菌属/哈夫尼菌属,条带8)和Brochothrix thermosphacta(热死环丝菌,条带9)所对应的条带逐渐增强,随后它们又逐渐变弱,而乳酸菌所对应的很多条带增强。至贮藏末期,除了热死环丝菌,还有Vagococcus(漫游球菌属,条带12)、Carnobacterium(肉食杆菌属,条带13)、Lactobacillus (乳杆菌属,条带14)及Lactococcus(乳球菌属,条带15)成为最主要的细菌。

表1 DGGE条带分离的细菌16S rRNA基因序列鉴定Table 1 DGGE bands identified by 16S rRNA gene sequencing
3 讨 论
应用传统微生物培养方法研究肉品真空包装条件下贮藏过程中微生物的组成及变化已有许多报道[1-3]。本实验直接提取肉样细菌DNA,应用PCR-DGGE方法,研究真空包装冷却猪肉冷藏过程中的菌相变化,结果表明,贮藏初期主要有沙雷氏菌属/假单胞菌属、无色杆菌属、不动杆菌属、乳酸菌等。本实验应用DGGE方法鉴定的冷却肉中的初始菌相与大多数传统微生物培养方法的研究报道一致[15-16],但也有不同的报道,这可能是冷却肉不同生产环境、不同加工工艺对猪胴体表面菌相会有影响[15],另外本实验并未对所有条带进行鉴定。
真空包装采用阻隔性能较好的包装材料,除去包装袋内的空气,导致好气性微生物的生长受到抑制,乳酸菌不断增殖成为主要优势菌,货架期延长[1-2]。本实验传统培养方法的结果亦表明,真空包装条件下乳酸菌生长迅速,贮藏后期即超过了细菌总数值。但由于本次实验肉样的初始菌数较高(>4lg(CFU/g)),第10天时细菌总数平均值已超过6lg(CFU/g)。我国对冷却肉的卫生指标尚无菌数指标,一般认为冷却肉的细菌总数为6lg(CFU/g)时为警戒线,达到7lg(CFU/g)时冷却肉外观有明显的腐败现象,达到8lg(CFU/g)时外表有黏液形成,不宜食用,部分国家也把6lg(CFU/g)作为冷却分割肉的细菌总数指标[17]。以此标准判断,本实验真空包装冷却猪肉4℃贮藏时货架期不足10d。为了达到更好的贮藏效果,控制初始污染菌量非常必要。Egan[18]研究发现,真空包装牛肉0℃下可贮藏10~12周,优势乳酸菌主要是肉食杆菌属、乳杆菌属和明串珠菌属。Nápravníková等[19]研究发现,真空包装猪肉(2.5±0.5)℃条件贮藏,依据肉初始pH值的不同,货架期在21~28d之间,乳酸菌增长迅速,应用API系统鉴定发现腐败菌中主要为弯曲乳杆菌(Lactobacillus curvatus)和木糖乳杆菌(Lactobacillus xylosus)。Li等[9]同样应用DGGE方法,对细菌16S rRNA基因V6~V8可变区的研究表明,真空包装猪肉贮藏末期主要腐败菌是乳杆菌和热死环丝菌。本实验应用DGGE结合16S rRNA基因序列分析,发现贮藏末期主要是多种乳酸菌(漫游球菌、肉食杆菌、乳杆菌、乳球菌)和热死环丝菌,这可能也是由于冷却肉生产加工环境的差异导致猪胴体表面初始菌相不完全相同[15],进而影响到贮藏末期的菌相组成。
为了增加PCR反应的灵敏性,巢式PCR方法已被成功应用于低丰度微生物的分子生物学研究中[20-21]。本实验中,直接提取冷却猪肉细菌DNA,采用引物GC338f/518r对细菌16S rRNA基因V3区片段进行一次PCR扩增,结果贮藏前期所得的PCR产物条带较弱(结果未显示),无法满足进一步的DGGE分析所需,故为了增加灵敏性,本实验亦采用了nested PCR方法,结果得到足够的PCR产物,可用于进一步的DGGE分析。
[1]GILL C D, MOLIN G. Modified atmospheres and vaccum packaging [M]//RUSELL N J, GOULD G W. Food preservatives. Glasgow: Blackie and Son Ltd., 1991: 172-199.
[2]CHURCH I, PARSONS A. Modified atmosphere packaging technology: a review[J]. Journal of the Science of Food and Agriculture, 1995, 67(2): 143-152.
[3]BORCH E, KANT-MUERMANS M L, BLIXT Y. Bacterial spoilage of meat and cured meat product[J]. International Journal of Food Microbiology, 1996, 33(1): 103-120.
[4]FISHCER S G, LERMAN L S. Length-independent separation of DNA restriction fragments in two dimensional gel electrophoresis[J]. Cell, 1979, 16(1): 191-200.
[5]MUYZER G, WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA [J]. Applied Environmental Microbiology, 1993, 59(3): 695-700.
[6]ERCOLINI D, RUSSO F, TORRIERI E, et al. Changes in the spoilagerelated microbiota of beef during refrigerated storage under different pack-aging conditions[J]. Applied and Environmental Microbiology, 2006, 72(7): 4663-467.
[7]FONTANA C, COCCONCELLI P S, VIGNOLO G. Direct Molecular approach to monitoring bacterial colonization on vacuum-packaged beef [J]. Applied and Environmental Microbiology, 2006, 72(8): 5618-5622.
[8]RUSSO F, ERCOLINI D, MAURIELLO G, et al. Behaviour of Brochothrix thermosphacta in presence of other meat spoilage microbial groups[J]. Food Microbiology, 2006, 23(8): 797-802.
[9]LI Miaoyun, ZHOU Guanghong, XU Xinglian, et al. Changes of bacterial diversity and main flora in chilled pork during storage using PCRDGGE[J]. Food Microbiology, 2006, 23(7): 607-611.
[10]GB4789.35—2010 食品微生物学检验乳酸菌检验[S].
[11]OLSSON C, AHRNE S, PETTERSSON B, et al. The bacterial flora of fresh and chill-stored pork: analysis by cloning and sequencing of 16S rRNA genes[J]. International Journal of Food Microbiology, 2003, 83 (3): 245-252.
[12]LI Zhiyong, HE Liming, WU Jie, et al. Bacterial community diversity associated with four marine sponges from the South China Sea based on 16S rDNA-DGGE fingerprinting[J]. Journal Experimental Marine Biological Ecology, 2006, 329(1): 75-85.
[13]AMPE F, MIAMBI E. Cluster analysis, richness and biodiversity indexes derived from denaturing gradient gel electrophoresis fingerprints of bacterial communities demonstrate that traditional maize fermentations are driven by the transformation process[J]. International Journal of Food Microbiology, 2000, 60(1): 91-97.
[14]ALTSCHUL S F, MADDEN T L, SCHAFFER A A, et al. Gapped BLAST and PSIBLAST: a new generation of protein database search programs[J]. Nucleic Acids Research 1997, 25(17): 3389-3402.
[15]JAY J M. Modern Food microbiology[M]. Fresh Meat and Poultry, Gaithersburg. 6th MD: Aspen Publishers, 2000: 59-85.
[16]DAINTY R H, SHAW B G, ROBERTS T A. Microbial and chemical changes in chill-stored red meat[M]//ROBERTS T A, SKINNER F A. Food Microbiology. London: Advances and Prospects Academic Press, 1983: 151-178.
[17]吕志平. 国内外技术法规和标准中食品微生物限量[M]. 北京: 中国标准出版社, 2002: 1-179.
[18]EGAN A F. Lactic acid bacteria of meat and meat products[J]. Antonie van Lccuwcnhock, 1983, 49(3): 327-336.
[19]NAPRAVNIKOVA E, VORLOVA L, MALOTA L. Changes in hygienic quality ot vacuum-packed pork during storage[J]. ACTA Veterinaria BRNO, 2002, 71(1/3): 255-262.
[20]GIBBONS C L, AWAD-EL-KARIEM F M. Nested PCR for the detection of Cryptosporidium parvum[J]. Parasitology Today, 1999, 15: 345.
[21]VERDIN E, SAILLARD C, LABBLE A, et al. A nested PCR assay for the detection of Mycoplasma hyopneumoniae in tracheobronchiolar washings from pigs[J]. Veterinary Microbiology, 2000, 76(1): 31-40.
Microfloral Change of Vacuum-packaged Pork during Chilled Storage
JIANG Yun1,2,GAO Feng1,XU Xing-lian1,YE Ke-ping1,ZHOU Guang-hong1,*
(1. National Center of Meat Quality and Safety Control, Nanjing Agricultural University, Nanjing 210095, China;2. Ginling College, Nanjing Normal University, Nanjing 210097, China)
The microfloral change in vacuum-packaged pork during 4 ℃ storage was explored by traditional bacterial cultivation and PCR-denaturing gradient gel eletrophoresis (PCR-DGGE). Results indicated that lactic acid bacteria (LAB) exhibited a rapid increase and excessive total bacterial counts in the late stage during chilled storage. DGGE combined with the analysis of 16S rRNA gene sequence revealed a complex microflora in chilled pork at the early stage during chilled storage; in contrast, Vagococcus, Carnobacterium, Lactobacillus and Lactococcus, and Brochothrix thermosphacta were the predominant spoilage bacteria at the end of storage period.
chilled pork;vacuum package;microfloral composition;polymerase chain reaction-denaturing gradient gel eletrophoresis (PCR-DGGE)
TS251.51
A
1002-6630(2011)04-0241-05
2010-04-06
国家“863”计划项目(2008AA100804);江苏省高校自然科学基础研究项目(08KJB550005);
上海市科学技术委员会重大攻关项目(07dz19508)
江芸(1971—),女,副教授,博士研究生,研究方向为肉品安全与质量控制。E-mail:jiangyun@njnu.edu.cn
*通信作者:周光宏(1960—),男,教授,博士,研究方向为肉品科学。E-mail:ghzhou@njau.edu.cn

