苯酚降解菌的筛选与鉴定
王太平
(河南省驻马店农业学校,河南驻马店463000)
随着石油化工、塑料、合成纤维和焦化等工业的迅速发展,各种含酚废水也相应增多,这是目前水体的主要污染源之一.苯酚属高毒性、难于降解的有机物[1],排放到环境中不仅毒害水生生物、破坏水生生态系统,而且对人类健康构成很大威胁[2].鉴于此,含酚废水被中国、美国等许多国家列为重点治理的有害废水之一[3].
目前,治理含酚废水的途径主要有化学法和生物降解法.其中,利用培养优势菌群的微生物降解酚类化合物,因其投资少、处理效率高、处理量大、生态协调等优点倍受青睐.国内外学者已筛选到的苯酚降解菌,如根瘤菌(Rhizobia)、醋酸钙不动杆菌(A.calcoaceticus)、假单胞菌(Pseudonomonas.sp)、真养产碱菌(A lcaligenes eutrophus)、酵母菌(Yeast trichosporon)、反硝化菌(Denitrifiying bacteria)等[4-8]因不能耐受高浓度的苯酚,且降酚能力低未能得到广泛应用.我们拟从焦化厂排水沟污泥中筛选对苯酚具有较高耐受能力和较高降解率的菌株,为其进一步利用奠定基础.
1 材料与方法
1.1 材料
从某焦化厂排水沟不同位置采集污泥,放入具有透气瓶塞的50 mL玻璃瓶中,于4℃冰箱中保存备用.
富集培养基(g/L):牛肉浸膏5,蛋白胨10,氯化钠5,苯酚0.5,pH 7.0.
筛选培养基(g/L):以KH2PO40.5,K2HPO40.5,MgSO47H2O 0.2,CaCl20.1,NaCl 0.2,MnSO4·H2O微量,FeCl2微量,NH4NO31.0为基础培养基,加入不同浓度的苯酚配制成筛选培养基.
琼脂平板和斜面培养基(g/L):蛋白胨5,酵母膏5,葡萄糖1,苯酚0.5,琼脂20,K2HPO41,pH 7.0.
1.2 苯酚降解茵株的筛选
取污泥样品1 g,加入苯酚浓度为500 mg/L的100 mL富集培养基中,置30℃、160 r/min条件下振荡培养24 h.在无菌操作条件下取2 mL培养液,接入100 mL初始苯酚浓度为600 mg/L的筛选培养基中驯化24 h,取驯化培养液0.1 mL涂布琼脂平板培养,若有菌落长出,则提高苯酚浓度继续驯化;若无菌落长出,则将此苯酚浓度定为最终驯化浓度.如此逐级驯化,筛选培养基的苯酚浓度梯度为200 mg/L.
用梯度稀释涂布法,取最终驯化培养液0.2 mL涂布琼脂平板培养.根据菌落大小、表面形状、隆起程度、透明程度、边缘形状和菌落颜色等差异,挑取单菌落,通过反复划线分离纯化菌株,编号,保存备用.
1.3 苯酚含量测定
苯酚含量测定采用4-氨基安替比林分光光度法[9].
1.4 苯酚降解率的测定
菌种母液以2%的接种量分别接入100 mL含不同浓度苯酚(400,800,l 200,1600,2000 mg/L)的筛选培养基中,在30℃、pH 7.0和160 r/min条件下振荡培养,每隔6 h取样测定苯酚的降解率.
1.5 形态及生理生化鉴定
参照文献[10]进行菌体形态观察;参照文献[11]进行生理生化试验.
1.6 16S rDNA基因片段分离与序列分析
细菌总DNA的提取参照文献[12]的方法进行.采用细菌16S rDNA通用引物F27(5′-AGAGTTTG ATCATGGCTCAG-3′)和R1492(5′-TACGGTTACCTTGTTACGACTT-3′)扩增筛选菌株的16S rDNA基因片段[13].PCR反应条件:94℃预变性3 min,94℃变性30 s,52℃退火30 s,72℃延伸1 min,30个循环后延伸5 min.测序工作由大连宝生物工程有限公司完成.
根据测序结果,用Blast搜索程序从GenBank中调出相似性较高的相关菌株的16SrDNA基因序列,用CLUSTALX进行多序列比对,根据Kimura模型估算系统进化距离矩阵,用MEGA 3.0(Molecular-EvolutionaryGenetics Analysis)软件包采用邻接法(Neighbor-Joining)进行聚类分析和系统进化树构建[14-15].
2 结果
2.1 苯酚降解菌株的筛选
经过逐步驯化、分离和降酚能力筛选,获得1株以苯酚为惟一碳源,耐苯酚浓度达2200 mg/L的菌株,编号为HNND-3.
2.2 降解苯酚特性初步测定
菌株HNND-3在pH 7.0的不同苯酚浓度的无机盐培养基中,于30℃培养时对苯酚的降解情况,如图1.
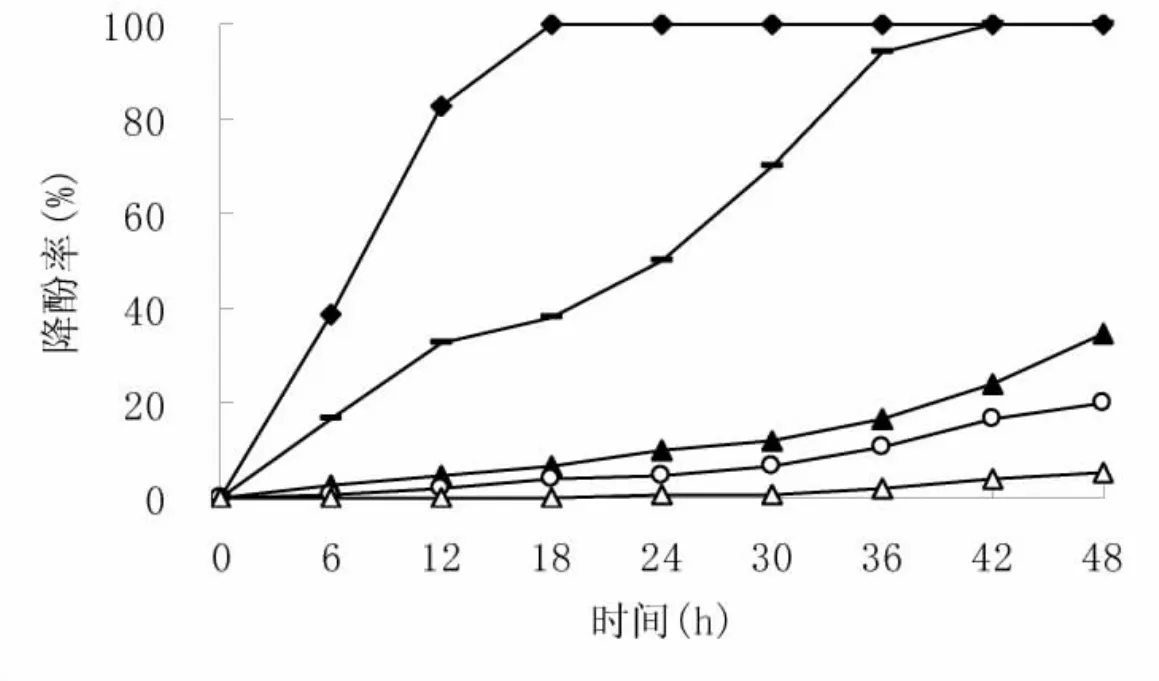
图1 不同浓度苯酚下HNND-3的降酚率
总体来说,随着起始苯酚浓度的提高,细菌生长抑制作用增强,菌株HNND-3彻底降解苯酚所需时间相应增加.对于低于800 mg/L的苯酚溶液,菌株HNND-3能以较快的速率降解.其中,400 mg/L的苯酚溶液仅18 h,800 mg/L的苯酚溶液在42 h内亦能彻底降解.但当苯酚浓度为1600 mg/L时,42 h时的降解率仅为13.1%,至2000 mg/L时细菌近乎处于休眠状态,苯酚几乎没有被降解.
2.3 苯酚降解菌株的初步鉴定
2.3.1 菌株的形态及生理生化特征菌株HNND-3在琼脂平板上培养2 d后菌落呈黄色、湿润、圆形、边缘不整齐、不光滑、有突起核心、不透明.镜检发现单个细胞呈杆状,细胞单个或多个链状排列,周生鞭毛运动,无荚膜,芽孢球形、膨大、端生或偏端生.菌株的生理生化特性检测结果见表1.根据形态和生理生化特征,可初步确定菌株HNND-3具有芽孢杆菌属(Bacillus)的典型特征,是芽孢杆菌属的成员.

表1 菌株HNND-3的生理生化特征
2.3.2 基于16S rDNA基因序列的系统发育分析以菌株HNND-3的总DNA为模板,利用细菌16S rDNA特异引物F27和R1429进行PCR扩增,成功分离出约1.5 kb的基因片段(图2).将纯化后的DNA片段测序,发现核苷酸序列长1501 bp.
利用Blast软件在GenBank等数据库中进行序列相似性搜索,发现菌株HNND-3与芽孢杆菌属菌株高度相关,这与形态和生理生化特征的鉴定结果一致.在用邻接法构建的系统进化树上,菌株HNND-3与芽孢杆菌属的典型菌株B.sphaericus DSM28T以极高的16S rDNA基因序列相似性(99.9%)和极高的自展值(bootstrap value,99%)支持聚在一起(图3),初步表明菌株HNND-3属于芽孢杆菌属有效发表种球形芽孢杆菌(B.sphaericus)的一个菌株(菌株编号为HNND-3).
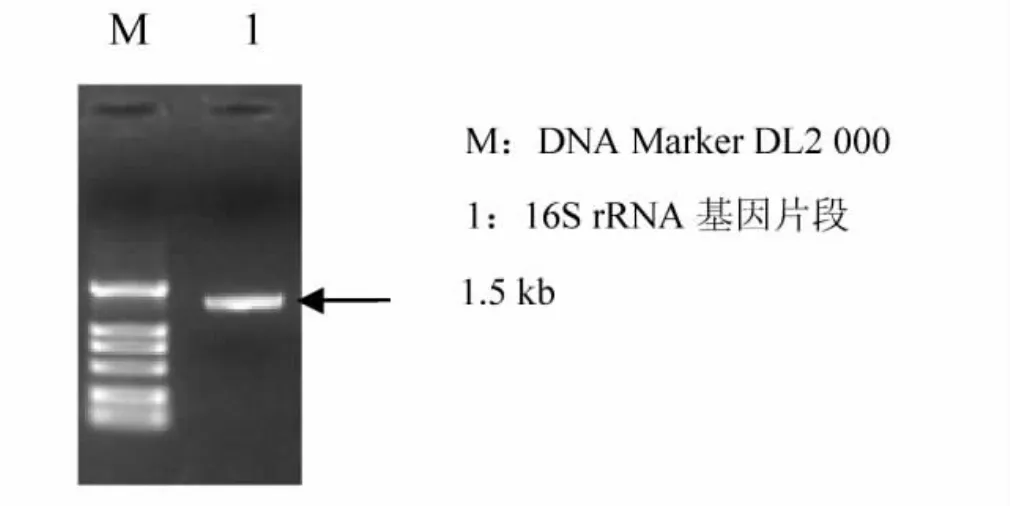
图2 菌株HNND-316S rDNA PCR扩增电泳
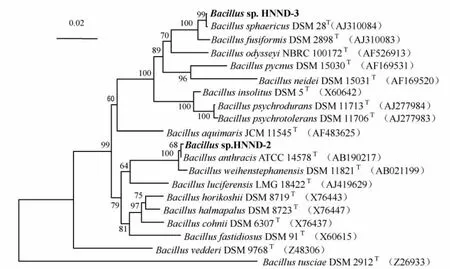
图3 苯酚降解菌株HNND-3以16S rDNA基因序列为基础的系统进化树
3 讨论
环境污染物的微生物降解法因经济有效且无二次污染而受到重视.但是,由于酚类化合物是一类能引起蛋白质变性、破坏微生物细胞膜的抑制剂,故在含酚废水处理过程中追求微生物降解苯酚能力的同时,必须考虑其对苯酚的耐受性.微生物耐受苯酚能力的提高对苯酚降解效率的提高是有利的.本研究采用以苯酚为惟一碳源的无机盐培养基筛选,并逐步提高苯酚浓度,目的是驯化和富集既能利用和降解苯酚,又能高度耐受苯酚的微生物.筛选获得的菌株HNND-3,其耐受苯酚达到2200 mg/L,这在目前分离的其他降酚菌中未见报道.通过降解苯酚能力的初步测定,发现菌株HNND-3在无机盐培养基中能将浓度为800 mg/L的苯酚较快地彻底降解.如果优化培养基组成和培养条件,其降解苯酚效率无疑将更高.目前,虽然分离了一些降解苯酚的微生物,但对苯酚降解效率高的菌种报道较少,大多数菌株的降酚能力都在600 mg/L以下[4-8].因此,菌株HNND-3是一株高效的苯酚降解菌,对其降解苯酚特性和降酚条件的深入研究,有望在含酚废水处理中获得实际应用.
[1] 王韬,李鑫钢,杜启云.含酚废水治理技术研究进展[J].化工进展,2008,27(2):231-234.
[2] 吕荣湖,付强.高浓度酚降解菌的选育及其降酚性能[J].环境科学,2005,26(5):147.
[3] 龚斌,刘津,赵斌.一株高效苯酚降解菌的分离、鉴定及降解特性的研究[J].环境科学学报,2006,26(12):2008-2012.
[4] Lee S G,Hung S P.Removal and bioconversion of phenol in wastewater by ZThermostable β-tryrosion[J].Enzyme and microbial technolgy,1996,(19):374-377.
[5] Kalin M,Neujahr H Y,Weissnahr R N,et al.Phenol hydroxylase from Trichosporon cutaneum:Gen-e cloning,sequence analysis,and functional expression in Escherichia coli[J].J Bacteriol,1992,174(2):7112-7120.
[6] 刘兴平.苯酚降解茵的分离及降解特性研究[J].环境保护科学,2008,34(2):38-40.
[7] Peters M,Heinary E,Talpsep E,et al.Acquisition of a deliberately introduced phenol degradation operon,pheBA,by different indigenous Pseudomonas species[J].Appl Environ Microbiol,1997,63(2):4899-4906.
[8] Paula M,Schie L,Yong Y.Isolation and characterization of phenol-degradation denitrifying bacteria[J].Appl Environ Microbiol,1998,64(7):2432-2438.
[9] 国家环保局《水和废水监测分析方法》编委会.水和废水监测分析方法[M].北京:中国环境科学出版社,1997:1-410.
[10] 沈萍,范秀荣,李广武.微生物学实验[M].北京:高等教育出版社,2002.
[11] 东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001.
[12] F.奥斯伯,R.布伦特,R.E.金斯盾,等.精编分子生物学实验指南[M].北京:科学技术出版社,2005:1-119.
[13] Weissburg WG,Barns SM.16Sribosomal DNA amplification for phylogenetic study[J].J.Bacteriol.,1991,173:687-703.
[14] Thompson J D,Gibson T J,Plewniak F.The Clustal X windows interface:flexible s-trategies for multiple sequence alignment aided by quality analysistools[J].Nucleic Acids Research,1997,24(4):876-488.
[15] Kumar S,Tamura K,Jakobsen,et al.MEGA2:Molecular Evolutionary Genetics Analysis software[J].Bioinformatics,2001,17:1244-1245.

