烟台海域暴发浒苔ITS及5.8S rDNA的克隆及序列分析
宁璇璇,纪灵,王刚,刘艳,于道德,宋培高,唐海田,伯云台
(1.国家海洋局烟台海洋环境监测中心站,山东 烟台 264006;2.山东省海水养殖研究所,山东 青岛 266071)
烟台海域暴发浒苔ITS及5.8S rDNA的克隆及序列分析
宁璇璇1,纪灵1,王刚1,刘艳1,于道德2,宋培高1,唐海田1,伯云台1
(1.国家海洋局烟台海洋环境监测中心站,山东 烟台 264006;2.山东省海水养殖研究所,山东 青岛 266071)
以烟台海域引发“绿潮”的浒苔为研究对象,采用PCR技术扩增出浒苔的ITS-1、5.8S rDNA及ITS-2片段,将扩增出的片段纯化后克隆至pGEM-T Easy载体,筛选阳性克隆进行序列测定。结果表明,浒苔的ITS-1序列长度为195 bp,5.8S序列为155 bp,ITS-2序列为181 bp,该序列与浒苔属的多种物种ITS序列具有很高的同源性,在ITS-1区、5.8S rDNA区和ITS-2区仅存在4个转换/颠换位点。结合GenBank注册序列和本研究的结果发现,单纯依靠ITS序列并不能对浒苔属种类进行有效的分类鉴定。
浒苔;内转录间隔区 (ITS);5.8S rDNA;序列分析
浒苔Enteromorpha prolifera(Muell.) J.Ag.隶属于绿藻门,绿藻纲,石莼目,浒苔属,是中国海洋野生植物资源极为丰富的大型经济藻类,广泛分布于中、低潮区的砂砾、岩石潮滩或石沼中[1]。
近年来,海洋污染的加剧造成了近海海域富营养化日益严重,导致藻华发生的频率增多、规模扩大、危害程度加重。除了赤潮的频繁暴发外,浒苔属 (Enteromorpha)、石莼属 (Ulva) 的多种绿藻也可在近岸海域形成“绿潮”(green tide)[2-5]。2007年夏季,青岛海域出现了浒苔过度繁殖造成的“绿潮”,2008年入夏以来,黄海海域再次出现了严重的浒苔过度繁殖现象,烟台海阳海域也发生了浒苔“绿潮”现象。浒苔的过度繁殖将对海洋生态系统产生严重的负面作用,不仅可造成海洋沉积物生物地球化学循环的解耦合[6],而且能够通过遮蔽阳光影响海床生物的正常生长[7]。
为探讨浒苔过度增殖的机制,除了开展浒苔来源及分布情况的相关调查外,还需要利用分子鉴定手段对浒苔来源及其动态变化过程进行快速分析。ITS (Internal Transcribed Spacer) 是核糖体DNA中18 S和28S rDNA之间的内转录间隔区,由于ITS序列在生物进化过程中显示种的特征,因而可以选择其中保守程度不同的片段作为分子标记来鉴定生物体不同种、亚种和地理株,以及研究其系统进化关系[8-10]。目前,在赤潮藻的分子鉴定方面,ITS序列已得到了较为广泛的应用[11-16]。
本研究以采自烟台海阳海域引发“绿潮”的浒苔为研究对象,通过PCR扩增技术获得了浒苔的ITS和5.8S rDNA序列并进行了相关分析,探讨了其作为浒苔分子鉴定标记的可能性和局限性。
1 材料与方法
1.1 实验材料
本研究中所用浒苔样品采集自烟台海阳海域,样品经蒸馏水反复冲洗,洗净后直接用于DNA提取。
1.2 菌株与质粒
大肠杆菌Escherichia coliJM109感受态细胞、pGEM-T easy Vector购自Promega公司。
1.3 主要试剂及仪器
氯仿、异戊醇、异丙醇购自上海生物工程有限公司;β-巯基乙醇、CTAB、PVP等购自Sigma公司;Ex-Taq酶、DL2000 DNA Marker购自大连宝生物工程有限公司;DNA片段凝胶回收试剂盒为Omega公司产品;其他常用试剂购自国药集团化学试剂有限公司。
主要仪器包括:高速冷冻离心机(Eppendorf,Eppendorf Centrfuge 5804R)、PCR仪(Biometra,T3 thermocycler)、电泳仪(北京君意,JY200系列)、凝胶成像系统(Bio-Rad,Quantity one system)等。
1.4 引物
根据绿藻纲18S和28S rDNA的保守序列设计PCR扩增引物(F1: 5’-ACAAGGTTTCCGTAGGTGA-3’;R1: 5’-TTTAGTTTCTTATCCTCCG-3’),委托大连宝生物工程有限公司合成。
1.5 浒苔DNA提取
浒苔DNA的提取参照CTAB法[17,18],并略做改动,主要步骤如下:浒苔样品经液氮充分研磨后,加入适量裂解缓冲液(3% CTAB,100 mmol/L Tris-HCl,100 mmol/L EDTA,1.4 mol/L NaCl,0.2% β-巯基乙醇,1% PVP),置于60℃温浴30 min;向上述体系中加入1/3体积的5 mol/L KAc,充分混匀,加入等体积的氯仿-异戊醇(24:1),轻缓摇匀后,8 000 r/min离心10min;移取上清,加入2/3体积的异丙醇,-80℃放置30 min;8 000 r/min离心10 min,弃上清,沉淀用70%乙醇洗涤两次;沉淀晾干后用适量无菌水溶解,8 000 r/min离心10 min,取上清备用。
1.6 PCR扩增
以提取的浒苔DNA为模板,经PCR扩增出ITS及5.8S rDNA片段,扩增体系为25 μL:含有50 ng DNA模板,2 mmol/L MgCl2,0.2 mmol/L dNTP,0.2 µmol/L 引物,1.25单位Ex-Taq。PCR反应条件如下:94℃预变性5 min,94℃变性30 s,50℃退火30 s,72℃延伸30 s,进行30个循环,最后72℃延伸5 min。
1.7 扩增片段的克隆及筛选
PCR扩增产物以1.0%琼脂糖凝胶电泳检测,用凝胶成像系统Quantity one system观察并拍照记录结果。以DNA片段凝胶回收试剂盒对所扩增片段进行纯化,纯化产物连接到pGEM-T Easy载体上,将连接产物转化E.coli感受态细胞JMl09。采用蓝白斑法挑选阳性克隆,选取经PCR鉴定为阳性的5个克隆,送大连宝生物工程有限公司进行双向测序。
1.8 序列分析
利用http://www.ncbi.nlm.nih.gov/提供的在线软件工具对获得的序列进行同源性分析[19],表明所获得的序列为ITS区域。采用Clustal W软件[20]对所测得的浒苔ITS序列和GenBank中注册的浒苔属其它种类的ITS序列进行多重对位排列 (Multiple alignments)。
2 实验结果与讨论
2.1 浒苔ITS及5.8S rDNA序列的扩增
海洋藻类通常具有坚硬的细胞壁,而且多糖含量较高,提取的模板DNA用于PCR扩增时往往效率不高。本研究采用改良的CTAB方法,辅以氯仿/异戊醇抽提,提取的浒苔DNA样品具有良好的PCR扩增效果。
以上述提取的浒苔DNA为模板,以F1和R1作为引物进行PCR扩增。PCR产物经1.0%琼脂糖凝胶电泳检测,可见约650 bp的条带(见图1),与预期目的片段大小一致。
2.2 序列测定与分析
纯化上述PCR产物,连接至pGEM-T easy载体,PCR筛选阳性克隆进行序列测定。测序结果表明,该引物对扩增产物长度为643 bp,包含了部分18S和28S rDNA 序列。比对分析表明,烟台海域引发“绿潮”的浒苔ITS-1序列为195 bp,5.8 S序列为155 bp,ITS-2序列为181 bp。该序列已经提交GenBank数据库,注册号为FJ539192。
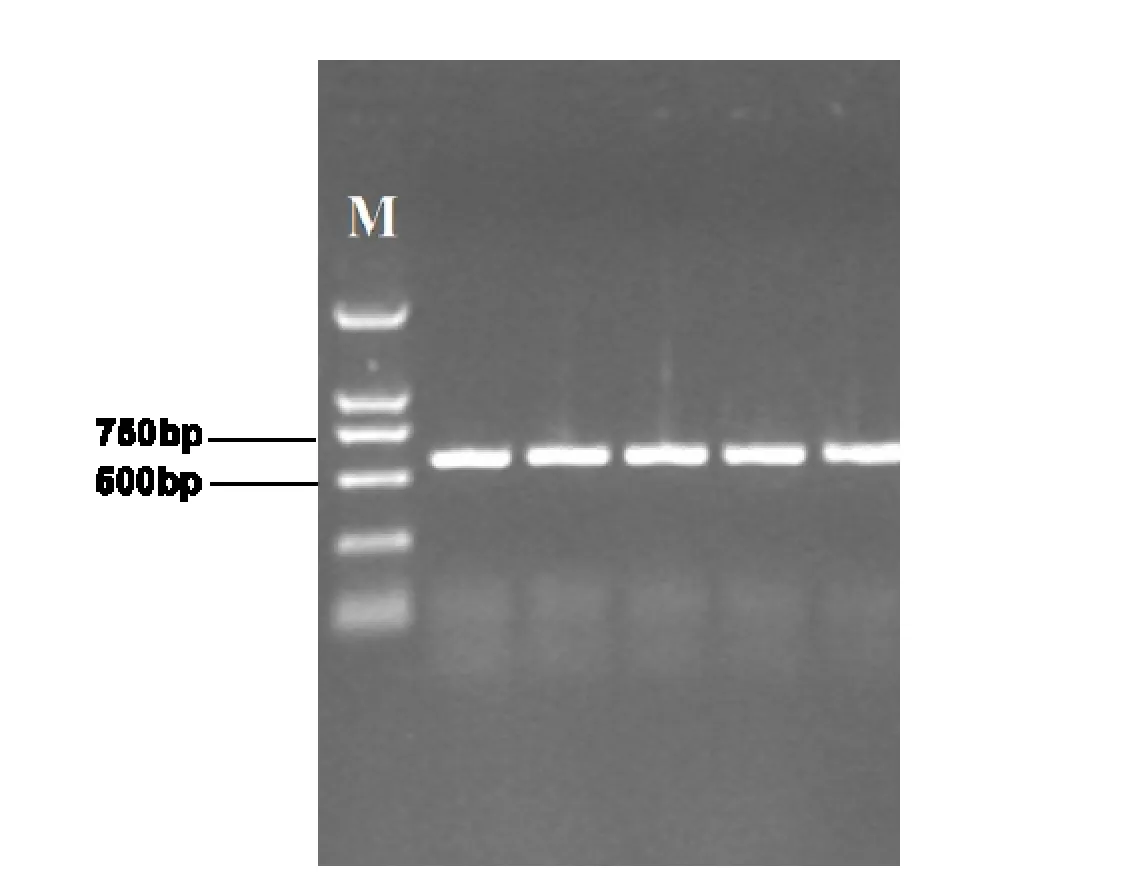
图 l 烟台海域浒苔ITS rDNA的PCR扩增产物Fig.1 The amplified product of ITS rDNA from Enteromorpha prolifera around the coast of Yantai by PCR technique
Blastn分析结果显示,烟台海域浒苔ITS序列与GenBank数据库中浒苔属的浒苔(GenBank注册号:AB298314)、缘管浒苔Enteromorpha linza(GenBank注册号:AJ000203)、Enteromorpha ahlneriana(GenBank注册号:AJ012276)、Enteromorpha procera(GenBank注册号:AY260558)ITS序列均具有高度同源性。多重比对结果表明,浒苔属各物种的ITS序列非常保守,在ITS1区、5.8S rDNA区和ITS2区总共存在4个转换/颠换位点(图2)。
2.3 讨论
ITS是介于核糖体18S和5.8S rDNA之间(ITS-1)以及5.8S和28S rDNA之间(ITS-2)的非编码间隔区。由于ITS序列变异较快,可提供较丰富的变异位点和信息位点,通常具有种内相对一致、种间差异比较明显的特点。这些特点使得ITS区适合于属内物种间或种内差异较明显的种群间系统发育关系的分析,而且已被广泛应用于绿藻的分子鉴定[21-24]。结合GenBank注册序列和本研究的结果发现,单纯依靠ITS序列并不能对浒苔属种类进行有效的分类鉴定。上述原因的产生可能是由于浒苔属各物种的分化较晚,ITS区域尚未形成显著的差异。因此,有必要采用其它的分子标记技术,并结合浒苔属各物种的形态特征、生活史等方面进行分类鉴定。

图 2 烟台海域浒苔的ITS序列与浒苔属其它物种的多序列比对结果Fig.2 Alignment of the ITS sequences of Enteromorpha prolifera from Yantai coast and four other species in the same genus
[1]曾呈奎, 张德瑞, 张竣甫.中国经济海藻志 [M].北京: 科学出版社, 1962: 43-50.
[2]Schories D, Reise K.Germination and anchorage ofEnteromorphaspp.in sediments of the Wadden Sea [J].Helgoländer Meeresuntersuchungen, 1993, 47: 275-285.
[3]Fletcher R L.Marine benthic vegetation: recent changes and the effects of eutrophication [M].Berlin: Springer press, 1996: 7-43.
[4]Tan I H.Molecular phylogenetic evidence for a reversible morphogenetic switch controlling the gross morphology of two common genera of green seaweeds,UlvaandEnteromorpha[J].Molecular Biology and Evolution, 1999, 16: 1 011-1 018.
[5]Blomster J, Back S, Fewer D P, et al.Novel morphology inEnteromorpha (Ulvophyceae) forming green tides [J].American Journal of Botany, 2002, 89: 1 756-1 763.
[6]Valiela I, Mcclelland J, Hauxwell J, et al.Macroalgal blooms in shallow estuaries: controls and ecophysiological and ecosystem consequences [J].Limnology and Oceanography, 1997, 42: 1 105-1 118.
[7]Raffaelli D G, Raven J A, Poole L J.Ecological impact of green macroalgal blooms [J].Oceanography and Marine Biology: An Annual Review, 1998, 36: 97-125.
[8]Kimura M.Evolutionary rate at the molecular level [J].Nature, 1968, 217: 624-626.
[9]Dover G.Molecular drive: a cohesive mode of species evolution [J].Nature, 1982, 299: 111-117.
[10]David M H, Scott K D.Ribosomal DNA: intraspecific polymorphism, concerted evolution and phylogeny reconstruction [J].Systematic Zoology, 1988, 37: 63-66.
[11]庄丽, 陈月琴, 李钦亮, 等.赤潮叉角藻18S rDNA和ITS区序列测定与分析 [J].海洋与湖沼, 2001, 32 (2) : 148-153.
[12]苟万里, 刘东艳, 甄毓, 等.利用rDNA和ITS序列对一株裸甲藻的初步鉴定 [J].中国海洋大学学报, 2004, 34 (1) : 75-83.
[13]张宝玉, 王广策, 张炎, 等.东海原甲藻 (Prorocentrum donghaiense) 和海洋原甲藻APBM (P.micansAPBM) 的5.8S rDNA及其转录间隔区(ITS) 的克隆和序列分析 [J].海洋与湖沼, 2004, 35 (3): 264-271.
[14]张宝玉, 王广策, 吕颂辉, 等.三株赤潮硅藻5.8S rDNA及转录间隔区 (ITS) 的克隆及序列分析 [J].海洋学报, 2006, 28 (1): 111-117.
[15]黄健, 梁姗, 秦洁.利用ITS-5.8S rDNA序列对2株海洋亚历山大藻进行的分子鉴定 [J].中国海洋大学学报, 2008, 32 (2): 285-290.
[16]陈国福, 王广策, 张春云, 等.一株裸甲藻类似种的形态和系统进化分析 [J].科学通报, 2008, 53 (3), 299-305.
[17]Doyle J J, Doyle J L.Isolation of plant DNA from fresh tissue [J].Focus, 1990, 12: 13-15.
[18]Hughey J R, Silva P C, Hommersand M H.Solving taxonomic and nomenclatural problems in PacificGigartinaceae(Rhodophyta) using DNA from type material [J].Journal of Phycology, 2001, 37: 1 091-1 109.
[19]Zhang Z, Schwartz S, Wagner L, et al.A greedy algorithm for aligning DNA sequences [J].Journal of Computational Biology, 2000, 7: 203-214.
[20]Larkin M A, Blackshields G, Brown N P, et al.ClustalW2 and ClustalX version 2 [J].Bioinformatics, 2007, 23: 2947-2948.
[21]Kooistra W H C F, Stam W T, Olsen J L, et al.Biogeography ofCladophoropsis membranacea (Chlorophyta) based on comparisons of nuclear rDNA ITS sequences [J].Journal of Phycology, 1992, 28: 550-668.
[22]Coleman A W, Suarez A, Goff L J.Molecular delineation of species and syngens in Volvocacean green algae (Chlorophyta) [J].Journal of Phycology, 1994, 30: 80-90.
[23]Marks J C, Cummings M P.DNA sequence variation in the ribosomal internal transcribed spacer region of freshwaterCladophoraspecies (Chlorophyta) [J].Journal of Phycology, 1996, 32: 1 035-1 042.
[24]Blomster J, Maggs C A, Stanhope M J.Molecular and morphological analysis ofEnteromorpha intestinalisandE.compressa(Chlorophyta) in the British Isles [J].Journal of Phycology, 1998, 34: 319-340.
Cloning and sequence analysis of the ITS and 5.8S rDNA ofEnteromorpha proliferaforming green tide around the coast of Yantai
NING Xuan-xuan1, JI Ling1, WANG Gang1, LIU Yan1, YU Dao-de2,
SONG Pei-gao1, TANG Hai-tian1, BO Yuntai1
(1.Yantai Oceanic Environmental Monitoring Central Station of State Oceanic Administration,Yantai 264006, China; 2. Mariculture Institute of Shandong Province, Qingdao 266071, China)
The internal transcribed spacer and 5.8S rDNA ofEnteromorpha proliferaforming green tide around the coast of Yantai were amplified by polymerase chain reaction (PCR) .The resultant PCR product was cloned into pGEM-T Easy vector and subjected to nucleotide sequencing.Sequence analysis revealed that the ITS-1, 5.8 S and ITS-2 rDNA of E.prolifera were of 195 bp, 155 bp and 181 bp in length, respectively.TheEnteromorpha proliferaITS sequence shared significant homology with other reported ITS sequences ofEnteromorphawith only one transition site and three transversion sites.These results suggested that analysis of ITS sequence could not effectively distinguish the species ofEnteromorpha.
Enteromorpha prolifera; internal transcribed spacer; 5.8S rDNA; sequence analysis
Q179.1; Q178.531
A
1001-6932 (2010) 01-0091-05
2008-12-30;
2009-06-17
宁璇璇(1983-),女,山东蓬莱人,助理工程师,硕士,主要从事海洋环境监测与评价工作。电子邮箱:ningxx05@163.com通讯作者:于道德(1978-),男,山东青岛人,博士。电子邮箱:wensentte@163.com

