齿瓣石斛AP3基因的克隆及序列分析
陈 凯,应奇才,徐祥彬,沈 波,王慧中
(杭州师范大学 生命与环境科学学院,浙江 杭州 310036)
花发育作为植物生活史的重要环节,是植物发育中最为引人注目的阶段[1].自Coen和Meyerowitz(1991)[2]经典ABC模型提出,已经鉴别和克隆出许多与花器官发育相关的重要基因,并逐渐完善为A-E发育模型.B类AP3基因属于植物中广泛存在的序列特异的同源异型MADS-box家族,它所编码的蛋白转录因子在生物体中通过调节其他基因的转录来发挥其调控作用[3-4].拟南芥中有AP3和PI两个B功能基因,二者定位于不同染色体上,共同控制花瓣和雄蕊的发育[5].
石斛属(Dendrobium)是兰科第二大属,全球共有1 500多种,主要分布于热带东南亚及大洋洲,我国有76种,主要分布在华南及西南地区[6].石斛因其特殊的观赏性能和药用功效广受研究者的青睐.对于石斛的研究国外着重于石斛花发育基因的功能分析和植株培养条件的优化;国内则侧重于石斛药用性能、组培苗的菌根化研究[7-9].为了揭示石斛花发育的分子机理,通过转基因提升石斛观赏价值,该文利用RT-PCR和RACE技术从齿瓣石斛花序中克隆了DdAP3基因的全长cDNA序列,并对DdAP3同源蛋白的理化性质、二级结构、疏水性/亲水性、功能结构域及其高级结构和同源性等进行了分析.
1 材料与方法
1.1 材料
1.1.1 植物材料
植物材料为兰科石斛属齿瓣石斛(DendrobiumDevonianum),取自该实验室药用花卉植物栽培中心,初夏取开花不久的石斛花序用于总RNA提取.
1.1.2 菌种质粒
克隆载体pMD19-T购自Takara公司,E.coli DH5α由本实验室保存.
1.1.3 试 剂
核酸限制性内切酶、PfuDNA Polymerase、M-MLV反转录体系购自Promega公司,cDNA末端快速扩增(RACE)试剂盒3′-Full RACE Kit和5′-Full RACE Kit、T4DNA连接酶购自Takara公司,DNA片段回收纯化试剂盒购自上海生工.其它生化试剂和常规试剂均为超纯和分析纯.
1.2 方 法
1.2.1 AP3基因保守序列的克隆
根据GenBank登录的烟草、水稻、小麦、大麦和拟南芥AP3基因的保守序列设计合成了一对引物AP3-F (5′-GAA CTG GAG GGG ATG GAA AT-3′)和AP3-R (5′-CAC ATG ATC TTA GCC TCG CA-3′).齿瓣石斛花序总RNA提取采用改良后樊洪泓等[10]的方法进行,cDNA第一链合成参照M-MLV逆转录酶的20 μL标准反应体系:2 μg模板总RNA、0.5 g·L-1Oligo(dT)151 μL、加入DEPC灭菌水至12.5 μL;70 ℃水浴5 min,冰上骤冷2 min;加入5×Reaction Buffer 4 μL、40 u·μL-1RNA酶抑制剂0.5 μL、10 mmol·L-1dNTP 2 μL,37 ℃孵育5 min;加入200 μ·μL-1M-MLV反转录酶1 μL,轻微混匀,42 ℃反应1 h,70 ℃ 15 min终止反应,-20 ℃保存备用.PCR反应总体积为25 μL,其中包含3 μL cDNA第一链,10 mmol·L-1Tris-HCl (pH 8.3),50 mmol·L-1KCl,2.0 mmol·L-1MgCl2,100 μmol·L-1dNTPs,0.3 μmol·L-1引物,Pfu酶1 U.反应程序为:94 ℃ 4 min;94 ℃ 45 s、56 ℃ 45 s,72 ℃ 60 s,33个循环;72 ℃ 10 min.扩增产物于1.2%琼脂糖凝胶上电泳分离,回收纯化,产物的连接按pMD19-T Vector(Takara)的说明书进行,重组质粒的转化、鉴定按Sambrook[11]的常规方法进行.阳性克隆送上海生物工程公司测序.
1.2.2 AP3基因全长序列的克隆
根据获得的石斛AP3基因保守序列设计合成特异性巢式PCR引物:5′DdAP3 outer (CAG TGC AGA AAA TCA CAA CAG AG)、5′DdAP3 inner (GAC TCT TCC AAA GTT TGC TCA AG)、3′DdAP3 outer (CTT GAG CAA ACT TTG GAA GAG TC)和3′DdAP3 inner (CAG TGC AGA AAA TCA CAA CAG AG).采用基于RNA连接酶的RACE技术进行cDNA的5′和3′端扩增,PCR产物经电泳分离、纯化回收、连接转化后送上海生工测序.
根据测序结果设计合成该基因全长引物DdAP3-F(CAA AAC AGA GCA AAA CAG AGC A)和DdAP3-R(TTG GTG AAA CTG ACA ATT TGA GA),以cDNA第一链为模板进行PCR扩增.利用Blastn程序将获得的核苷酸序列在GenBank数据库中进行同源性序列搜索比对,并利用DNAMAN6.0翻译成氨基酸序列,再与其它作物的AP3同源蛋白进行序列比对.
1.2.3 生物信息学分析
用Expasy的ProtParam程序(http://cn.expasy.org/tools/protparam.html)统计齿瓣石斛AP3蛋白中各种氨基酸含量,并预测得到理论分子量和等电点;通过在线分析软件(http://www.cbs.dtu.dk/services/SignalP)进行DdAP3蛋白质序列的信号肽分析;应用ProtScale程序(http://www.expasy.ch/tools/protscale.html)计算蛋白质的疏水性.通过网站Expasy的GOR4方法对该蛋白进行二级结构分析.使用NCBI数据库中的Protein Blast分析全长序列中的蛋白功能结构域;从Genbank数据库搜索其它物种AP3同源基因,利用DNAMAN6.0进行多重序列比对,建立相应的系统关系树.
2 结果与分析
2.1 齿瓣石斛AP3基因全长序列的获得
根据模式植物AP3同源基因序列设计保守引物,以齿瓣石斛mRNA反转录的cDNA为模板进行PCR扩增,测序获得与预期大小相一致的348 bp片段.根据保守序列设计合成RACE引物,使用RACE Kit(Takara)方法进行DdAP3基因的5′和3′端序列扩增,试验结果显示5′和3′RACE均能扩增出目的条带.将特异性条带切胶回收纯化,克隆测序,5′和3′端序列经电子拼接后设计合成全长引物,扩增得到786 bp全长序列,编码227个氨基酸(GenBank accession No. GU126414.1).
2.2 DdAP3蛋白的理化性质分析
利用http://cn.expasy.org/tools/protparam.html网站对DdAP3基因所编码的227个氨基酸序列进行分析,预测结果表明:AP3蛋白的分子量是26 483.3 D;理论等电点是9.1;原子组成是C1162H1883N335O351S10;负电荷残基(Asp+Glu)30个,正电荷残基(Arg+Lys)36个;不稳定系数(Instability index)为26.35,表明该蛋白质状态稳定;脂肪系数为85.42;总平均亲水性为-0.694,说明该蛋白是一个疏水性蛋白.DdAP3蛋白由20种氨基酸组成,其中Leu、Glu、Lys含量最为丰富,Phe、Cys、Trp含量最少.
2.3 DdAP3蛋白二级结构预测
用GOR4方法(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)对齿瓣石斛AP3蛋白进行了二级结构分析.结果表明(见图1),该蛋白的二级结构中49.78%为螺旋结构(Helices),线状结构与卷曲结构为12.78%和37.44%.由此可推测,螺旋结构和卷曲结构散布于其整个蛋白质中,构成DdAP3蛋白质二级结构的骨架.
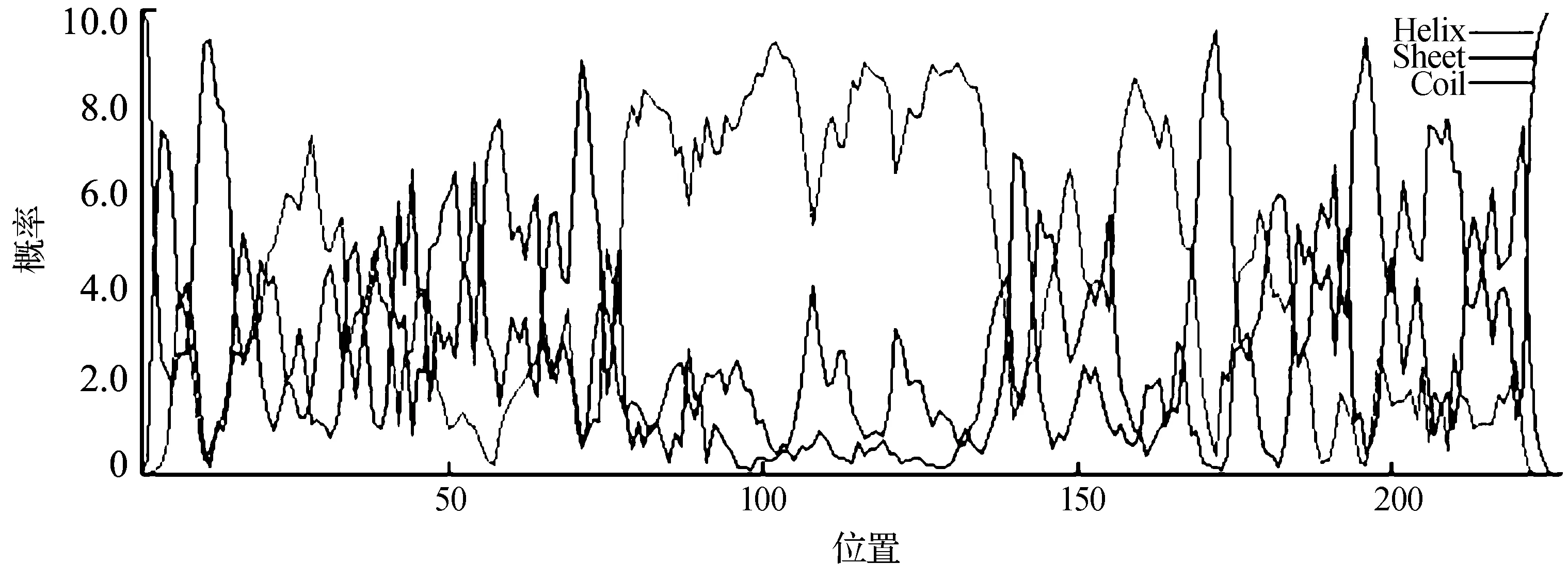
图1 石斛AP3蛋白二级结构预测Fig. 1 Predicting of second structure of DdAP3 protein
2.4 DdAP3蛋白的疏水性/亲水性预测
用ProtScale对AP3蛋白的氨基酸序列的疏水性/亲水性进行预测(见图2).结果表明:多肽链的第45号与47号的亮氨酸(Leu)和甲硫氨酸(Met)分别具有最高的分值1.544和1.567,亲水性最强,第106号的精氨酸(Arg)具有最低的分值为-2.689,疏水性最强.
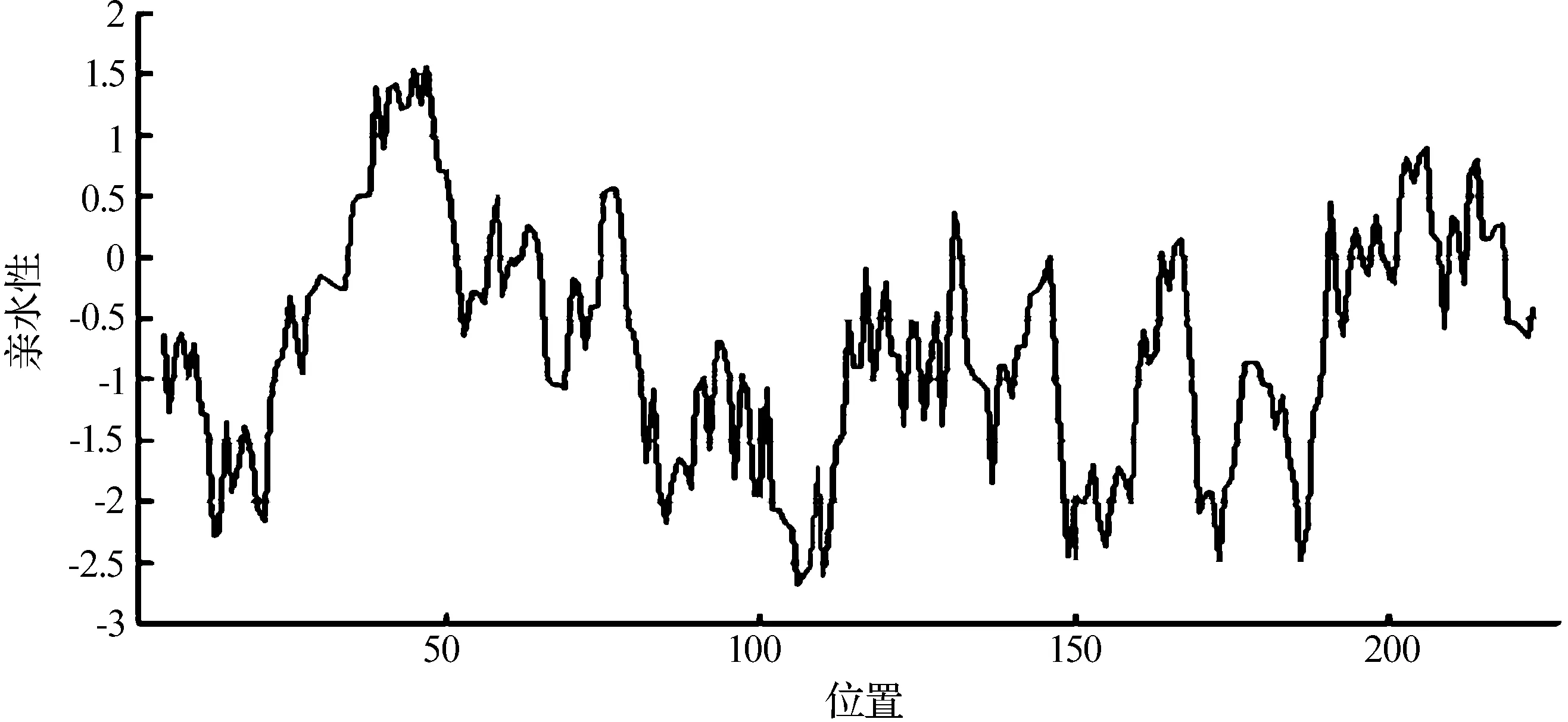
图2 AP3蛋白的疏水性/亲水性曲线Fig. 2 Analysis for hydrophobicity/hydrophilicity of DdAP3 protein
2.5 DdAP3蛋白的信号肽预测
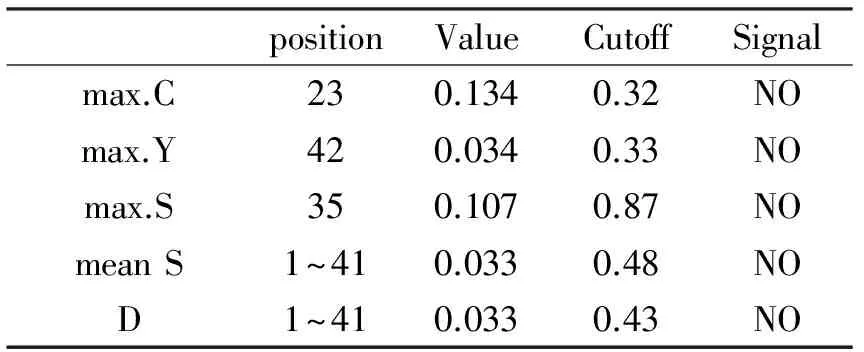
表1 DdAP3蛋白的信号肽预测结果(长度=70)
利用http://www.cbs.dtu.dk/services/SignalP网站对DdAP3蛋白的信号肽进行预测.分析结果如表1所示,第23位氨基酸残基具有最高的原始剪切位点分值0.134,第35位氨基酸残基具有最高的信号肽分值0.107,第1位到第41位氨基酸残基的信号肽分值为0.033,第42位氨基酸残基具有最高综合剪切位点,分值为0.034.由此可以推断,齿瓣石斛AP3蛋白不存在信号肽,是非分泌蛋白,在细胞质中合成后,不进行蛋白转运.
2.6 DdAP3蛋白的高级结构预测
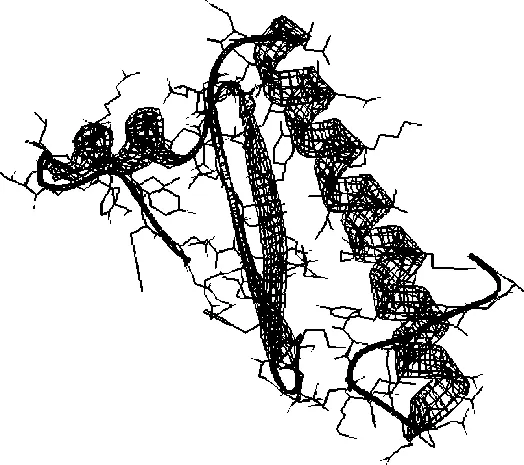
图3 DdAP3蛋白的三维结构预测Fig. 3 Higher structure analysis of DdAP3 protein
利用Expasy的工具中Swiss-model把DdAP3蛋白的氨基酸序列生成原子坐标(PDB格式),然后用spdbv软件观看编辑该蛋白的三维立体分子结构.从图3中可以看出,该蛋白高级结构主要由绿色部分的α-螺旋结构和β-折叠组成,灰色标志的区域为线状结构.这与图2的蛋白质二级结构预测结果相一致.
2.7 DdAP3蛋白的保守结构域分析
使用NCBI数据库中的Blastp分析全长序列中可能的蛋白功能结构域.结果表明(见图4),齿瓣石斛AP3蛋白有1个MADS-box家族保守结构域和1个K-box结构域.N端为约70个氨基酸的MADS结构域,中度保守;中部约100个氨基酸的半保守K-box结构域.K-box因其结构类似于卷曲螺旋而得名,是蛋白因子之间相互作用的区域,与基因转录有关.
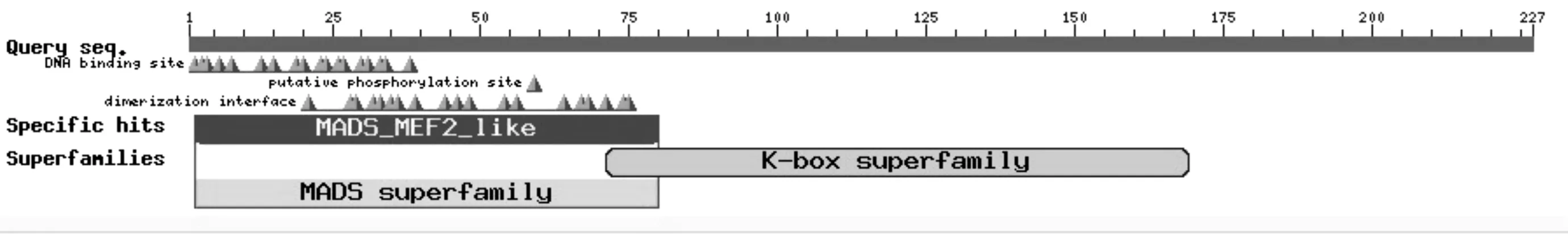
图4 AP3蛋白保守结构域预测Fig. 4 Conserved domains of DdAP3 protein
2.8 DdAP3蛋白同源性比较
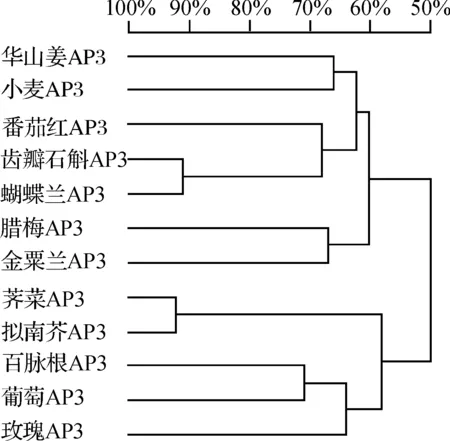
图5 齿瓣石斛与其它植物AP3基因编码的氨基酸序列同源性树状图Fig. 5 Phylogenetic tree analysis of homologous AP3 protein
齿瓣石斛DdAP3蛋白(D.devonianum, ACY68439.1)与华山姜(Alpiniaoblongifolia, EF52817.1)、小麦(Triticumaestivum, BAD15267.1)、荠菜(Brassicajuncea, DQ 060332.1)、腊梅(Chimonanthuspraecox, ABK34952.1)、金粟兰(Chloranthusspicatus, AAR06664.1)、番茄红(Crocussativus, AY948339.1)、百脉根(Lotusjaponicus, AAX13301.1)、蝴蝶兰(Phalaenopsisaphrodite, AAV28492.1)、拟南芥(Arabidopsisthaliana,AY070397)、玫瑰(Rosarugosa,BAC79180.1)和葡萄(Vitisvinifera,ACZ26526.1)通过DNAMAN6.0进行同源性比对和相似性分析.图5和图6结果表明,齿瓣石斛AP3蛋白与蝴蝶兰、荠菜和拟南芥之间的同源性最高达到92%,平均相似性为60.27%,其中N端64个氨基酸的平均相似性高达84.62%,中间87个氨基酸相似性为69.70%,这与NCBI预测的蛋白保守结构域的结果相符,表明不同植物的AP3同源基因其编码的氨基酸序列在近N端有较高的同源性,该区域属于高等植物非常保守的MADS基序(motif),中间序列为相对保守的K-box结构域,是转录因子的一个重要特征,因而推测获得的AP3同源基因可能具有转录因子的功能.

图6 齿瓣石斛与其它植物AP3基因编码的氨基酸序列比较Fig. 6 Multiple sequence Alignment of AP3 homologous protein
3 讨 论
在植物的整个生活周期中,开花是植物生长发育的核心过程,除了外部环境因素外,内部发育信号影响植物从营养生长向生殖生长阶段的状态转变.目前,植物花发育的经典ABC模型已经发展为ABCDE模型,许多控制花色、花形、花香和开花时间的调控因子及相互作用机制也已经被提出.植物花发育的分子机制、基因表达调控始终是一个令人兴奋和极具前瞻性的研究热点领域.文章利用RT-PCR和RACE技术从药用观赏性植物石斛的花序中克隆到花发育MADS-box家族AP3基因全长cDNA序列.生物信息学分析显示,DdAP3是分子量为26.5 KD、理论等电点9.1的碱性蛋白,主要由螺旋和卷曲结构组成的疏水性蛋白,包含有保守的MADS和半保守K-box结构域,与拟南芥、蝴蝶兰和荠菜的AP3蛋白高度同源.DdAP3的克隆为揭示石斛花发育的分子机理、提升石斛观赏价值鉴定了基础.
[1] 许智宏,刘春明.植物发育的分子机理[M].北京:科学出版社,1998:89-104.
[2] Coen E S, Meyerowitz E M. The war of the whorls: genetic interactions controlling flower development[J]. Nature,1991,353(6339):31-37.
[3] Eckardt N A. Functional divergence ofAP3 Genes in the MAD world of flower development[J]. Plant Cell,2006,18(8):1779-1781.
[4] Kater M M, Dreni L, Colombo L. Functional conservation of MADS-box factors controlling floral organ identity in rice and Arabidopsis [J]. Journal of Experimental Botany,2006,57(13):3433-3444.
[5] Clinton J W, Pietro C, Christopher M P,etal. Conservation of B-class floral homeotic gene function between maize and Arabidopsis[J]. Development,2004,131:6083-6091.
[6] Lavarack P S, Wayne F, Geoff Stocker. Dendrobium and its relatives[M]. Portland: Timber Press,2002.
[7] Mitra A S, Dey S, Sawarkar S K. Photoautrophic in vitro multiplication of the orchid Dendrobium under CO2enrichment[J]. Biological plantarum,1998,41(1):145-148.
[8] Xu Hong, Liu Jun, Wang Zhengtao,etal. Studies on tissue culture of Dendrobium chrysotoxum[J]. China Journal of Chinese Material Medica,2001,21(6):378-391.
[9] 黄运峰,杨小波.兰科菌根研究综述[J].热带亚热带植物学报,2008,16(3):283-288.
[10] 樊洪泓,李廷春,林毅,等.石斛总RNA提取方法的研究[J].激光生物学报,2007,16:105-108.
[11] Sambrook J, David W R.分子克隆实验指南[M].黄培堂,译.3版.北京:科学出版社,2002.

