中国沿海10种方蟹16S rRNA基因序列分析及系统发育研究
徐敬明
(重庆文理学院 生命科学与技术学院, 重庆 永川 402168)
中国沿海10种方蟹16S rRNA基因序列分析及系统发育研究
徐敬明
(重庆文理学院 生命科学与技术学院, 重庆 永川 402168)
中国沿海10种方蟹线粒体16S rRNA基因部分片段的序列长度为517 bp~533 bp。它们的核苷酸序列A、T、G、C的含量相似, A+T的含量(69.8%~76.0%)明显高于G+C的含量; 10种方蟹的16S rRNA基因序列比对获得541 bp的同源序列(含插入/缺失位点), 除插入/缺失位点外共检测到146个变异位点, 其中81个为简约信息位点。4种厚蟹与2种近方蟹的遗传距离(0.054~0.085)都显著小于与其他方蟹之间的遗传距离, 甚至明显小于与4种厚蟹原本属于同一相手蟹科的2种相手蟹之间的遗传距离(0.105~0.155); 而基于16S rRNA基因片段序列采用NJ法构建的系统进化树的拓扑结构也显示, 原本属于相手蟹科的侧足厚蟹、天津厚蟹、日本仿厚蟹和伍氏仿厚蟹没有与 2种相手蟹聚为一支, 而是最终与属于弓蟹科的2种近方蟹聚为一大支, 且有高达99%的支持率。结果表明, 4种厚蟹与2种近方蟹的亲缘关系相对较近, 而与2种相手蟹等其他方蟹的亲缘关系则相对较远。因此, 研究结果支持将4种厚蟹从相手蟹科移到弓蟹科。此外, 属于相手蟹科的 2种相手蟹聚为一支, 属于方蟹科的白纹方蟹和属于斜纹蟹科的瘤突斜纹蟹又各自成为一支; 表明 16S rRNA基因的分子数据支持其形态学分类结果的正确性, 提示上述4科蟹类可能分别为单系。
方蟹; 16S rRNA; 序列; 系统发生
方蟹是我国沿海常见的具有一定经济价值的蟹类, 隶属于甲壳纲(Crustacea)、十足目(Decapoda)、方蟹科(Grapsidae), 该科又分为方蟹亚科(Grapsinae)、弓蟹亚科(Varuninae)、相手蟹亚科(Sesarminae)和斜纹蟹亚科(Plagusiinae)[1]。近来, 一些学者根据幼体形态学和分子系统学的研究而将方蟹科提升为方蟹总科(Grapsoidea), 其中包括原方蟹科中的 4个亚科被提升为科的方蟹科(Grapsidae)、相手蟹科(Sesarmidae)、弓蟹科(Varunidae)和斜纹蟹科(Plagusiidae)以及地蟹科(Gecarcinidae)和 2001年新建立的雕刻方蟹科(Glyptograpsidae); 并将厚蟹属(Helice)等蟹类从相手蟹科移到弓蟹科[2~6]。
动物线粒体 DNA(mtDNA)因其分子量小、母系遗传、比核DNA进化速率快等特征而广泛地应用于进化生物学研究中; 而mtDNA 16S rRNA基因有较高的保守性, 易于进行 PCR引物的设计和扩增, 非常适合于种及其以上分类阶元的差异研究[7,8]。16S rRNA基因序列已被广泛用于蟹类的分子系统学研究[9~14], 已有一些学者基于16S rRNA基因序列分别对美国、日本等地的部分方蟹进行了分子系统学分析[2~4,15]。
本研究采用线粒体 16S rRNA基因作为分子标记, 对产于中国沿海的10种方蟹进行了序列测定和分子系统关系分析, 探讨它们之间的遗传差异及亲缘关系, 以期为蟹类的种质鉴定、物种保护和资源评价提供基础的分子遗传学资料, 为进一步研究蟹类分子系统学提供理论依据。
1 材料与方法
1.1 实验材料
实验所用标本(包括红螯相手蟹和褶痕相手蟹[16])的采集地点、数量及时间等信息见表1。标本浸泡于95%乙醇或保存于-40℃冰箱中备用。
1.2 DNA提取
从蟹类的步足和螯足中取约 50 mg肌肉, 采用传统法(酚/氯仿抽提法)从肌肉组织中提取基因组DNA。将乙醇沉淀后DNA溶解入40 µL超纯水, 放入4 ℃冰箱6 h, 最后-20℃保存备用。

表1 测序用蟹类取样信息Tab. 1 Crabs used in sequence analysis of mitochondrial 16S rRNA gene
1.3 PCR扩增
以 L2510 5′-CGCCTGTTTAACAAAAACAT-3′和 H3059 5′-CCGGTCTGAACTCAGATCATGT-3′为引物对16S rRNA部分片段进行PCR扩增[17]。扩增时的反应体积为 25 µL, 反应液中含 2.5 µL 10× PCR buffer, 2.0 µL dNTPs(2.5 mmol/L), 2.0 µL MgCl2(25 mmol/L), 1 µL 模板 DNA, 引物各 0.5 µL(10 µmol/L),0.2 µLTaq酶(5 U/µL), 无菌去离子水补足到 25 µL。PCR循环参数为: 94℃预变性1.5 min后, 94 ℃变性30 s, 49 ℃退火45 s, 72 ℃延伸1 min, 循环39次, 然后在72℃延伸5 min, 于4 ℃保存。
1.4 序列测定
PCR产物用含有溴化乙锭的1.0%琼脂糖凝胶电泳检测, 凝胶成像系统观察、照相。对于扩增效果良好的样品进行回收, 回收时用 1.0%琼脂糖凝胶, Ta-KaRa Agarose Gel DNA Purification Kit(宝生物工程(大连)有限公司)进行回收和纯化, 纯化产物送至上海英骏测序公司, 用ABI3730XL测序仪进行正反链双向测序。测序所用引物和PCR扩增时的引物相同。
1.5 数据分析
首先仔细核对测序胶图, 所有序列均由DNASTAR软件包(DNASTAR, Inc., Madison, USA)进行编辑、校对和比对, 并对排序结果进行分析; 所有序列为两端去引物后的序列。用DNASP软件检测变异位点数(Variable sites)、简约信息位点数(Parsimony informative sites)。用 MEGA(Version4.0)软件统计序列的碱基组成, 计算种间的遗传距离,进行系统发生和分子进化分析。所有序列之间基于Kimura-双参数距离模型估计其遗传距离; 用 NJ法(Neighbor-Joining)构建分子系统发生树,系统树各结点的支持率以序列数据集 1 000次重复抽样检验的自引导值(Bootstrap value)表示, 各分支上的数字为重抽样分析得到的大于50%的支持率。
2 结果
对表1中除红螯相手蟹和褶痕相手蟹[16]之外的9种蟹类的线粒体16S rRNA基因片段进行PCR扩增和序列测定。每种蟹类的 3个不同个体之间序列没有差异。它们的序列长度、碱基组成和GenBank登录号分别见表2。由表2可见, 10种方蟹的A、T、G、C含量只有略微的差异, 表现为与其他蟹类相同的特点[2,14], 即 A+T含量(69.8%~76.0%)明显高于G+C含量。
10种方蟹的16S rRNA基因序列比对获得541 bp的同源序列(含插入/缺失位点), 除插入/缺失位点外共检测到146个变异位点, 其中81个为简约信息位点。
利用Kimura-双参数法计算得到了10种方蟹种间的遗传距离(表3)。遗传距离显示, 2种厚蟹和2种仿厚蟹与2种近方蟹的遗传距离为0.054~0.085, 都显著的小于与其他方蟹之间的遗传距离。
以宽身大眼蟹(DLA)为外群, 基于16S rRNA基因片段序列采用NJ法构建的系统进化树的拓扑结构显示(图 1): 侧足厚蟹和天津厚蟹, 日本仿厚蟹和伍氏仿厚蟹首先各自聚到一起成为两个分支, 然后二者聚到一起成为一支, 再与弓蟹科的肉球近方蟹和绒螯近方蟹聚为一支, 支持率高达 99%; 但厚蟹在形态分类上原本属于相手蟹科。此外, 2种相手蟹聚为一支, 属于相手蟹科; 白纹方蟹和瘤突斜纹蟹各自成为一支, 分别属于方蟹科和斜纹蟹科。
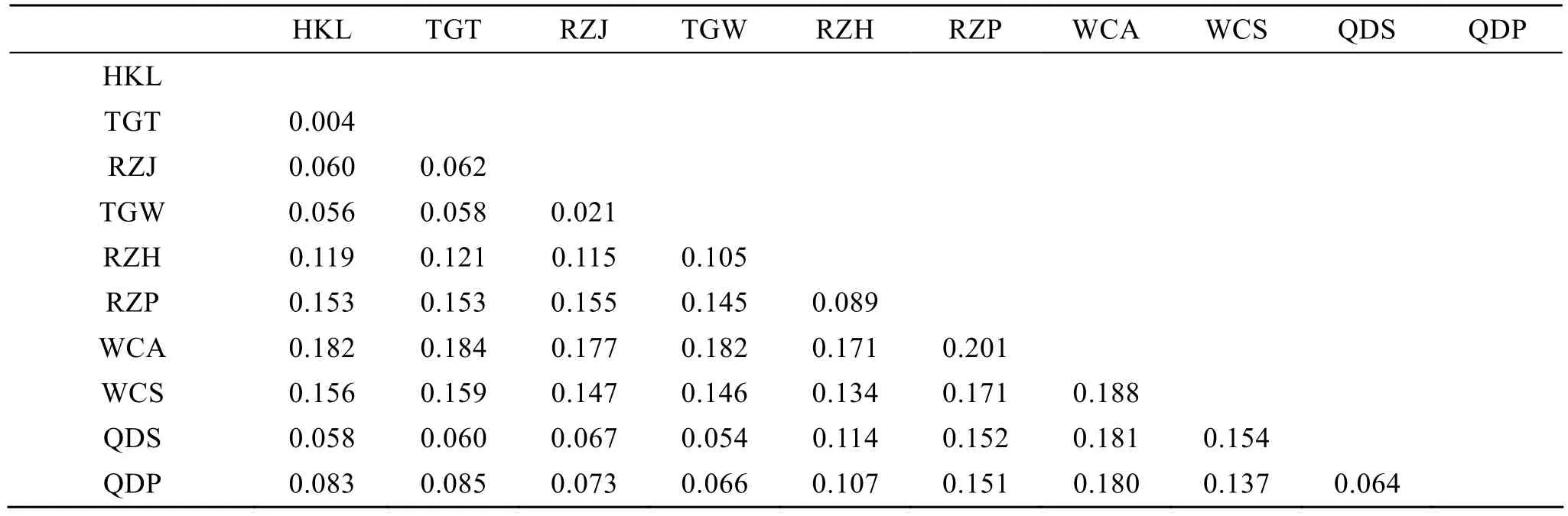
表3 10种方蟹之间的遗传距离Tab. 3 Interspecific genetic distances among ten Grapsoidea species
3 讨论
Schubart等[3]研究了Helice crassamtDNA的16S rRNA部分序列, 结果显示其与弓蟹科蟹类的亲缘关系最近, 建议将其从相手蟹科移到弓蟹科; Kitaura等[4]研究了三齿厚蟹(H. tridens)mtDNA的16S rRNA全序列, 孙红英等[5]研究了天津厚蟹(Helice tientsinensis)mtDNA的 16S rRNA部分序列, 以及Schubart 等[15]研究了短螯厚蟹(H. leachii)mtDNA的12S rRNA和16S rRNA部分序列, 研究结果分别支持将天津厚蟹、三齿厚蟹和短螯厚蟹从相手蟹科移到弓蟹科。

图1 10种方蟹及外群16S rRNA基因序列NJ系统树Fig. 1 Neighbor-Joining tree for 16S rRNA gene of ten Grapsoidea species and outgroup
上述研究均仅通过对某一种厚蟹的研究来探讨厚蟹的分类地位。而本研究的所有4种厚蟹与2种近方蟹的遗传距离(0.054~0.085)都显著的小于与其他方蟹之间的遗传距离, 特别是 4种厚蟹与原本属于同一相手蟹科的 2种相手蟹之间的遗传距离达到了0.105~0.155(表3); 而基于16S rRNA基因片段序列采用 NJ法构建的系统进化树的拓扑结构(图 1)也显示, 原本属于相手蟹科的侧足厚蟹、天津厚蟹、日本仿厚蟹和伍氏仿厚蟹没有与2种相手蟹聚为一支,而是最终与属于弓蟹科的 2种近方蟹聚为一大支,且有高达 99%的支持率。此结果表明 4种厚蟹与 2种近方蟹的亲缘关系相对较近, 而与 2种相手蟹等其他方蟹的亲缘关系则相对较远。因此, 本研究结果支持将 4种厚蟹从相手蟹科移到弓蟹科, 从而进一步明确了厚蟹的分类地位。从形态上来看, 厚蟹等蟹类的雄性生殖孔的位置与弓蟹科蟹类胸孔的位置相一致; 而厚蟹等蟹类从 状幼体到大眼幼体的形态特征亦与弓蟹科蟹类相一致[3]。因此, 形态学的有关特征亦支持将厚蟹从相手蟹科移到弓蟹科。
此外, 属于相手蟹科的2种相手蟹聚为一支, 属于方蟹科的白纹方蟹和属于斜纹蟹科的瘤突斜纹蟹又各自成为一支; 表明16S rRNA基因的分子数据支持其形态学分类结果的正确性[6], 提示上述4科蟹类可能分别为单系, 但这还有待于对这些科的更多蟹类进行相关研究后才能确定。
[1] 戴爱云, 杨思谅, 宋玉枝, 等. 中国海洋蟹类[M]. 北京: 海洋出版社, 1986. 12-514.
[2] Schubart C D, Cuesta J A, Diesel R. Molecular phylogeny, taxonomy, and evolution of nonmarine lineages within the American Grapsoid crabs (Crustacea:Brachyura) [J]. Molecular Phylogenetics and Evolution, 2000, 15(2): 179-190.
[3] Schubart C D, Cuesta J A, Felder D L. Glyptograpsidae,a new brachyuran family from Central America: larval and adult morphology and a molecular phylogeny of the Grapsoidea[J]. Journal of Crustacean Biology,2002, 22(1): 28-44.
[4] Kitaura J, Wada K, Nishida M. Molecular phylogeny of grapsoid and ocypodoid crabs with special reference to the generaMetaplaxandMacrophthalmus[J]. Journal of Crustacean Biology, 2002, 22(3): 682-693.
[5] 孙红英, 周开亚, 景开颜, 等. 从线粒体 16S rDNA部分序列探讨厚蟹属的系统学位置[J]. 南京师大学报(自然科学版), 2002, 25(1): 15-19.
[6] Ng P K L, Guinot D, Davie P J F. Systema brachyurorum: partⅠ . An annotated checklist of extant brachyuran crabs of the world[J]. The Raffles Bulletin of Zoology (Supplement), 2008, 17: 1-286.
[7] 徐敬明. 蟹类线粒体 DNA的研究与应用[J]. 中国海洋大学学报(自然科学版), 2006, 36(6): 879-884.
[8] 吕国庆, 李思发. 鱼类线粒体 DNA多态研究和应用进展[J]. 中国水产科学, 1998, 5(3): 94-103.
[9] Schubart C D, Diesel R, Hedges S B. Rapid evolution to terrestrial life in Jamaican crabs[J]. Nature, 1998,393: 363-365.
[10] Schubart C D, Neigel J E, Felder D L. The use of the mitochondrial 16S rRNA gene for phylogenetic and biogeographic studies of Crustacea[J]. Crustacean Issues, 2000, 12: 817-830.
[11] 邱高峰, 徐巧婷, 王丽卿, 等. 四种绒螯蟹分子分类与系统发育[J]. 动物学报, 2001,47(6):640-647.
[12] Weinberg J R, Dahlgren N D, Halanych K M. Genetic differences within and between species of deep-sea crabs (Chaceon)from the North Atlantic Ocean[J].Biological Bulletin, 2003, 204: 318-326.
[13] 孙红英, 周开亚, 杨小军. 从线粒体 16S rDNA序列探讨绒螯蟹类的系统发生关系[J]. 动物学报, 2003,49(5): 592-599.
[14] Harrison J S. Evolution, biogeography, and the utility of mitochondrial 16S and COI genes in phylogenetic analysis of the crab genus Austinixa (Decapoda: Pinnotheridae)[J]. Molecular Phylogenetics and Evolution, 2004, 30: 743-754.
[15] Schubart C D, Cannicci S, Vannini M, et al. Molecular phylogeny of grapsoid crabs (Decapoda, Brachyura)and allies based on two mitochondrial genes and a proposal for refraining from current superfamily classification[J]. Journal of Zoological Systematics and Evolutionary Research, 2006, 44 (3): 193-199.
[16] 徐敬明, 张俊丽, 方华华, 等. 相手蟹属两种蟹类线粒体16S rRNA基因序列的比较[J]. 水产科学, 2006,25(9): 443-447.
[17] Bouchon D, Souty-grosset C, Raimond R. Mitochondrial DNA variation and markers of species identity in two penaeid shrimp species: Penaeus monodnn Fabricius and P. japonicus Bate[J]. Aquiculture, 1994, 127:131-144.
Molecular phylogeny of grapsoid crabs (Crustacea, Decapoda)based on partial sequences of mitochondrial 16S rRNA gene from China
XU Jing-ming
(College of Life Science and Technology, Chongqing University of Arts and Sciences, Chongqing 402168,China)
Jan., 22, 2010
Grapsoid crabs; 16S rRNA; Sequence; Phylogeny
Partial sequences of mitochondrial 16S rRNA gene of ten species crabs of Grapsoidea from the coast of China were determined and subjected to phylogenetic analysis. The lengths of sequences were from 517 to 533bp.The A, T, G and C contents of them were similar, and AT contents(69.8%~76.0%)were higher than GC contents.Furthermore, the 541bp homologous segments were analyzed. The results showed that there were 146 variable sites and 81 parsimony-information sites in the nucleotides. The genetic distances between four species, Helice latimera,H. tientsinensis, Helicana japonica and H. wuana, and two species, Hemigrapsus sanguineus and H. penicillatus,were from 0.054 to 0.085, which were much smaller than that (0.105~0.155) between the four species of Helice and Helicana and two species of Chiromantes haematocheir and Parasesarma plicatum. However, the four species of Helice and Helicana and the two species of C. haematocheir and P. plicatum had been believed to belong to family Sesarmidae. Topological structure of the molecular phylogenetic tree constructed by 541bp homologous segments with Neighbor-Joining method showed that the four species of Helice and Helicana eventually were clustered into a distinct clade (99% confidence level) with the two species of H. sanguineus and H. penicillatus. But, the two species of Hemigrapsus belonged to family Varunidae. Therefore, the results supported that the four species of Helice and Helicana were transferred from Sesarmidae to Varunidae. In addition, the two species of C. haematocheir and P. plicatum that belong to family Sesarmidae were clustered into a distinct clade, and two other distinct clades were formed by Grapsus albolineatus and Plagusia squamosa that belong to families Grapsidae and Plagusiidae respectively. The results revealed that Grapsidae, Sesarmidae, Varunidae and Plagusiidae should be monophyletic, respectively.
Q178.53;Q953
A
1000-3096(2010)10-0013-05
2010-01-22;
2010-05-19
重庆文理学院引进人才专项(200803)
徐敬明(1963-), 男, 山东日照人, 博士, 教授, 研究方向:动物分子与生理生态, E-mail: xjingming@163.com
(本文编辑:梁德海)

