海蜇养殖群体及自然捕获群体ITS序列遗传分析
孙国华, 刘相全, 杨建敏, 张锡佳, 刘爱英, 谭福奕
(1. 山东省海洋水产研究所, 山东 烟台 264006; 2. 山东好当家海洋发展股份有限公司, 山东 威海264305)
海蜇养殖群体及自然捕获群体ITS序列遗传分析
孙国华1,2, 刘相全1, 杨建敏1, 张锡佳1, 刘爱英1, 谭福奕2
(1. 山东省海洋水产研究所, 山东 烟台 264006; 2. 山东好当家海洋发展股份有限公司, 山东 威海264305)
为调查海蜇( Rhopilema esculentum)自然海区群体和养殖群体遗传多样性, 对烟台莱州湾和江苏海州湾自然海区捕获群体及威海养殖群体 24个个体的 ITS序列进行了 PCR扩增和序列分析, 获得
DNA片段长度在1 080~1 096 bp之间, 包括完整的ITS1, 5.8S和完整ITS2序列。结果表明, 在所测碱基序列中共发现26个变异位点, 4处多碱基插入位置, 29个插入缺失位点。群体内平均核苷酸差异数K和平均核苷酸多样性指数Pi为2.179~2.750和0.002 02~0.002 54, 群体间平均核苷酸差异数K值波动在2.797~3.031之间, 遗传距离在0.002 30~0.002 81之间, 遗传分化指数(Fst)值在0.032 50~0.730 8之间, 群体内及群体间遗传多样性指标数值比较相近, 群体间的遗传距离比较小, 群体遗传结构相似,群体间没有明显的遗传分化。
海蜇( Rhopilema esculentum); 转录间隔区(ITS); 遗传多样性; 遗传结构
海蜇(Rhopilema esculentum)是腔肠动物门、钵水母纲、根口水母目、根口水母科、海蜇属的大型食用水母, 为暖水性河口种类, 分布于北自辽宁南至海南岛近岸海域, 资源丰富, 经济价值大, 是我国重要渔业种类之一[1]。海蜇营养价值高, 含有丰富的蛋白质和脂肪酸[2,3], 是深受人民喜爱的水产品; 并且海蜇头、皮、腹面黑膜等部位均可入药, 具有清热解毒、化痰软坚、降压消肿等药用价值[4,5]。海蜇以浮游动物为食, 食物链短, 种群生命周期短, 资源数量年间波动大, 加之近几年来海蜇价格逐年增高, 捕捞过度, 资源受到严重破坏[6,7]。海蜇池塘养殖业及增殖放流工作近几年得到重视和发展, 但是海蜇分子生物学方面的基础研究甚少, 有关群体遗传多样性研究尚比较欠缺。
核糖体基因转录间隔区(internal transcribed spacer ITS), 是位于核糖体DNA(rDNA)上18S和28S基因之间的区域片段, 主要包括内转录间隔区 1(ITS1)、5.8S rDNA、内转录间隔区2(ITS2) , 其两侧分别是18 S RNA基因和28 S RNA基因。与核糖体DNA中的 18S、5.8S和 28S的基因组序列相比较,ITS1和ITS2作为非编码区, 承受的进化选择压力较小, 相对变化较大[8], ITS区段的扩增已普遍用来作为分类鉴定及分析近缘种和种群的系统发育关系的方法[9,10], 在水产动物中也有广泛的应用[11~13]。
本研究利用 ITS序列多态性对两个自然海区捕获海蜇群体和一个人工养殖海蜇群体进行了遗传结构分析, 以期从分子水平上了解海蜇资源的遗传背景, 为海蜇种质资源保护及海蜇遗传育种提供依据。
1 材料和方法
1.1 实验材料
海蜇养殖群体于2008年7月取自山东好当家集团有限公司, 平均伞径 420 mm; 江苏野生群体于2008年8月取自海洲湾海域, 平均伞径330 mm; 烟台野生群体于2008年8月取自莱州湾海域, 平均伞径360 mm。所有样品取伞缘部组织冻存于-20℃。
1.2 DNA提取
样品100 mg机械剪碎, 加入700 mL CTAB提取缓冲液(100 mmol/L Tris-HCl pH8.0, 20 mmol/L EDTA-Na2, 1.4 mol/LNaCl, 2% CTAB, 0.1%β-巯基乙醇)和终浓度为100 µg/mL蛋白酶K, 55℃消化3 h或37℃过夜, 等体积酚氯仿、氯仿抽提, 2倍体积乙醇沉淀, TE溶解, 4℃保存。
1.3 ITS区片段扩增、纯化与克隆测序
ITS序列扩增使用 Heath等[14]设计的通用引物,引 物 序 列 为 : ITSF1(5′-GGTTTCCGTAGGTGAAC-CTGCGGAAGGATC-3′)和 ITSR1(5′-GCTTTGGGCT GCAGT CCCAA GCAA CCCACTC-3′)。一次扩增整个ITS区, 包括ITS1、5.8S rRNA和ITS2。PCR反应条件如下: 25 µL PCR反应体系: 包含约80 ng的基因组DNA, 10 pmol的引物, 2.5 µmolMgCl2, 0.2 µmol dNTP,Taq DNA聚合酶1 U。PCR反应条件为: 94℃ 4 min;94℃40 s, 退火温度55℃4 0 s, 72℃延长40 s, 30个循环;72℃ 10 min; 4℃保温, 每次均设空白对照。PCR产物1%琼脂糖凝胶电泳检测, 凝胶回收, PCR产物连接到pMD18T, 然后转化到E.coliDH5α细胞, 37℃培养, 涂平板, 挑克隆, 载体引物检测, 双向测序。
1.4 序列分析用MEGA 4.0进行同源排序比对, 确定序列长度;计算不同序列间的碱基组成、变异位点、简约信息位点和不同地理种群的 Kimura 2-paramter遗传距离。采用Kimura 2-paramter距离矩阵采用邻接法(NJ)构建单倍型分子系统树, 系统树中节点的自举置信水平应用自引导(bootstrap)估计, 共 1 000次循环。用DNASP 4.50软件计算各个群体的单倍型, 单倍型多态性, 多态位点数, 平均核苷酸差异数, 核苷酸多样性指数, 基因流Nm等。用ARLEQUIN 3.11中的分子变异分析(AMOVA)分析方法估算遗传变异在群体内和群体间的分布及遗传分化系数(F-statistics,Fst), 计算并用排列测验法(permutation test)检验Fst的显著性(重复次数为1000)。
2 结果
2.1 扩增序列长度、结构及碱基组成
PCR扩增3个群体各8个个体共24条序列, 所得测序序列去载体及引物序列, 经Clustal对位排序,ITS序列长度在 1 080~1 096 bp之间, 包括完整的ITS1, 5.8S和完整ITS2序列, 图1表示单倍型1碱基序列, 碱基 A、G、T、C和(A+T)含量分别 23.97%,25.56%, 25.98%, 24.42%和49.95%。同源片段序列对位排序和分析后, 在所测碱基序列中共发现26个变异位点, 这些变异位点包括19个转换位点(其中T/C 12处, A/G 7处)和7个颠换位点(其中A/T 4处, G/T 2处, A/C1处, G/C1处), 其中简约信息位点2个, 4处多碱基插入位置29个插入缺失位点, 插入序列分别为GTCGTTCG, AACAA, GAG, TGTC/TGTCTGTC。

图1 海蜇转录间隔区(ITS)核苷酸序列Fig. 1 ITS nucleotide sequences of Rhopilema esculentum
2.2 三群体ITS序列的多态性分析
不计入插入缺失位点, 26个变异位点定义了17个单倍型, 在 17种单倍型中, 群体间共享单倍型有2个, 占单倍型总数的11.8%。单倍型2分别为烟台,江苏和养殖群体共有, 单倍型 12分别为烟台, 江苏群体所共有, 其他单倍型仅为某一个体独有(表 1)。仅以29个插入缺失位点计算, 24条序列归为9个单倍型, 共享单倍型5个, 其中单倍型Ⅱ和单倍型Ⅳ为 烟台、江苏和养殖3个群体所共有(表2)。

表1 海蜇3个群体ITS序列变异位点及定义单倍型Tab. 1 Variable nucleotide positions in ITS region of 17 haplotypes and number of individuals of each haplotype found in each locality

表2 海蜇3个群体ITS插入缺失位点及定义单倍型Tab. 2 Insertion-Deletion polymorphysm and the number of each haplotype in Rhopilema esculentum populations
2.3 海蜇各群体内及群体间遗传多样性分析
用DnaSP4.0软件对海蜇各群体的遗传多样性参数进行计算, 结果列入表3。数据显示, 在3个群体中, 烟台和江苏自然捕获群体的单倍型(Nhap)要多于养殖群体, 3个群体中江苏自然捕获群体的碱基多态位点(S)比例是最高的。在平均核苷酸差异数K和平均核苷酸多样性指数Pi这两个指标上由ITS基因片段核苷酸序列反映出来的群体遗传多样性指数的绝对值水平不高, 3个群体数值比较相近, 江苏自然捕获群体遗传多样性相对而言比较高, 平均核苷酸差异数K和平均核苷酸多样性指数Pi分别为 3.214和 0.004 29。
各群体之间的相关遗传参数进行计算结果列入表 4。群体间平均核苷酸差异数K值波动在2.797~3.031之间, 群体间平均每位点核苷酸替代数(Dxy)与居群间每位点净核苷酸替代数(Da)的值相应也不是很高。K值、Dxy值和Da值的数据显示海蜇3个群体间遗传多样性指标的差异不大。

表3 海蜇各群体内的遗传多样性参数Tab. 3 Summary of genetic diversity of different Rhopilema esculentum populations

表4 海蜇各群体间遗传多样性参数及遗传分化系数Tab. 4 Summary of genetic diversity between different Rhopilema esculentum populations and Fst
2.4 海蜇 3个群体间遗传距离及群体遗传分化
海蜇3个群体间遗传距离在0.002 30~0.002 81之间, 遗传差异AMOVA分析结果显示, 3个群体间两两比较的遗传分化指数(Fst)值在 0.032 50~0.730 8之间(表4), 三群体间遗传分化系数Fst=0.058 02, 表明在整个遗传变异中群体间占 5.802%, 其余的遗传变异来源于群体内, 群体内具有程度较高的遗传分化(表5)。
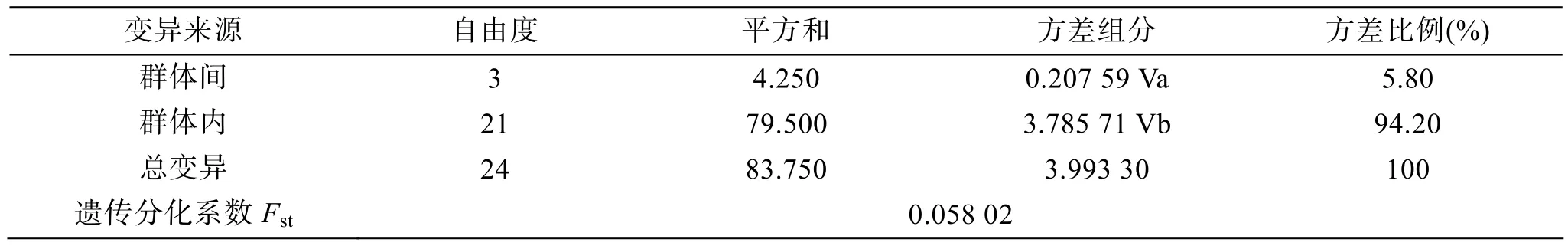
表5 海蜇各群体间遗传差异的分子方差分析表(AMOVA)Tab. 5 Analysis of molecular variance (AMOVA) among populations of Rhopilema esculentum
2.5 分子系统树的构建
根据所得ITS序列, 以Nemopilema nomurai作为外群, 利用MEGA4.0用NJ法构建17个单倍型个体间的分子系统树, 自展一致树显示作为外群的Nemopilema nomurai独立于海蜇 17个单倍型之外,但是一个群体的不同个体并没有先聚为一起, 分属于3个不同群体的17个单倍型没有明显的聚类, 不同群体之个体间的亲缘关系没有明显分群(图2)。
3 讨论
作者对两个自然捕获群体和一个养殖群体24个海蜇个体的核糖体转录间隔区进行了扩增、测序, 得到长度为1 080~1 096 bp的序列, 其中包括完整的ITS1, 5.8S和ITS2序列。在所获得序列结构中值得注意的一点是, 序列中简单重复单元较多, 序列中存在的插入缺失位点均由简单重复单元数目改变引起, 如在第一个位点(C/TTCG)重复数目为 5~7个。简单重复单元数目改变的原因推测是在复制时发生滑动突变, 使其拷贝数发生改变, 进而引起序列的变异。在海蜇的ITS序列中, 共发现4处简单重复序列的插入和缺失, 至于什么原因促成这种简单重复序列的插入和缺失, 以及这种插入和缺失的调控机理, 目前尚未见相关研究报道。其形成是否与适应生长环境及表型性状相关, 此方面还需更深入的研究探讨。

图2 海蜇ITS序列17个单倍型NJ聚类图Fig. 2 Phylogenic tree of 17 haplotypes based on ITS sequences of Rhopilema esculentumYT. 烟台自然捕获群体; JS. 江苏自然捕获群体; YZ. 威海养殖群体YT. wild population of Yantai; JS. wild population of Jiangsu; YZ.cultured population
许多海洋经济种类养殖基本是依靠采捕野生亲本进行大量繁殖, 采用这种方式繁育, 由于从自然群体中选用亲本数目有限, 势必造成一定程度的随机漂变, 引起养殖群体的遗传结构发生改变, 总体遗传多样性下降[15,16], 多年的累代养殖将造成不同程度的近交繁殖, 引起种质退化。同时因过度捕捞使野生资源逐渐减少, 而养殖个体不断逃逸到自然水体, 也将对野生群体的遗传本质、种质资源和遗传多样性产生不可低估的影响[17]。海蜇发展养殖开始的比较晚, 发展十几年来也是依靠从自然海域捕获亲本进行育苗, 因此, 随着养殖规模的不断扩大, 研究海蜇野生种群和养殖种群的遗传结构以及遗传多样性对开展海蜇自然资源调查与保护使之可持续利用;开发种质资源, 进行优良品种培育, 最大限度地保持养殖群体的遗传多样性具有重要意义。
本研究中两个自然捕获群体和一个养殖群体由分析 ITS序列相关参数所反映出来的三群体群体内遗传多样性参数差异不大, 江苏群体略高; 两两群体间遗传多样性指标数值也比较相近, 群体间遗传距离在0.002 30~0.002 81之间, 遗传分化指数(Fst)值在0.032 50~0.730 8之间。根据Wright[18]关于遗传分化指数的大小和分化程度的解释, 当Fst接近于 0时, 说明群体间的没有发生遗传分化,Fst值在0.05~0.15之间, 遗传分化达到中等水平。而本研究中 3个群体间遗传分化指数均比较小, 说明大群体之间几乎没有遗传分化。相应的, 分属 3个群体的17单倍型在分子系统树中也没有明显的聚类。目前,山东沿海地区海蜇的育苗捕获亲本基本是来自于江苏附近海域, 而同时, 由于海蜇自然野生资源的衰退, 山东省近几年一直在开展海蜇的增殖放流工作,这种情况, 从海蜇苗种溯源上解释了本研究的结果。
作者分析得到海蜇 ITS片段序列多态数据, 群体遗传结构分析表明, 群体遗传多样性比较低, 群体间的遗传距离比较小, 群体遗传结构相似, 群体间没有明显的遗传分化, 说明养殖群体和两个自然捕获群体尚没有形成自己独立的遗传结构。从海蜇遗传育种种质资源角度来看, 再捕获不同烟台和江苏自然群体作为亲本对繁育后代影响不大; 养殖群体由于并不是累代繁育, 其遗传结构与自然捕获群体也无很大区别。在未来养殖过程中, 为了防止人工选育中的近交衰退的问题, 除了注意保持选育亲本的数量外, 还应注意利用分子生物学手段对选育群体的遗传多样性的检测分析, 在保持选育性状稳定性的同时, 最大限度地保留群体的遗传多样性, 使养殖产业健康、高效地持续发展。本研究结果同时也说明, 自然捕获群体遗传结构相似, 黄渤海海蜇种质资源遗传多样性比较低, 应加大对海蜇自然资源的保护力度, 再继续开展海蜇的增殖放流工作的同时, 更重要的是控制自然海区海蜇资源的捕捞量,保护海蜇自然种质资源, 维护黄渤海生态平衡, 保证其自然资源可持续利用。
[1] 高尚武, 洪慧馨, 张士美.中国动物志: 无脊椎动物刺细胞动物门(第二十七卷): 水螅虫纲钵水母纲[M].北京: 科学出版社, 2003.222-225.
[2] Hsieh Y H P, Leong F M, Rudloe J. Jellyfish as food [J].Hydrobiogia, 2001, 451: 11-17.
[3] 刘希光, 于华华, 赵增芹, 等.海蜇不同部位脂肪酸的组成研究[J].分析化学, 2004, 32(12): 1 635-1 638.
[4] 罗元凯.食用药物和药膳[J].新中医, 1996, 28(12):8-9.
[5] 计光辅.海蜇的药用功效[J].家庭中医药, 2007, 14(7):68.
[6] 王明德, 谢勇.中国渤海海蜇资源的保护和利用[J].自然资源, 1996, 1: 64-66.
[7] 关松, 张鹏刚, 刘春洋, 等.我省海蜇池塘养殖现状与存在问题[J].水产科学, 2004, 23(8): 30-31.
[8] Hillis D M, Dixon M T. Ribosomal DNA: molecular evolution and phylogenetic inference [J]. Q Rev Biol,1991, 66: 411-453.
[9] 周伯平, 周开亚, 宋大祥.核r DNA ITS区序列在无脊椎动物分子系统学研究中的应用[J].动物学杂志,2002, 37(4): 67-73 .
[10] 汪文俊, 王飞久, 陈松林, 等.浒苔 ITS区的扩增和分析[J].海洋水产研究, 2008, 29(5): 124-129.
[11] Coleman A W, Vacquier V D. Exploring the phylogenetic utility of ITS sequences for animals: A test case for abalone (Haliotis) [J]. J Mol Evol, 2002, 54:246-257.
[12] 丁小雷, 何毛贤, 邓凤姣, 等.双壳纲动物核糖体RNA 18S -ITS1序列及其在分子系统发育研究中的应用[J].遗传, 2004, 26(3): 319-324.
[13] 陈琳琳, 孔晓瑜, 周立石.魁蚶核糖体 DNA基因转录间隔区的序列特征魁蚶核糖体DNA基因转录间隔区的序列特征[J].中国水产科学, 2005, 12(1):104-108.
[14] Heath D D, Rawson P D, Hilbish T J. PCR-based nuclear markers identify alien blue mussel (Mytilus spp.)genotypes on the west coast of Canada [J]. Can J Fish Aquat Sci, 1995, 52 (12): 2621-2627.
[15] Sunden S L F, Davis S K. Evaluation of genetic variation in a domestic population of Penaeus vannamei(Bonne): A comparison whit three natural population[J]. Aquaculture, 1991, 151:131-142.
[16] Wolfus G M, Garcia D K, Alcivar-Warren A. Application of the microsatellite technique for analysing genetic diversity breeding programs [J]. Aquaculture,1997, 152:35-47.
[17] Allendorf F W, Phelps S R. Loss of genetic variation in a hatchery stock of cutthroat trout [J]. Trans Am Fish Soc, 1980, 109: 537-543.
[18] Wright S. The genetical structure of population [J].Annals of Eugenics, 1951, 15: 323-334.
Genetic diversity of ITS sequences in farmed and natural Rhopilema esculentum populations
SUN Guo-hua1,2, LIU Xiang-quan1, YANG Jian-min1, ZHANG Xi-jia1, LIU Ai-ying1,TAN Fu-yi2
(1. Marine Fisheries Research Institute of Shandong Provience, Yantai 264006, China; 2. Shandong Homey Aquatic Development Co.LTD, Weihai 264305, China)
Feb., 25, 2010
Rhopilema esculentum, internal transcribed spacers(ITS), population diversity, genetic structure
For the purpose of investigating genetic diverstiy in wild and cultured populitions of Rhopilema esculentum, ITS regions of 24 individuals within Yantai and Jiangsu wild populitions and Weihai cultured population were amplified and analyzed. The whole sequences were between 1 080~1 096 bp, including ITS1-5.8S-ITS2. The result showed that 26 variable nucleotide positions were detected and four nucleotide sites had 29 insertion-deletion positions. Indexes of Piand K among three populations were 2.179~2.750 and 0.002 02~0.002 54, respectively. Indexes of K, genetic distance and fixation indices (Fst) between populations were 2.797~3.031, 0.002 30~0.002 81 and 0.032 50~0.730 8, respectively. The indexs of genetic diversity among and between populations were closed and genetic distances between populations were small, which indicated that the genetic structure of populations were smilar and there were no distinct genetic differentiation between populations.
Q347
A
1000-3096(2010)10-0090-06
2010-02-25;
2010-05-12
国家海洋局项目(海科字[2006]16号)
孙国华(1979-), 女, 山东莱西人, 博士, 助理研究员,主要从事海洋生物分子遗传研究, E-mail: sghqingdao@hotmail.com;杨建敏, 通信作者, E-mail: ladderp@126.com
(本文编辑:梁德海)

