细菌降解萘、菲的代谢途径及相关基因的研究进展
张丹,李兆格,包新光,李江波,梁海华,段康民,沈立新
西北大学生命科学学院 西部资源与现代生物技术省部共建教育部重点实验室,西安 710069
综 述
细菌降解萘、菲的代谢途径及相关基因的研究进展
张丹,李兆格,包新光,李江波,梁海华,段康民,沈立新
西北大学生命科学学院 西部资源与现代生物技术省部共建教育部重点实验室,西安 710069
多环芳烃 (Polycyclic aromatic hydrocarbons,PAHs) 是一类在环境中广泛存在的具有毒性的污染物,微生物降解是其在自然界中降解的主要途径,因而尤为重要。随着研究的深入,关于微生物降解PAHs的分子降解机制、途径等的认识逐渐积累。以下对细菌降解萘、菲的研究进展进行了概述,介绍了萘的水杨酸降解途径,菲的水杨酸、邻苯二甲酸及其他降解途径,同时也包括降解过程中涉及的降解基因簇,如nah-like、phn、phd、nid和nag等以及细菌在PAHs胁迫条件下其他相关基因的表达与调节等方面的最新进展。这些进展可为降解菌株的分子及遗传机制研究提供理论依据,将促进通过基因工程优化降解菌、更有效地检测PAHs环境污染及实现PAHs污染的生物修复。
多环芳烃,微生物降解,降解途径,降解基因
Abstract:Polycyclic aromatic hydrocarbons (PAHs) are toxic pollutants that exist extensively in the environment. Microbial degradation is the main pathway of PAHs eradication in natural environment and therefore is of importance to investigate.Advancement has been made in recent years regarding the PAHs molecular degradation mechanisms in bacteria. In this review, we summarized some of the research progresses in microbial PAHs biodegradation pathways (including salicylate pathway and protocatechuate pathway), key enzymes (nah-like,phn,phd,nidandnag) and genes involved. Emphasis was given on naphthalene and phenanthrene which were often used as the representatives of PAHs. It is likely that the new information will promote further research and applications of microbial PAHs biodegradation technology.
Keywords:polycyclic aromatic hydrocarbons (PAHs), microbial degradation, degradation pathway, degradation gene
多环芳烃 (PAHs) 是一类由两个或多个芳香环构成的广泛分布于自然界中的有机污染化合物,尤其常见于煤和石油加工场所排放的废气、污水中。这类物质毒性大,多数致癌、致突变,降解困难。一般来说,随着PAHs苯环数量的增加,其降解速率降低。PAHs已被多个国家列为优先控制的环境污染物。
在 PAHs的各种降解方法中,微生物降解是其主要降解途径,一直受到各国科学家的高度重视。可降解 PAHs的微生物包括细菌、真菌和藻类,其中细菌在大多数环境中发挥主要作用[1]。近年来分离到的 PAHs降解细菌主要包括假单胞菌属、芽孢杆菌属、分枝杆菌属、鞘氨醇单胞菌属、节杆菌属、伯克氏菌属和黄杆菌属等[2]。
目前人们的研究大多集中在 PAHs微生物降解菌株的筛选、降解性能测定等方面。如 Seo从石油污染土壤中分离鉴定出19株降解菌株,并测定了这些菌的降解范围和能力[3]。随着研究的深入,对微生物降解 PAHs的分子机制、途径的认识也逐渐加深。如Kasai对解环菌Cycloclasticussp. A5降解基因的研究中发现了一个包含10个开放阅读框、命名为phn的基因簇[4]。Kim报道在分枝杆菌Mycobacterium vanbaaleniiPYR-1的6.5 Mb基因组中有194个基因可能与降解有关,通过基因定位与分析探讨了该菌株降解特性的分子基础[5]。
本文在实验室前期研究的基础上,结合前人对PAHs生物降解的代谢途径及某些关键降解酶的研究,就细菌对萘、菲的代谢途径及降解过程中相关基因方面的研究进行了综述,有助于揭示 PAHs降解菌株的分子及遗传机制,为检测 PAHs环境污染及其环境生物修复提供了理论依据。
1 萘、菲在细菌中的代谢途径
1.1 细菌对萘的降解
萘是分子量最小的PAHs,由于其结构简单、较高水溶性和容易从自然界分离得到其降解菌株等原因,常被用作PAHs生物降解的模式化合物[2,6-7]。目前已知萘分解代谢的基本途径为:萘首先代谢为水杨酸,再转化为邻苯二酚,随后进入三羧酸循环(TCA) 进一步氧化开环彻底降解为二氧化碳和水,实现完全降解。
在由萘转化为水杨酸的上游代谢途径中,萘在萘双加氧酶作用下转化为顺-萘双氢二醇,随后被顺-萘双氢二醇脱氢酶催化成l,2-双羟萘,接着被l,2-双羟萘双加氧酶转化为 2-羟-2H-苯并吡喃-2-羧酸,在2-羟-2H-苯并吡喃-2-羧基异构酶作用下再生成顺-o-羟基-苯亚甲基-丙酮酸,之后被顺-o-羟基苯脱萘丙酮酸水合-醛缩酶催化为水杨醛,水杨醛在水杨醛脱氢酶催化作用下生成水杨酸。
单环产物水杨酸通过水杨酸羟化酶转化为邻苯二酚,邻苯二酚在间位裂解酶邻苯二酚-2,3-双加氧酶的作用下开环形成己二烯半醛酸,最后形成乙醛和丙酮酸,或者在邻位裂解酶邻苯二酚-1,2-双加氧酶的作用下开环生成己二烯二酸,最后形成琥珀酸和乙酰辅酶A,这些中间产物进入TCA循环后最终形成二氧化碳和水[8]。水杨酸也可以通过羟化酶转化为龙胆酸进一步降解[2]。以假单胞菌属细菌为例,总结萘的降解途径 (图1)[2]。
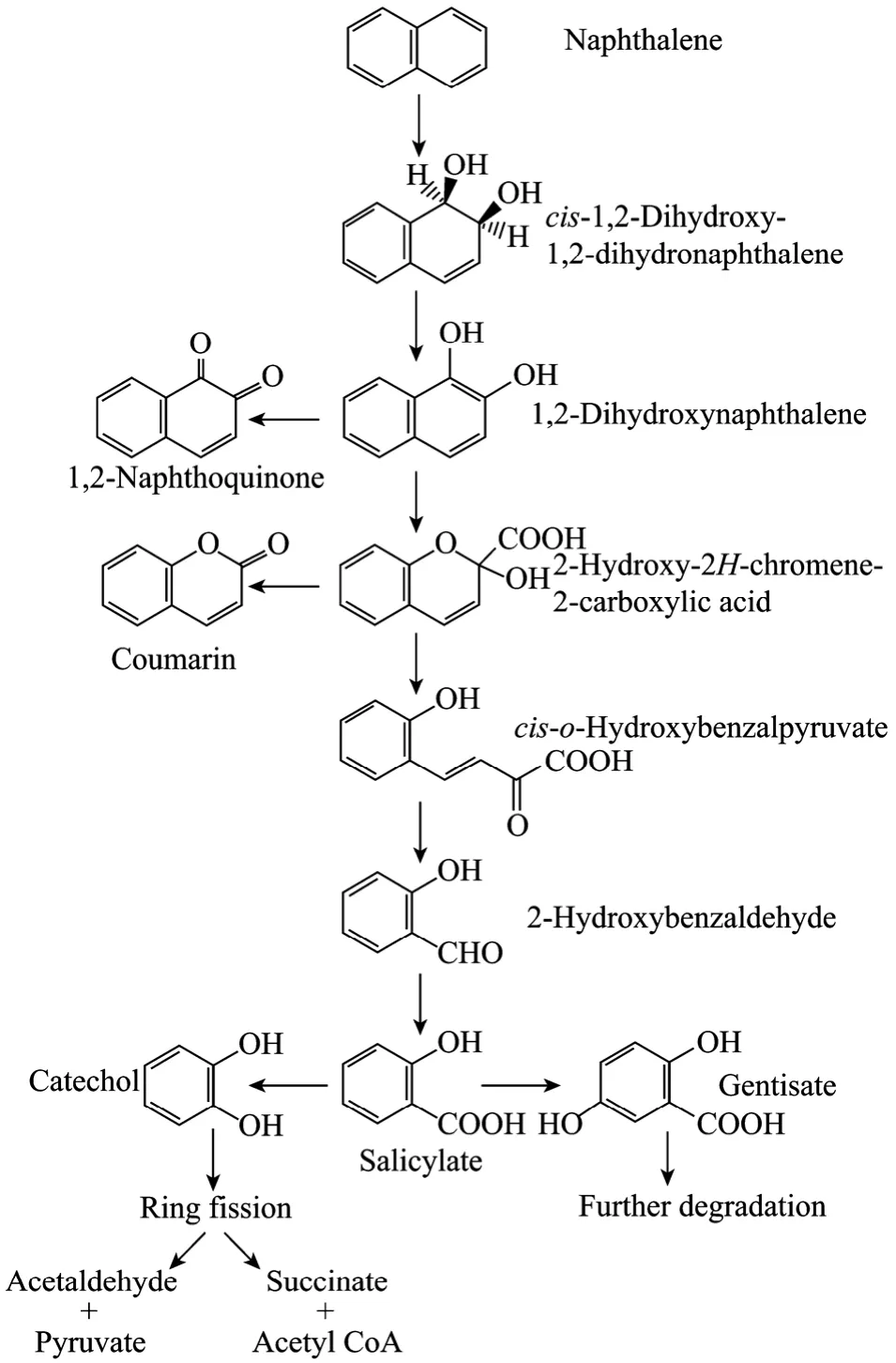
图1 推测的假单胞菌属细菌中萘的降解途径[2]Fig.1 Proposed metabolic pathways of naphthalene inPseudomonasssp.[2].
1.2 细菌对菲的降解
菲由3个苯环成一定角度联结而成,见图2[1]。菲本身没有明显的致癌性, 但因兼具K区和湾区结构,是致癌 PAHs的最小结构单元,其衍生物大都具有微弱或中等强度的致癌性,也常用作研究PAHs生物降解的模式化合物[2]。

图2 菲的化学结构图[1]Fig.2 Chemical structure of phenanthrene[1].
以假单胞菌和粪产碱菌为例,总结菲常见的降解途径 (图3)[9-10]。首先菲在3、4位C处被双加氧酶羟基化,随后依次利用顺-3,4-二氢二羟基菲脱氢酶、3,4-双羟基菲双加氧酶、2-羟基-2H-苯芘[h]色原烯-2-羧酸盐异构酶、水合-醛缩酶和 1-羟-2-萘甲醛脱氢酶转化成1-羟-2-萘甲酸,随后根据降解菌株的不同,分别通过水杨酸或邻苯二甲酸途径进入TCA循环,最终达到完全降解。
在既可以菲为唯一碳源生长,又可以萘为唯一碳源生长的多环芳烃降解菌株中,菲一般经过水杨酸途径进行降解[7,9]。该途径中,1-羟-2-萘甲酸通过1-羟-2-萘甲醛 (盐) 羟化酶转化为1,2-二羟萘,然后通过与萘类似的降解途径完成最终降解。假单胞菌属细菌多利用此途径。
对于那些可以菲为唯一碳源而不能利用萘的细菌,菲通过邻苯二甲酸途径降解[10-11]。即菲代谢为1-羟-2-萘甲酸后,在1-羟-2-萘甲酸双加氧酶作用下再次加氧开环生成反式2-羧基苯并丙酮酸,反式2-羧基苯并丙酮酸在水合-醛缩酶的作用下生成 2-羧基苯甲醛,然后在 2-羧基苯甲醛脱氢酶作用下生成邻苯二甲酸,随后经邻苯二甲酸双加氧酶、邻苯二甲酸二氢二醇脱氢酶及邻苯二甲酸脱羧酶转化为原儿茶酸,原儿茶酸进一步氧化开环后最终进入TCA循环彻底降解为二氧化碳和水。采用这种途径的细菌多见于分枝杆菌属、芽胞杆菌属、微球菌属及粪产碱菌等。

图3 推测的恶臭假单胞菌NCIB 9816 (水杨酸途径)、粪产碱菌AFK2 (邻苯二甲酸途径) 中菲的降解途径[9-10]Fig.3 Proposed metabolic pathways of phenanthrene inPseudomonas putidaNCIB 9816 (salicylate pathway) andAlcaligenes faecalisAFK2 (protocatechuate pathway)[9-10].
在有些菌株中,菲被羟基化C的初始位置有所不同,之后的降解也会采用不同的方式。如在节杆菌Arthrobactersp. P1-1降解菲的过程中,菲除了在3,4位C被羟基化后通过水杨酸或邻苯二甲酸途径进行降解外,还可以在l,2位C处被羟基化生成1,2-二羟菲,随后在脱氢酶和间位裂解酶的催化下产生2-羟-1-萘甲酸,进一步脱氢后生成 1,2-二羟萘,再通过水杨酸或邻苯二甲酸途径降解;或者9,10位C(K区) 被羟基化双加氧酶攻击,生成联苯甲酸[12],代谢过程见图 4A。在Mycobacterium vanbaaleniiPYR-1对菲降解中,也存在3条路径:一条是常见的3,4位C被羟基化后通过邻苯二甲酸途径降解;另外两条则是菲环的9,10位C (K区) 或者被双加氧酶攻击形成顺式-9,10-二氢二醇菲,而后在环裂解酶的作用下形成联苯甲酸;或者受单加氧酶的攻击形成9,10-环氧化菲,然后在环氧化物水解酶的催化下形成反式-9,10-二氢二醇菲[13-14],代谢过程见图4B。PAHs降解过程中,由于菌株及降解条件的不同,在降解过程中也可能会出现其他额外的中间代谢产物,如菲的降解在鞘氨醇单胞菌Sphingomonassp.GY2B中,经由菲→1-羟-2-萘甲酸→萘酚→水杨酸途径完成[15],代谢过程见图4C。
事实上,一个菌株通常并不一定仅仅含有一种菲降解途径,它可能同时拥有两条甚至两条以上的代谢途径,并且根据环境的条件来决定降解酶促反应的方向。
2 萘、菲细菌降解基因的遗传学分析
在PAHs降解中涉及到众多降解基因或基因簇,细菌利用这些基因编码的酶通过一系列酶促反应完成对萘、菲的降解。
2.1 降解基因及分类
PAHs的降解始于羟基化双加氧酶对苯环的羟基化,因此多组分的羟基化双加氧酶系统是整个降解过程中的关键酶、限速酶。在对该基因结构和功能的研究基础上,许多降解菌株的 PAHs上、下游降解途径的基因或基因簇被发现、定位和研究。nah-like基因是很大的一类降解基因,此外具有同样功能的其他基因也相继被报道,如phn基因、phd基因、nid基因和nag基因等[16]。
2.1.1nah-like基因
在以同源性关系为依据对 PAHs降解基因进行的分类中有很大一部分是经典的nah-like基因,如nah、pah、ndo和dox基因等[17]。

图4 推测的节杆菌P1-1 (A)、分枝杆菌PYR-1 (B) 和鞘氨醇单胞菌GY2B (C) 中菲的降解途径[12-13,15]Fig.4 Proposed metabolic pathways of phenanthrene inArthrobactersp. P1-1(A),Mycobacterium vanbaaleniiPYR-1(B) andSphingomonassp. GY2B(C)[12-13,15].
Pseudomonas putidaG7的 NAH7质粒和P. putidaNCIB 9816-4的pDTG1质粒是研究nah基因簇的经典代表[7-8]。以质粒NAH7中的nah基因簇为例,上游基因簇排列顺序为:nahAaAbAcAdBFCQED,其中nahAa编码萘羟基化双加氧酶 (NDO) 的铁氧还蛋白还原酶,nahAb编码 NDO铁氧还蛋白,nahAc编码NDO大亚基,nahAd编码NDO小亚基。nahB编码顺-萘双氢二醇脱氢酶,nahF编码水杨醛脱氢酶,nahC编码 1,2-双羟萘双加氧酶 (外二醇双加氧酶),nahQ为未知功能的开放阅读框 (Open reading frame,ORF),nahE编码顺-O-羟基苯脱萘丙酮酸水合-醛缩酶,nahD编码 2-羟-2H-苯并吡喃-2-羧基异构酶。下游基因簇排列顺序依次为:nahG(水杨酸羟化酶),nahH(儿茶酚 2,3-双加氧酶),nahI(羟基粘康酸半醛脱氢酶),nahN(羟基粘康酸半醛水解酶),nahL(2-氧戊-4-烯酸水合酶),nahM(2-O-4-羟基戊酸醛缩酶),nahK(4-草酰巴豆酯脱羧酶) 和nah J(4-草酰巴豆酯异构酶)[8]。与质粒 NAH7和质粒pDTG1极为相似的nah基因簇也分别在P.fluorescens菌株的质粒pKAl和pLP6a中发现[18-20]。
此外,P. putidaOUS82染色体中的pah基因簇pahAaAbAcAdBFCQED和P. aeruginosaPak1中的pah基因簇pahA1A2A3A4BFCQED、P. putida9816中质粒pWW60-1编码的ndo基因簇ndoABC以及Pseudomonassp. C18中质粒C18编码的dox基因簇doxABDEFGHIJ的基因排列顺序和DNA序列与P. putidaG7质粒NAH7的nah基因均有极高的相似性[7]。
nah、pah、ndo和dox这些基因通常被称为“经典的nah-like基因”,是一大类广泛存在并高度保守的PAHs降解基因。
2.1.2phn基因
phn基因与经典的nah-like基因功能相同,但不具同源性,两者在基因排列顺序和DNA序列上均不相同。虽然目前可分离培养的降解菌株主要以nah-like基因为主,但phn基因也是一类较为常见的降解基因。
菌株Burkholderiasp. RP007中的phn基因簇中有 9个开放阅读框,顺序为phnRSFECDAcAdB。其中phnS和phnR为调节基因,phdAc和phdAd分别编码羟基化双加氧酶大、小亚基,phdB编码顺-双氢二醇脱氢酶,phdC编码外二醇双加氧酶,phdD编码异构酶,phdE编码水合-醛缩酶,phdF编码醛脱氢酶[17]。与之极为相似的phn基因簇还有Sphingomonassp. CHY-1的phnA1aA2aB、phnA4CA3A2bA1bD基因簇 (其中phnA1aA2a编码羟基化双加氧酶 (PhnI)的大、小亚基,phnA2bA1b编码水杨酸-1-羟化酶(PhnII)),Acidovoraxsp. NA3 与Alcaligenes faecalisAFK2的phnAaBAcAdD、phnHG、phnCF基因簇,Cycloclasticussp. A5的phnA1bA1aA2C、phnA4A3D基因簇等[4,16,21]。
虽然在目前已知的可分离培养的降解菌中,phn基因并不是最优势基因型,但是 Laurie通过对phn和nah基因的竞争性PCR实验,指出在自然污染环境中 (包括可培养及不可培养菌群),phn基因的数量比nah基因更多[22]。
2.1.3phd基因
同源性分析表明phd是不同于nah-like和phn基因的另一类降解基因。
如Nocardioidessp. KP7的phd基因簇,包括两个定位于染色体上的降解基因簇phdIJK和phdEF-orf131-phdAB-orf72-phdGHCD。在第一个基因簇phdIJK中,phdI编码1-羟-2-萘甲酸双加氧酶,phdJ编码反式 2-羧基苯并丙酮酸水合-醛缩酶,phdK编码2-羧基苯甲醛脱氢酶。第二个基因簇中,phdA、phdB、phdC和phdD分别编码羟基化双加氧酶大、小亚基,铁氧还蛋白和还原酶,phdE编码双氢二醇脱氢酶,phdF编码外二醇双加氧酶,phdG编码水合-醛缩酶,phdH编码醛脱氢酶[11]。此外,与Nocardioidessp. KP7同源性很高的phd基因簇在菌株Mycobacteriumsp. SNP11 (基因簇phtCphdEFAB,其中phtC编码脱羧酶) 与Mycobacteriumsp. 6PY1中也存在,但后者具有两组分别由pdoA1B1和pdoA2B2基因编码的双加氧酶 Pdo1和 Pdo2,使得该菌株具有不同的PAHs底物选择性[23-24]。
2.1.4其他降解基因
环境中还存在一些其他类型的降解基因簇,如nid和nag基因。
Mycobacterium vanbaaleniiPYR-1的基因簇为phtRAaAbUBAcAdIJFG和nidB2DBA。其中phtR为调节基因,phtAa和phtAb编码邻苯二甲酸双加氧酶大、小亚基,phtU为未知功能基因,phtB编码邻苯二甲酸双氢二醇脱氢酶,phtAc和phtAd编码邻苯二甲酸双加氧酶的铁氧还蛋白和还原酶,phtI编码 1-羟-2-萘甲酸双加氧酶,phtJ编码反式2-羧基苯并丙酮酸水合-醛缩酶,phtF编码外二醇双加氧酶,phtG编码水合-醛缩酶,nidB2和nidB编码羟基化双加氧酶小亚基,nidD编码醛脱氢酶,nidA编码羟基化双加氧酶大亚基[14]。萘降解菌株Ralstoniasp. U2的基因簇为nagAaGHAbAcAdBFCQED和nagIKLMN。Dionisi采用该簇中nagAc基因 (编码萘羟基化双加氧酶大亚基) 的引物对煤焦油污染水样中降解菌株的nagAc-like基因进行rt-PCR实验,结果表明nagAc-like基因也是一类在萘降解中广泛存在的基因[25]。
2.2 降解基因的定位及水平转移
编码降解酶的基因可存在于细菌质粒上或染色体上,也可能同时由质粒和染色体编码。在长期的进化过程中不同菌株间存在一定程度的降解基因水平转移,为菌株适应污染环境提供了分子基础。
细菌中很多编码降解酶系的基因都存在于质粒DNA上,对微生物适应污染环境和污染物的降解有着特殊意义。在对萘、菲降解基因的研究中,人们首先考虑它们在质粒DNA上存在的可能性。已经发现的经典质粒有P. putidaG7的NAH7质粒、P. putidaNCIB 9816-4的pDTG1质粒和Pseudomonassp. C18的C18质粒等[7-8,11]。随着芳香环数量增加,所需的降解基因及基因簇更大,这时降解基因通常位于染色体上。如采用分子杂交技术发现Nocardiodessp.KP7包含的8个降解基因均位于菌株的染色体DNA上[11]。Acidovoraxsp. NA3和P. putidaOUS82也具有类似的状况[7,16]。在有的降解菌株中,染色体分担质粒的负担或质粒线性扩大化,降解基因会同时定位于染色体和质粒中。如菲降解菌株P. putidaBS3701的降解质粒丢失后,它利用菲、萘、1-羟-2-萘甲酸的能力也随着消失,但仍可利用水杨酸,说明有部分降解基因定位于染色体 DNA上[26]。Herrick从煤焦油污染环境中分离出的一株萘降解细菌,其nah基因在菌株的染色体上和质粒上均有分布[27]。当然,降解基因也会在染色体和质粒上进行交换、转移[28]。
污染环境对细菌的筛选作用,使得细菌在长期的进化过程中存在一定水平的降解基因水平转移,包括种内和种间。如 Wilson从山坡地带分离的降解菌株的降解基因属phnAc类基因,沉积地带属nahAc类基因,但是这些含有相同类型降解基因菌株的16S rRNA基因同源性很低[27,29]。这种出现在不同菌株中相互关系紧密的降解基因是基因水平转移的结果,是生物适应环境的物质基础。
3 萘、菲的细菌降解过程中其他相关基因
PAHs高的正辛醇-水分配系数使其倾向结合于细胞膜中,破坏膜的结构和功能,进一步影响和抑制降解菌的生长[30-32]。因此在 PAHs微生物降解过程中,细菌不仅表达分解代谢途径中的降解酶,通常还会表达大量的应答相关蛋白。
转运蛋白的高表达是菌体抵御PAHs胁迫的一个主要方面。如P. fluorescensLP6a中编码转运蛋白的外排泵基因emhB与编码膜融合蛋白的emhA基因及编码外膜蛋白的emhC基因构成了一个属于RND超家族的PAHs外排泵基因簇emhABC,该簇在 PAHs胁迫条件下高表达并可介导PAHs的主动外排。此外,P. putidaDOT-T1E中的3个甲苯耐受外排泵 TtgABC、TtgDEF和TtgGHI以及P. putidaS12中受 PAHs诱导的溶剂抗性蛋白 ABC泵(SrpABC) 都可以通过转运蛋白表达提高介导的PAHs外排,产生对有机溶剂及PAHs的高抗性[30,33]。
在 PAHs胁迫条件下,菌体的压力相关蛋白也会对细菌产生重要的保护作用。如大肠杆菌压力相关基因katG、recA、fabA和grpE分别编码应对氧化损伤、DNA、细胞膜和蛋白质破坏的压力蛋白,在菲、萘和苯并芘胁迫条件下的表达量均有不同程度的改变[34]。
对甲苯胁迫条件下P. putidaS12的蛋白质组学研究表明:除了转运蛋白 ABC transporter和 BraC(Branched-chain amino acid ABC transporter),以及压力相关蛋白 GroEL (Chaperonin)、Ohr (Organic hydroperoxide resistance protein)、TerZ (Tellurium resistance protein) 和 Cold-shock domain family protein之外,参与应答反应的蛋白质还包括能量代谢相关蛋白 SdhB (Succinate dehydrogenase,iron-sulfur protein)、SucD (Succinyl-CoA synthetase,α-subunit)、AtpF (ATP synthase F0,B subunit)、LpdG(Dehydrogenase component) 等;外膜蛋白 OprH(Outer membrane protein H1)、OprF (Outer membrane protein);基因翻译相关蛋白RplA (Ribosomal protein L1)、RbfA (Ribosome-binding factor A)、Frr(Ribosome recycling factor) 等和未知功能蛋白PP3611[32]。
本实验室在前期研究中也发现 PAHs降解菌株P. aeruginosaPAO1中一些表达明显受萘、菲调节的基因。如编码能量代谢相关蛋白 (PA3171和PA3392)、生物大分子合成代谢相关蛋白 (PA2584和PA5013)、转运蛋白 (PA4687)、膜蛋白 (PA4008)及未知功能蛋白 (PA5220) 等。
在 PAHs作为降解底物和压力物质的双重作用下,大量应答相关蛋白的表达使得细菌得以应对PAHs的胁迫作用,最终实现PAHs的降解。
4 小结及展望
萘、菲属于低分子PAHs,在环境中普遍存在,通常被用作研究 PAHs降解的模式化合物。通过对萘、菲降解途径、机理的不断研究,已经确定的相关降解基因越来越多,功能也越来越明确,这些都为 PAHs的细菌降解途径以及降解菌株分子遗传机制的研究提供了理论依据。
目前,人们对 PAHs的生物降解已有较广泛和深入的认识,但 PAHs生物修复工程仍然存在很多问题,以下几方面的研究有待于进一步深入:1) 以芴、芘等为代表的四环以上高分子量 PAHs的降解机理研究,为PAHs尤其是高分子量的PAHs生物降解的应用奠定理论基础。2) 生物多样性导致了PAHs降解途径的多样性,全面把握不同种属细菌的PAHs降解机理及它们降解途径间的异同,为PAHs混合物的生物降解或混合菌群降解 PAHs的应用提供理论依据。3) 不同生态环境,尤其是极端环境条件下,PAHs降解菌株的生存及降解能力或途径变化的研究,为实际环境中 PAHs生物降解的应用提供理论依据。4) PAHs降解基因的水平转移及在PAHs降解过程中菌株基因调控机制的研究,为从全基因组水平对降解菌株的分子及遗传机制的研究提供理论依据。
因此,在筛选、分离能够高效降解 PAHs的微生物、研究菌株降解特性的基础上,从分子水平探索其降解途径,丰富降解酶及其他相关基因的信息,全面了解和完善 PAHs的生物降解过程,最终通过基因重组构建 PAHs高效降解菌株,并实际应用于PAHs的生物修复,是实现有效地检测PAHs环境污染以及修复PAHs污染的重要手段。
REFERENCES
[1] Haritash AK, Kaushik CP. Biodegradation aspects of polycyclic aromatic hydrocarbons (PAHs): a review.J Hazardous Mat, 2009, 169(1/3): 1−15.
[2] Seo J, Keum Y, Li QX. Bacterial degradation of aromatic compounds.Int J Environ Res Public Health, 2009, 6:278−309.
[3] Seo J, Keum Y, Harada RM,et al. Isolation and characterization of bacteria capable of degrading polycyclic aromatic hydrocarbons (PAHs) and organophosphorus pesticides from PAH-contaminated soil in Hilo, Hawaii.J Agric Food Chem, 2007, 55: 5383−5389.
[4] Kasai Y, Shindo K, Harayama S,et al. Molecular characterization and substrate preference of a polycyclic aromatic hydrocarbon dioxygenase fromCycloclasticussp. strain A5.Appl Environ Microbiol, 2003, 69(11):6688−6697.
[5] Kim S, Kweon O, Jones RC,et al. Genomic analysis of polycyclic aromatic hydrocarbon degradation inMycobacterium vanbaaleniiPYR-1.Biodegradation,2008, 19: 859−881.
[6] Davies JI, Evans WC. Oxidative metabolism of naphthalene by soilPseudomonads. The ring-fission mechanism.Biochem J, 1964, 91: 251−261.
[7] Takizawa N, Iida T, Sawada T,et al. Nucleotide sequences and characterization of genes encoding naphthalene upper pathway ofPseudomonas aeruginosaPaKl andPseudomonas putidaOUS82.J Biosci Bioeng, 1999,87(6): 721−731.
[8] Harayama S, Rekik M, Wasserfallen A,et al. Evolutionary relationships between catabolic pathways for aromatics:conservation of gene order and nucleotide sequences of catechol oxidation genes of pWW0 and NAH7 plasmids.Mol Gen Genet, 1987, 210: 241−247.
[9] Yang Y, Chen RF, Shiaris MP. Metabolism of naphthalene,fluorene, and phenanthrene: preliminary characterization of a cloned gene cluster fromPseudomonas putidaNCIB 9816.J Bacteriol, 1994, 176(8): 2158−2164.
[10] Kiyohara H, Nagao K, Kouno K,etal.Phenanthrene-degrading phenotype ofAlcaligenes faecalisAFK2.Appl Environ Microbiol, 1982, 43(2): 458−461.
[11] Saito A, Iwabuchi T, Harayama S. A novel phenanthrene dioxygenase fromNocardioidessp. strain KP7: expression inEscherichia coli.J Bacteriol, 2000, 182(8): 2134−2141.
[12] Seo J, Keum Y, Hu Y,et al. Phenanthrene degradation inArthrobactersp. P1-1: initial 1,2-, 3,4- and 9,10-dioxygenation, and meta- and ortho-cleavages of naphthalene-1,2-diol after its formation from naphthalene-1,2-dicarboxylic acid and hydroxyl naphthoic acids.Chemosphere, 2006, 65(11): 2388−2394.
[13] Moody JD, Freeman JP, Doerge DR,et al. Degradation of phenanthrene and anthracene by cell suspensions ofMycobacteriumsp. strain PYR-1.Appl Environ Microbiol,2001, 67(4): 1476−1483.
[14] Stingley RL, Khan AA, Cerniglia CE. Molecular characterization of a phenanthrene degradation pathway inMycobacterium vanbaaleniiPYR-1.Biochem Biophys Res Commun, 2004, 322(1): 133−146.
[15] Tao X, Lu G, Dang Z,et al. A phenanthrene-degrading strainSphingomonassp. GY2B isolated from contaminated soils.Proc Biochem, 2007, 42: 401−408.
[16] Singleton DR, Ramirez LGN, Aitken MD.Characterization of a polycyclic aromatic hydrocarbon degradation gene cluster in a phenanthrene-degradingAcidovoraxstrain.Appl Environ Microbiol, 2009, 75(9):2613−2620.
[17] Laurie AD, Lloyd-Jones G. Thephngenes ofBurkholderiasp. strain RP007 constitute a divergent gene cluster for polycyclic aromatic hydrocarbon catabolism.J Bacteriol,1999, 181(2): 531−540.
[18] Sanseverino J, Applegate BM, King JMH,et al.Plasmid-mediated mineralization of naphthalene,phenanthrene, and anthracene.Appl Environ Microbiol,1993, 59(6): 1931−1937.
[19] Menn F, Applegate BM, Sayler GS. NAH Plasmid-mediated catabolism of anthracene and pPhenanthrene to naphthoic acids.Appl Environ Microbiol, 1993, 59(6): 1938−1942.
[20] Foght JM, Westlake DWS. Transposon and spontaneous deletion mutants of plasmid-borne genes encoding polycyclic aromatic hydrocarbon degradation by a strain ofPseudomonas fluorescens.Biodegradation, 1996, 7:353−366.
[21] Che SD, Meyer C, Micoud J,et al. Identification and functional analysis of two aromatic-ring-hydroxylating dioxygenases from aSphingomonasstrain that degrades various polycyclic aromatic hydrocarbons.Appl Environ Microbiol, 2004, 70(11): 6714−6725.
[22] Laurie AD, Lloyd-Jones G. Quantification ofphnAcandnahAcin contaminated New Zealand soils by competitive PCR.Appl Environ Microbiol, 2000, 66(5): 1814−1817.
[23] Pagnout C, Frache G, Poupin P,et al. Isolation and characterization of a gene cluster involved in PAH degradation inMycobacteriumsp. strain SNP11:expression inMycobacterium smegmatismc2155.Res Microbiol, 2007, 158: 175−186.
[24] Krivobok S, Kuony S, Meyer C,et al. Identification of pyrene-induced proteins inMycobacteriumsp. strain 6PY1: evidence for two ring-hydroxylating dioxygenases.J Bacteriol, 2003, 185(13): 3828−3841.
[25] Dionisi HM, Chewning CS, Morgan KH,et al. Abundance of dioxygenase genes similar toRalstoniasp. strain U2nagAcis correlated with naphthalene concentrations in coal tar-contaminated freshwater sediments.Appl Environ Microbiol, 2004, 70(7): 3988−3995.
[26] Balashova NV, Kosheleva IA, Golovchenko NP,et al.Phenanthrene metabolism byPseudomonasandBurkholderiastrains.Proc Biochem, 1999, 35: 291−296.
[27] Herrick JB, Stuart-Keil KG, Ghiorse WC,et al. Natural horizontal transfer of a naphthalene dioxygenase gene between bacteria native to a coal tar-contaminated field site.Appl Environ Microbiol, 1997, 63(6): 2330−2337.
[28] Ka JO, Tiedje JM. Integration and excision of a 2,4-dichlorophenoxyacetic acid-degradative plasmid inAlcaligenes paradoxusand evidence of its natural intergeneric transfer.J Bacteriol, 1994, 176(17):5284−5289.
[29] Wilson MS, Herrick JB, Jeon CO,et al. Horizontal transfer ofphnAcdioxygenase genes within one of two phenotypically and genotypically distinctive naphthalene-degrading guilds from adjacent soil environments.Appl Environ Microbiol, 2003, 69(4):2172−2181.
[30] Ramos JL, Gallegos M, Marqués S,et al. Responses of Gram-negative bacteria to certain environmental stressors.Curr Opin Microbiol, 2001, 4: 166−171.
[31] Trautwein K, Hner SK, Hlbrand LW,et al. Solvent stress response of the denitrifying bacterium “Aromatoleum aromaticum” strain EbN1.Appl Environ Microbiol, 2008,74(8): 2267−2274.
[32] Volkers RJM, de Jong AL, Hulst AG,et al.Chemostat-based proteomic analysis of tolueneaffectedPseudomonas putidaS12.Environ Microbiol, 2006, 8(9):1674−1679.
[33] Hearn EM, Dennis JJ, Gray MR,et al. Identification and characterization of theemhABCefflux system for polycyclic aromatic hydrocarbons inPseudomonas fluorescenscLP6a.JBacteriol, 2003, 185(21):6233−6240.
[34] Kim YS, Min J, Hong HN,et al. Gene expression analysis and classification of mode of toxicity of polycyclic aromatic hydrocarbons (PAHs) inEscherichia coli.Chemosphere, 2007, 66: 1243−1248.
Recent advances in bacterial biodegradation of naphthalene,phenanthrene by bacteria: a review
Dan Zhang, Zhaoge Li, Xinguang Bao, Jiangbo Li, Haihua Liang, Kangmin Duan, and Lixin Shen
Key Laboratory of Resources Biology and Biotechnology in Western China,Ministry of Education,Faculty of Life Sciences,Northwest University,Xi’an710069,China
Received:December 31, 2009;Accepted:March 15, 2010
Supported by:National Science Foundation of Shaanxi Province (No. 2004B18), Scientific Technology Program of Xi’an City (No. GG04065).
Corresponding author:Lixin Shen. Tel/Fax: +86-29-88302132; E-mail: shenlx68@nwu.edu.cn
陕西省自然科学基金项目 (No. 2004B18),西安市科技计划基金项目 (No. GG04065) 资助。

