玉米种质资源遗传多样性分析
程莉莉,刘丽君,林 浩,唐晓飞,魏 崃,吴广锡,杨 吉吉,张 颖
(1.东北农业大学农学院,哈尔滨 150030;2.黑龙江省农业科学院大豆研究所,哈尔滨 150086;3.黑龙江省菽锦科技有限责任公司,哈尔滨 150086)
玉米是我国重要的粮食和饲料作物[1],玉米生产在国民经济中占有重要的地位。玉米种质资源包括各类地方品种、综合种、复合种、杂交种、中间材料、自交系及玉米野生近缘种等,是玉米生产、利用和新品种选育的物质基础[2]。玉米种质遗传基础狭窄一直是限制我国玉米育种发展的重要因素[3],研究种质的遗传多样性对玉米育种和种质利用与保护具有重要的指导意义[4]。随着分子生物学的发展,分子标记技术为种质遗传多样性研究提供了新手段和新方法[1]。目前,各种分子标记技术已广泛地应用于玉米遗传多样性的研究中。郑得刚等利用SSR标记技术研究了黑龙江省常用玉米自交系的遗传多样性,并将玉米种质归纳为Reid和非Reid两大类[5];杜金友等利用AFLP分子标记技术对55份玉米种质材料进行了研究,结果表明,AFLP有利于玉米遗传多样性分析[6];Mumm等在采用46个探针酶的RFLPs基础上聚类分析了美国148个玉米自交系,并将材料进行了类群的划分[7];黄世全等通过RAPD分子标记技术对16个玉米自交系进行遗传多样性和聚类分析,其聚类分析结果与系谱分析大体相似[8];姜树坤等利用一种基于PCR的新型标记技术SRAP标记对16个玉米自交系进行了遗传多样性分析,结果表明SRAP标记是一种适合于玉米遗传多样性研究的分子标记[9]。
本研究采用具有方法简单、多态性好、可靠性高等优点的SSR分子标记技术[10],对383份玉米材料进行遗传多样性研究,利用UPGMA方法对材料进行聚类分析,进行优势群的初步归类与划分,为今后育种实践中有目的地选择亲本、拓宽遗传基础培育玉米新品种提供理论依据。
1 材料与方法
1.1 材料
供试材料是由黑龙江省菽锦科技有限责任公司提供的383份不同地区来源的玉米自交系。其中,黑龙江材料107份;内蒙古材料111份;美国材料12份;意大利材料25份;中国南方收集材料87份,以及其他来源材料41份。材料名称及来源见表1。
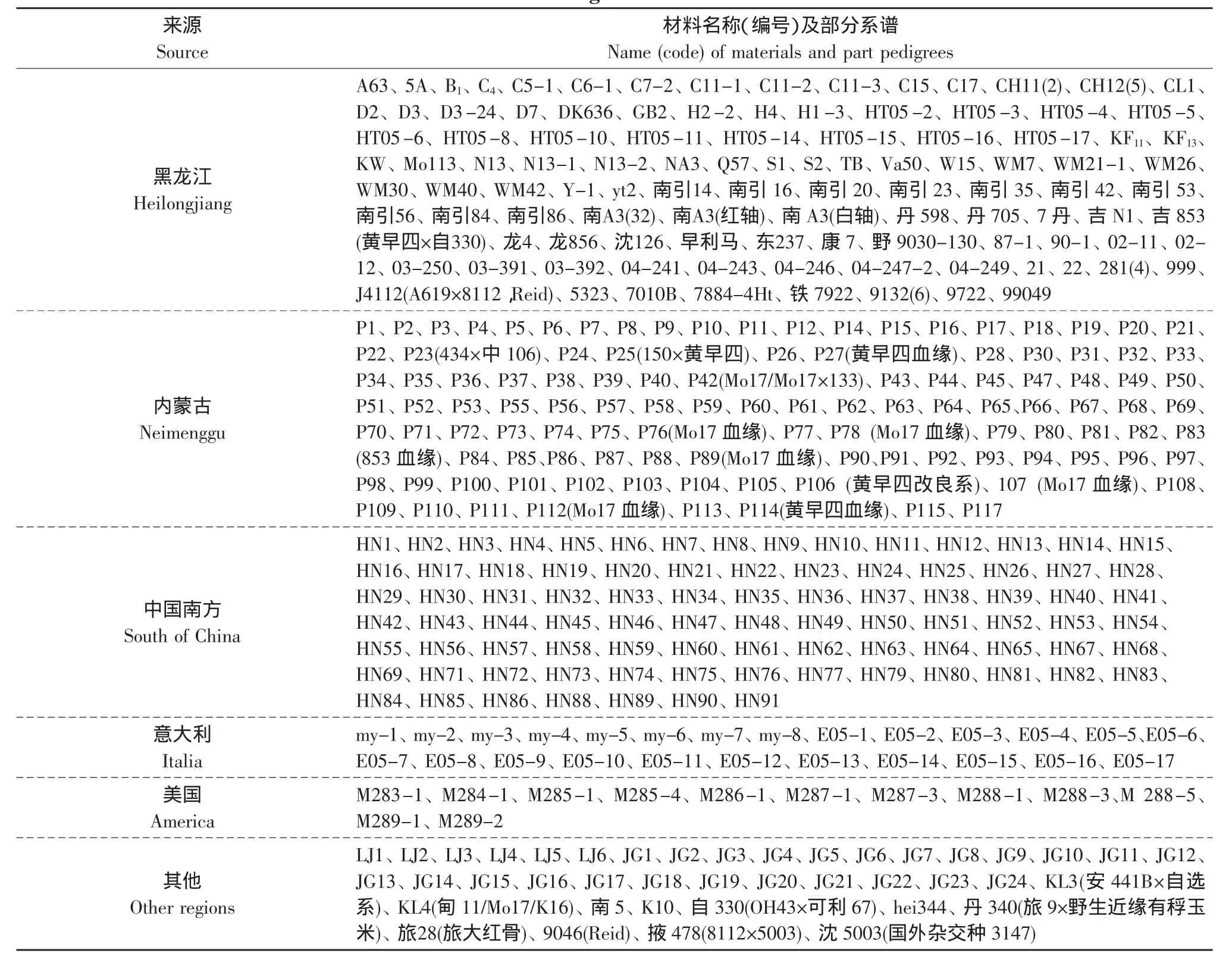
表1 383份玉米自交系名称及来源Table 1 Names and origins of 383 maize inbred lines
在383份玉米材料中,部分材料属于Lancaster群、Reid群、旅大红骨群、唐四平头群等玉米优势群,这些优势群是根据玉米种质资源系谱和地理来源进行划分的[11]。将已经确定优势群归属的玉米材料与其他遗传背景不详的材料进行聚类分析,可以作为判断材料优势群划分的一个参考。
1.2 方法
1.2.1 DNA提取
取玉米幼苗时期的叶片,利用CTAB的方法提取玉米总DNA[5]。用紫外分光光度计检测DNA质量和浓度。
1.2.2 SSR分析
SSR引物序列信息通过Maize GDB数据库中获得,选用的引物由上海生物工程公司(Sangon)合成,试验所选用的引物名称见表2。利用已筛选出多态性较好的26对SSR引物对玉米总DNA进行PCR扩增反应。反应体系为每20 μL体积中含10×buffer(含 20 mmol·L-1Mg2+)2 μL,dNTP(25 mmol·L-1)0.2 μL,引物(20 μmol·L-1)0.26 μL,TaqDNA 聚合酶(2 U·μL-1)0.5 μL,DNA 模板 5.0 μL,ddH2O 12.04 μL。PCR扩增反应程序为: 94℃预变性5 min,然后以94℃变性1 min,退火2 min(退火温度根据引物的具体Tm值进行调整),70℃延伸2 min,35个循环反应,最后70℃延伸5 min,4℃保存。
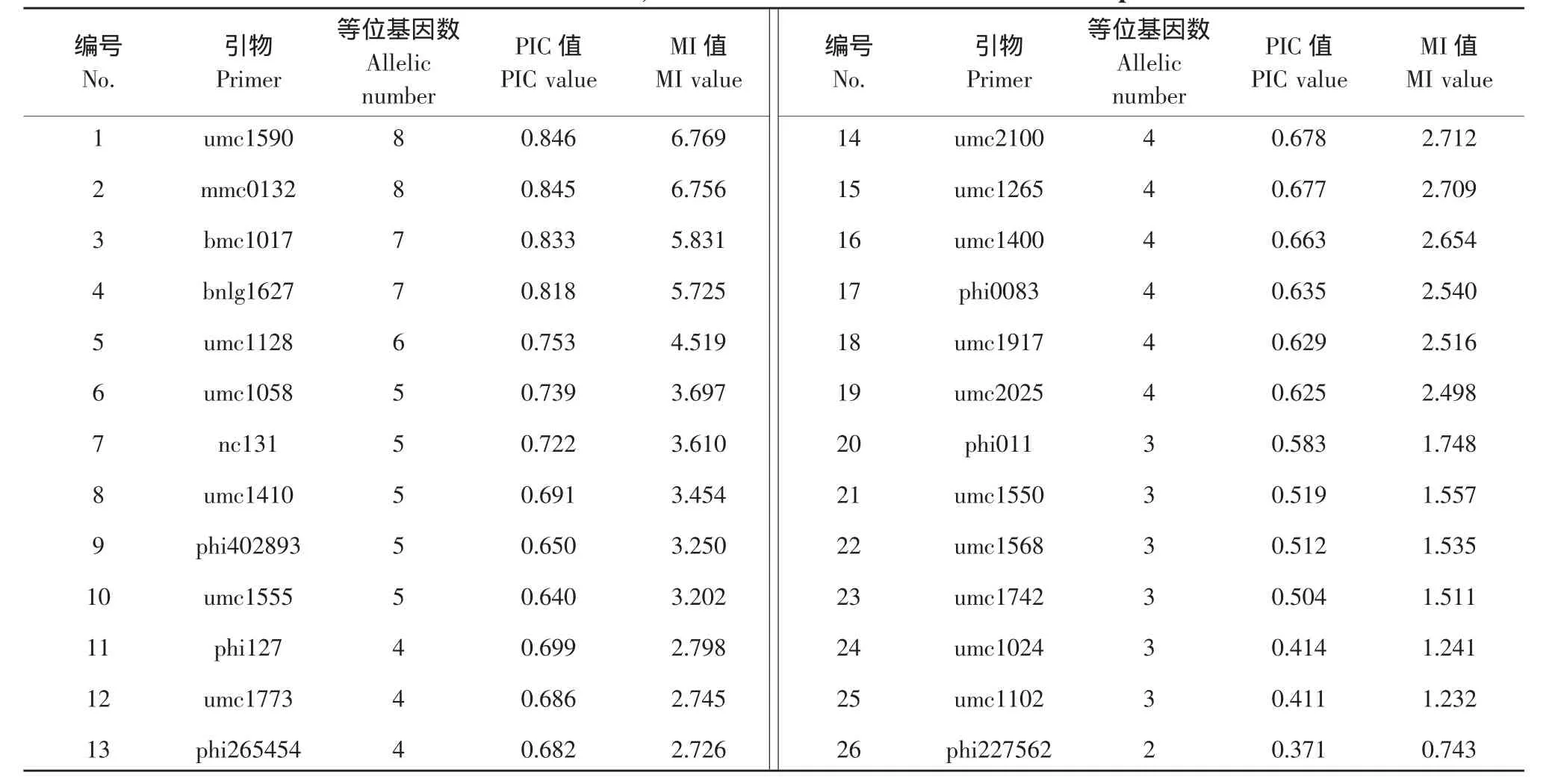
表2 26对SSR引物名称和检测到的多态性指标Table 2 Allelic numbers,PIC values and MI values for 26 SSR primers
1.2.3 电泳
PCR扩增产物变性后通过6%聚丙烯酰胺凝胶进行电泳,恒定功率60 W,时间约90 min。采用快速银染法进行染色。
1.2.4 数据处理
SSR扩增带型以0、1、9进行统计,即在相同迁移率位置上,有条带记为1,无条带记为0,缺失数据记为9,建立分子标记数据库。
以简单相配系数(Simple matching coefficient)估计基因频率,依据公式计算品种间的遗传相似系数(Genetic similarity coefficient),计算公式为:GS=m/(m+n),m表示基因型间共有带数目,n表示基因型间差异带数目。为了比较品种间遗传差异,利用Ntsyspc-2.10e软件对383份材料进行聚类分析,在遗传相似系数矩阵的基础上,用非加权平均法(Unweighted paired group method using arithmetic averages,UPGMA)构建383份材料的树状聚类分析图。
多态性信息量(Polymorphism information content,PIC)是指某个位点的预期异质结合度,指从群体中随机选取的两个个体在某位点具有不同等位变异的可能性,多用来表示标记检测遗传多样性。根据Senior等提出的公式[12]:PIC=1-∑fi2,对每对SSR引物的多态性信息量进行计算,其中fi表示某一位点的第i个等位变异在群体中出现的频率。
每对SSR引物的标记索引系数(Marker index,MI)根据Smith等提出的公式[10]:MI=等位基因数×PIC,进行计算。
2 结果与分析
2.1 遗传多样性分析
本研究利用筛选出的26对能够扩增出带型稳定、多态性好的玉米SSR引物,对383份玉米材料进行遗传多样性分析。结果表明,383份玉米材料中,共检测到117个等位基因变异,每个位点的等位基因数为2~8个,平均4.5个。检测等位基因数最多的引物为umc1590和mmc0132,等位基因数都为8个;最少的引物为phi227562,等位基因数为2个。26个SSR位点均表现出多态性,表明在分子标记水平上,研究的383份玉米材料具有较丰富的遗传变异。
26个SSR位点的多态性信息量(PIC值)变异范围在0.371~0.846之间,平均为0.647。标记索引值(MI)的变化范围介于0.743~6.769之间。综合数据表明,等位基因数、PIC值以及MI值,这三个衡量引物多态性指标的结果相一致,说明材料之间具有较高的遗传多样性。研究结果表明,SSR标记技术具有较强的检测玉米遗传多态性的能力。
2.2 聚类分析
根据统计的SSR标记数据结果,通过软件分析计算,得到383份玉米材料之间的遗传相似系数。根据383份玉米材料之间的遗传相似系数矩阵,利用UPGMA法进行分析,得到聚类分析图(见表3)。在遗传相似系数为0.536的水平上,可以将383份玉米材料划分为六类。其中367份玉米材料可被划分在第Ⅰ类中;第Ⅱ类有2份材料,HN43和HN51;第Ⅲ类有1份材料:9722;第Ⅳ类有1份材料,南引84;第Ⅴ类有7份材料,HN53、HN83、HN71、HN59、HN72、04-246 和WM40;第Ⅵ类有5份材料,HN90、P67、P69、P70、P71。
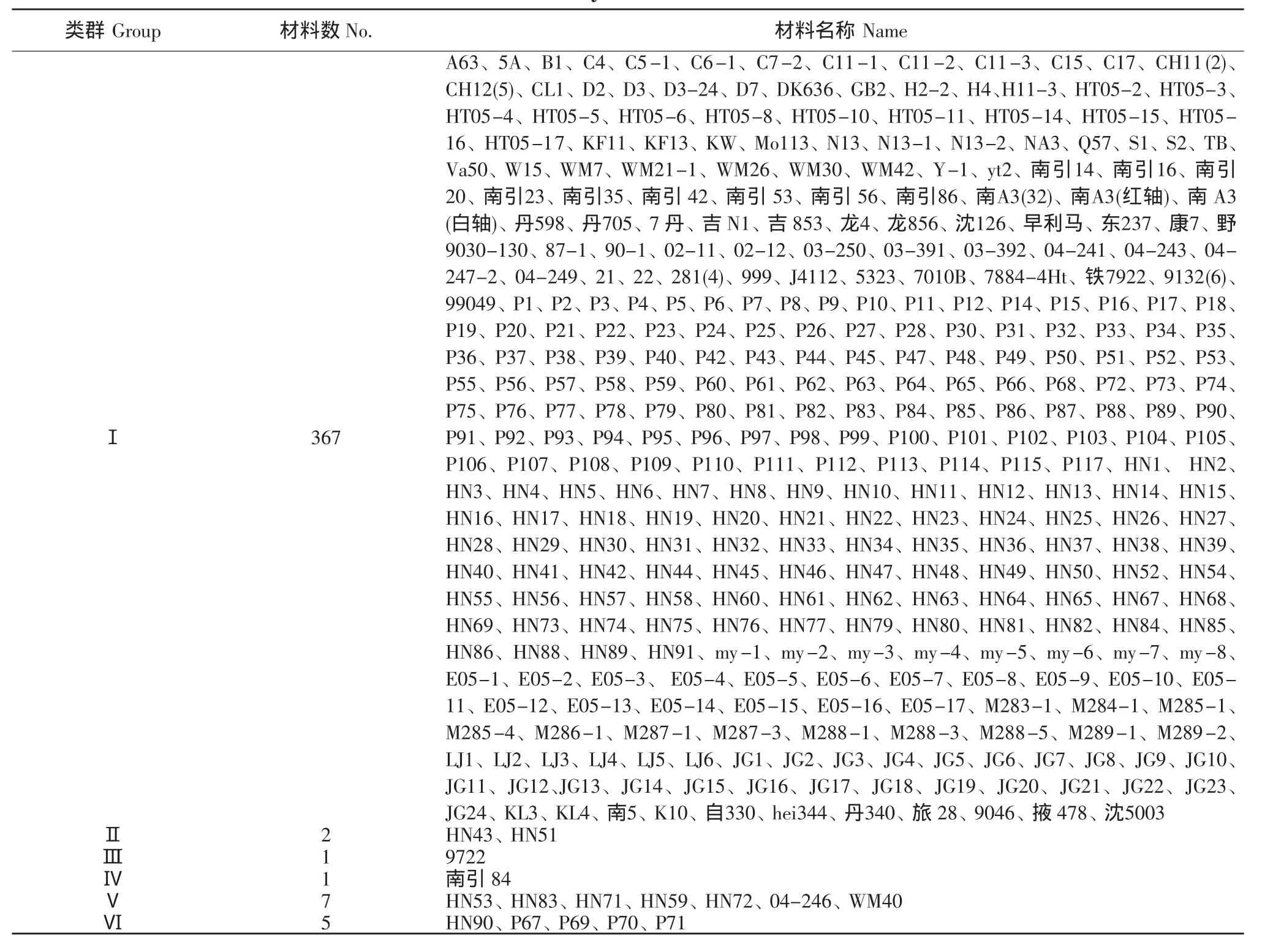
表3 383份玉米材料的SSR聚类结果Table 3 Cluster analysis of 383 maize inbred lines
由于第Ⅰ类材料数量多,可以根据遗传相似系数将第Ⅰ类进行亚类的划分。根据表3所显示的相关信息,参照其中已知系谱来源及优势群归属的玉米材料,对第Ⅰ类中的其他玉米材料进行具体的优势类群划分。
①在遗传相似系数为0.646的水平上(见图1),6份意大利材料:my-1、my-3、my-5、my-6、my-7和my-8,与丹598聚为一类,在这6份意大利材料中,与丹598亲缘关系较近的是my-6,与丹598亲缘关系较远的是my-7。
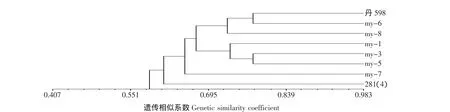
图1 6份意大利材料的聚类分析Fig.1 Cluster analysis for six maize inbred lines from Italia
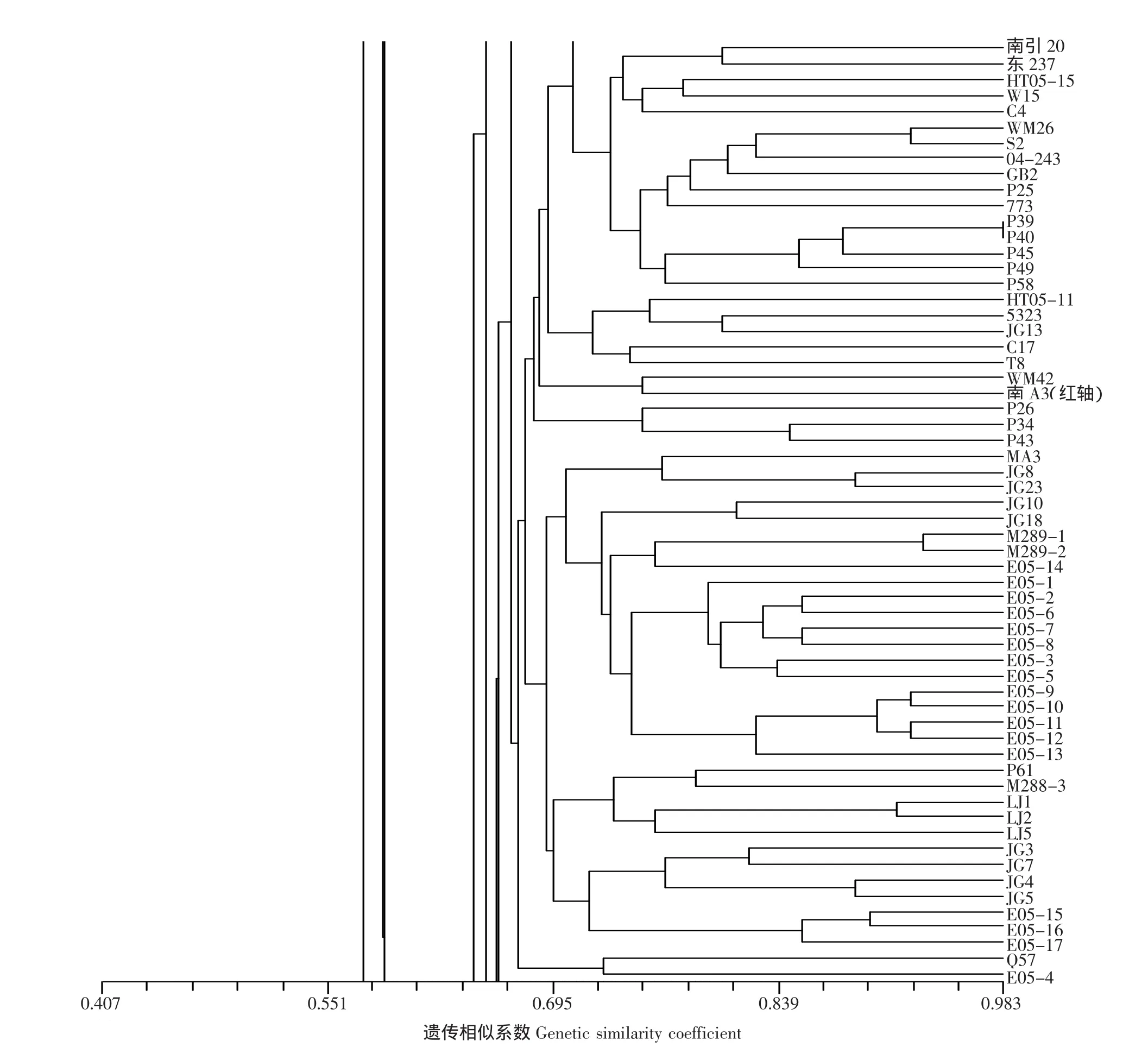
图2 意大利与美国材料的聚类分析Fig.2 Cluster analysis for maize inbred lines from Italia and America
②由图2可以看出,17份意大利E05系列材料,在遗传相似系数为0.669水平上被划分为一类,并且与材料东237,以及3份美国材料(M288-3、M289-1和M289-2)聚在同一类中,说明E05系列材料与部分美国材料亲缘关系相近。
③在遗传相似系数为0.666的水平上,12份美国材料中,有9份材料与已知属于Reid类群的材料(如:沈5003、掖478、9046)聚为一类,由此可以确定此材料所在类群属于Reid优势群。由图3可知,编号为P27的材料是黄早四的改良系,以及属于旅大红骨类群的旅28也被划分到Reid类群中。
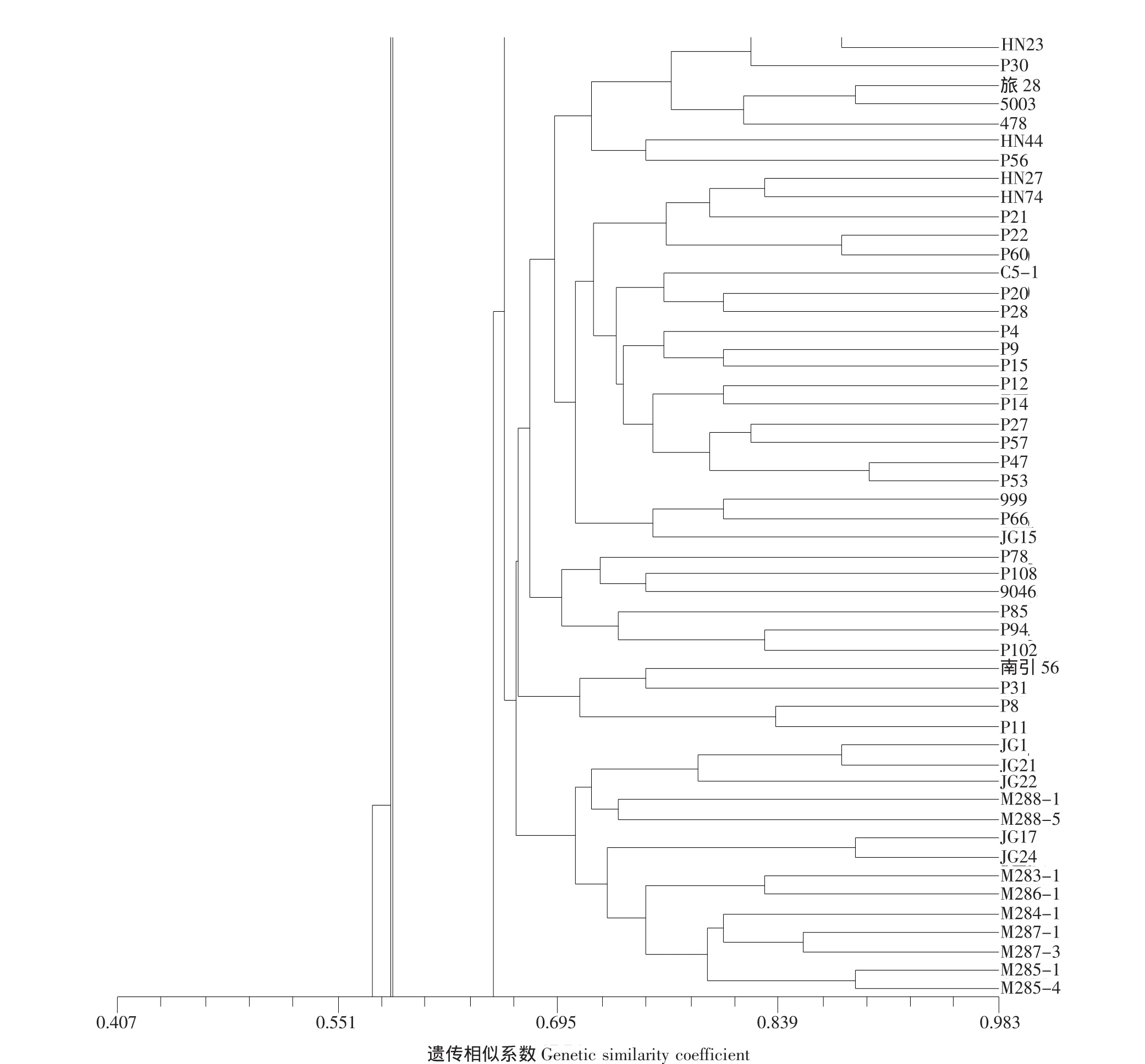
图3 12份美国材料的聚类分析Fig.3 Cluster analysis for 12 maize inbred lines from America
④ 在 JG16、JG11、P72、P75、P83、P87、P90、P91、P96、P97、P99、P100、P104、P105、P106、P109和P114这17份材料中,因P106、P114和P83都含有黄早四血缘,可将它们在遗传相似系数为0.683处,聚为一类,材料间亲缘关系相近,遗传背景相似,可能含有黄早四血缘,黄早四是唐四平头类群的代表,因此可将此类群称为唐四平头群。结果见图4。
⑤ 编号为 P74、P76、P77、P79、P80、P82、P89、P92、P93、P95、P98、P103和P107的材料,可以在遗传系数为0.695处,将它们聚为一类,这些材料多数属于Mo17的改良系,Mo17是Lancaster类群的代表,因此可以将这一类材料所在的类群称为Lancaster群。结果见图5。
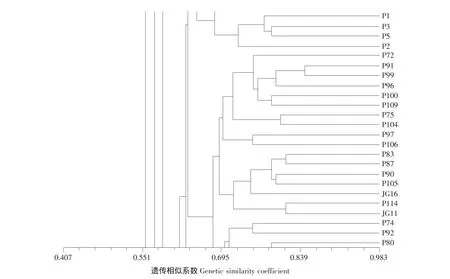
图4 17份玉米材料的聚类分析Fig.4 Cluster analysis for 17 maize inbred lines
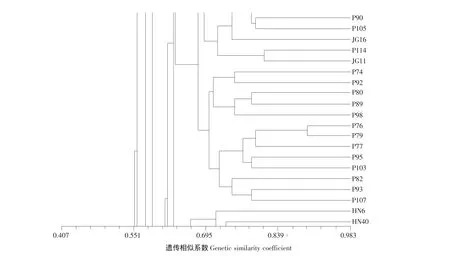
图5 13份玉米材料的聚类分析Fig.5 Cluster analysis for 13 maize inbred lines
除了367份玉米材料以外,只有16份材料未被划分在第一类中,而是被划分到另外的5类中。由图6可知,这5大类的品种与第Ⅰ类的遗传距离较大,亲缘关系较远。在第Ⅵ类中,P67为Y0261改良系,P69为郑32与3042的改良系,根据两个品种的相关信息,可以初步确定划分到同一类组群中其他材料的遗传背景与亲缘关系。
由聚类分析图可以看出,根据已知遗传背景材料的分布,可以将玉米材料进行初步的类群归类,由于材料数量大,仍有一部分材料未被划分到已知的玉米优势群体中。通过对玉米自交系材料的聚类分析,可对玉米种质的整理和分类,选育自交系材料、选配组合和种质改良等研究提供一定的参考依据。
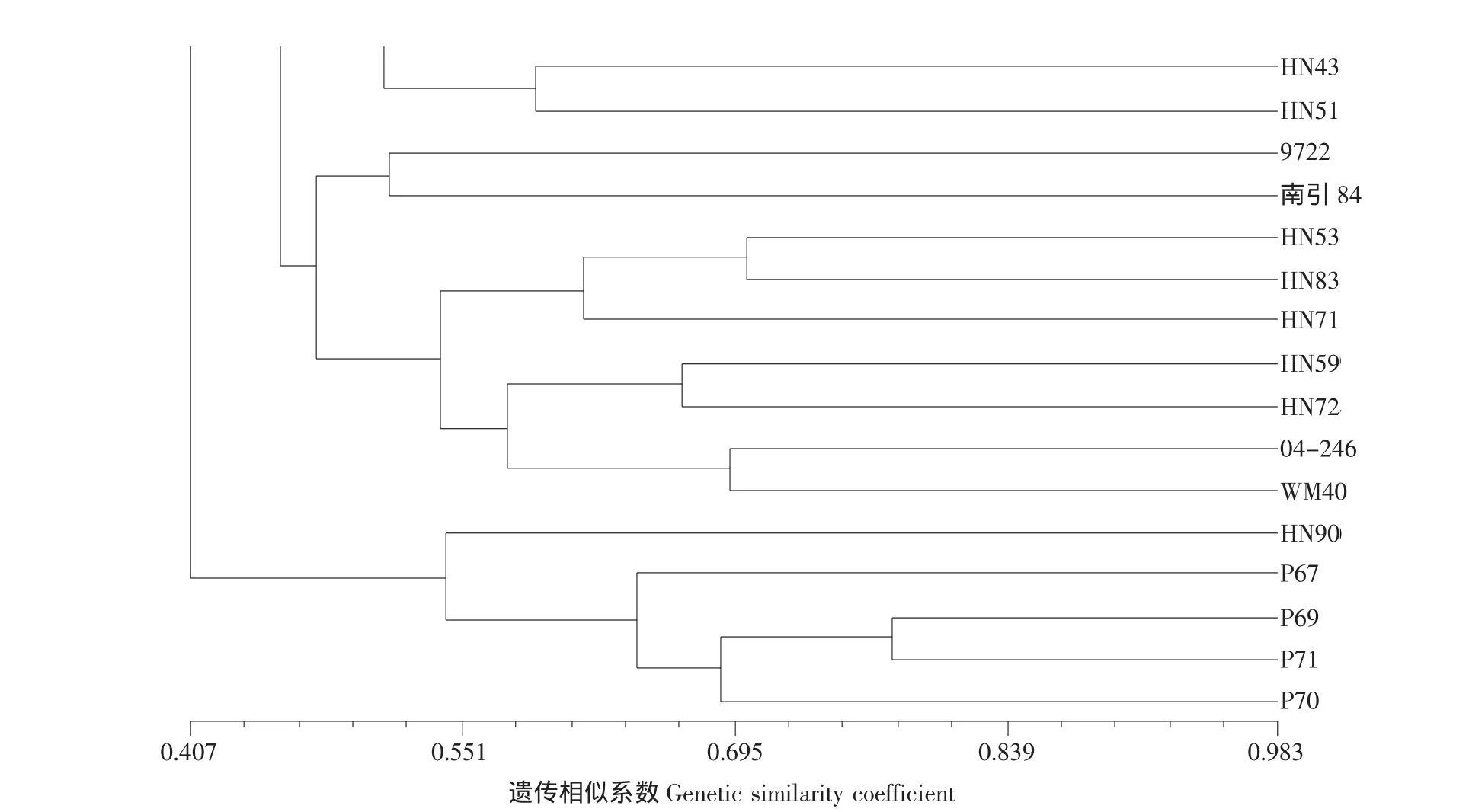
图6 16份玉米材料的聚类分析Fig.6 Cluster analysis for 16 maize inbred lines
3 讨论
3.1 SSR标记与玉米遗传多样性分析
目前,分子标记技术已经广泛地应用到玉米种质遗传多样性研究上。对常用的几种分子标记应用进行比较。RFLP标记需要DNA量多,技术复杂,费用大;RAPD标记虽然需要DNA量少,分析程序简单,但它属于显性标记,且重复性差;AFLP标记多态性好,但由于受到专利保护,进入商业化应用受到一定限制,且费用较高,技术复杂,工作量大,限制了它的应用[13]。SSR标记因其具有数量丰富、共显性遗传、实验重复性好、DNA质量要求不高以及费用低等优点,使得SSR标记被认为是最适合分析玉米种质遗传多样性及划分杂种优势群的分子标记[14-15]。
本文利用26对SSR引物分析了383份玉米材料的遗传多样性,平均每个位点的等位基因数为4.5个,平均PIC值为0.647。这一结果低于Taramino等得到的平均位点等位基因数6.6和平均PIC值0.76[16],低于张建华等得到的平均位点等位基因数11.4和平均PIC值0.709[17],高于吴永升等得到的平均位点等位基因数3.92和平均PIC值0.59和聂永心等得到的平均位点等位基因数4.01和PIC值0.599[18-19]。造成这些差异的可能原因有收集到的玉米材料绝大部分来自于我国,由于我国玉米种质资源的遗传基础较窄,因此需要引进外国种质进行玉米种质的改良。
3.2 玉米材料的优势类群的划分
利用SSR标记技术对材料进行DNA水平上的检测,可以不受环境影响,此方法比一般利用表现性状的聚类方法更稳定可靠[20]。本研究所收集的材料来源范围广、数量大、遗传背景复杂。利用SSR标记技术,通过软件进行数据分析得出聚类分析图,可将383份玉米材料进行类群划分,大部分材料被划分在第Ⅰ类中。
本研究选择了多态性较高的26对玉米SSR引物,能够准确地反应材料间的遗传变异,但所选的SSR引物并没有均匀分布在玉米各条染色体上,使聚类结果的应用具有一定的局限性。在以后的研究中,可以筛选出多态性高的玉米引物,并且所用引物总体上能够覆盖玉米各染色体,通过增加SSR引物数对材料DNA进行扩增,扩充分子标记数据库,来提高聚类图的精确度。
本研究通过与已知遗传背景材料的比较与聚类分析,可将系谱来源不清的材料进行已知优势群的划分与归类,能够初步确定材料间的遗传关系。此种方法可以避免育种的盲目性,并且能够提高育种效率。因研究所用材料数量大,材料间遗传关系复杂,有部分材料并没有聚到我国常见的几大类群之内,此聚类结果与所选材料和所用的引物有很大关系,同时从另一方面说明所收集的玉米种质资源相比之前的研究有了明显的拓宽。
4 结论
利用26对SSR引物对383份玉米材料进行遗传多态性分析。共检测到117个等位基因变异,每个位点的等位基因数为2~8个,平均4.5个,各SSR位点表现出的多态性,表明383份玉米材料具有较丰富的遗传变异。三个引物多态性指标:等位基因数、多态性信息量和标记索引值在数值变化趋势上相一致,说明SSR标记技术具有较强的检测玉米遗传多态性的能力。根据软件处理,可知材料间的遗传相似系数在0.407~0.983之间,通过UPGMA方法进行聚类作图分析,供试材料在遗传相似系数为0.536的水平上划分为6类。研究通过聚类将一些未确定遗传背景的材料与已知的材料划分为同一类群中,初步判断了材料的遗传背景。研究结果可以为今后育种实践有目的地选择亲本,培育玉米新品种提供技术支撑。
[1] 刘希慧,刘文希,张义荣,等.利用SSR分子标记鉴定若干玉米自交系的亲缘关系[J].分子植物育种,2005,3(2):179-187.
[3] 王振华,金益,王云生.黑龙江省主要玉米杂种种质基础分析[J].东北农业大学学报,1997,28(2):119-128.
[4] 肖木辑,李明顺,李新海,等.黄淮海地区主要玉米自交系的SSR遗传多样性分析[J].玉米科学,2008,16(2):1-7.
[5] 郑得刚,李明顺,王振华,等.黑龙江省部分常用玉米自交系遗传多样性分析[J].东北农业大学学报,2006,37(1):12-17.
[6] 杜金友,靳占忠,徐兴友,等.AFLP标记在玉米种质资源鉴定中的应用[J].西北植物学报,2006,26(5):927-932.
[7] Mumm R H,Hubert L J,Dudley W J A.Classification of 148 US maize inbreds:I.Cluster analysis based on RFLPs[J].Crop Science,1994,34:852-865.
[8] 黄世全,王爱琴,戴保威.利用醇溶蛋白、盐溶蛋白和RAPD标记划分玉米自交系类群的比较研究[J].玉米科学,2006,14(2):35-39.
[9] 姜树坤,马慧,刘君,等.利用SRAP标记分析玉米遗传多样性[J].分子植物育种,2007,5(3):412-416.
[10] Smith J S C,Chin E C L,Shu H,et al.An evaluation of the utility of SSR loci as molecular markers in maize(Zea mays L.):comparisons with data from RFLPS and pedigree[J].Theoretical and Applied Genetics,1997,95:163-173.
[11] 李新海,傅骏骅,张世煌,等.利用SSR标记研究玉米自交系的遗传变异[J].中国农业科学,2000,33(2):1-9
[12] Senior M L,Murphy J P,Goodman M M,et al.Utility of SSRs for determining genetic similarities and relationships in maize using an agarose gel system[J].Crop Science,1998,38:1088-1098.
[13] 谭君,丁仲芳,孙仕贤,等.西南常用玉米自交系SSR指纹图谱构建[J].西南农业学报,2003,16(2):1-6.
[14] 夏亮,赵琦,焦雨歆,等.微卫星标记在玉米群体遗传多样性研究中的应用[J].生物技术通报,2008(4):85-88.
[15] 袁力行,傅骏骅,Warburton M,等.利用RFLP、SSR、AFLP和RAPD标记分析玉米自交系遗传多样性的比较研究[J].遗传学报,2000,27(8):725-733.
[16] Taramino G,Tingey S.Simple sequence repeats for germplasm analysis and mapping in maize[J].Genome,1996,39:277-287.
[17] 张建华,张金渝,杨晓洪,等.利用SSR标记研究云南玉米骨干自交系的亲缘关系[J].玉米科学,2007,15(3):30-35.
[18] 吴永升,黄爱花,谭华,等.利用SSR标记分析热带、亚热带玉米自交系的遗传多样性[J].玉米科学,2008,16(1):6-10.
[19] 聂永心,张丽,潘光堂,等.四川省常用玉米自交系SSR遗传多样性分析[J].分子植物育种,2005,3(1):43-51.
[20] 王日新,盖树鹏,夏连胜,等.玉米自交系亲缘关系的SSR分析[J].中国农学通报,2008,24(5):100-104.

