反相高效液相色谱法测定人乳腺癌MCF7细胞中基因组DNA甲基化水平
蔡鑫泽,乔莹,都姝妍,刘楠,陈冬,车睿超,姜奕
(中国医科大学 附属第一医院中心实验室,沈阳 110001)
反相高效液相色谱法测定人乳腺癌MCF7细胞中基因组DNA甲基化水平
蔡鑫泽,乔莹,都姝妍,刘楠,陈冬,车睿超,姜奕
(中国医科大学 附属第一医院中心实验室,沈阳 110001)
目的 利用反相高效液相色谱法检测MCF7细胞系中基因组DNA的甲基化水平。方法 采用Nano LC(75μm×15cm,5μm)和 Micro LC(1.0mm×15cm,5μm)的 C18反相色谱柱,以甲醇-醋酸铵缓冲液(pH5.0)为流动相,使用线性梯度程序,将MCF7细胞中基因组DNA水解产物进行分离,两种体系流速分别是300nl/min和40μl/min,在273nm波长处检测,利用外标法分别检测DNA中脱氧胞嘧啶(dC)和甲基化脱氧胞嘧啶(5mdC)含量。结果 MCF7基因组DNA的水解产物,经反相色谱分离得到的峰图有较好的准确性与重复性;DNA水解后的产物为弱保留化合物,且Micro LC分离效果优于Nano LC;乳腺癌细胞MCF7甲基化水平为19.3%,高于正常细胞中基因组DNA甲基化水平。结论 成功的将MCF7细胞中基因组DNA水解产物经反相色谱分离;使用Micro LC分离DNA水解后的弱保留产物较佳;MCF7细胞系基因组DNA甲基化水平高于正常细胞。
反相高效液相色谱法;基因组;甲基化水平;乳腺癌
植物和动物体内基因组DNA广泛的甲基化,已被作为其不同生理条件下表观作用的一个标识[1]。常用的分析方法主要是配有紫外检测的高效液相色谱(HPLC)和毛细管电泳(CE)[2],HPLC结合电喷雾质谱(ESI-MS)联用技术也越来越常用,目前反相高效液相色谱技术被公认为检测核苷酸、碱基的理想方法[3]。
近年来的研究表明,DNA的甲基化水平与肿瘤的发生发展密切相关[4],因此甲基化的程度与状态为临床肿瘤等疾病的诊疗提供了可信的科学数据。本研究利用反相高效液相色谱法对乳腺癌细胞MCF7基因组DNA的甲基化水平进行检测,建立研究DNA甲基化的色谱技术平台,为进一步的研究奠定实验基础。
1 材料与方法
1.1 仪器与试剂
戴安公司Ultimate3000高效液相色谱仪(美国Dionex公司,包括WPS-3000自动进样阀、VWD-3400RS可调波长紫外检测器、FLM-3100柱温箱、DGP-3600M压力泵),溶剂过滤系统(美国Millipore公司)、安瓿瓶(美国Agilent公司)。
碱基标准品(购自美国Sigma公司):脱氧胞嘧啶(dC)和甲基化脱氧胞嘧啶(5mdC);甲醇为色谱纯(德国Merck公司);实验用水为Milli-Q超纯水;醋酸铵、核酸酶P1、DNA酶Ⅰ、RNA酶A购自美国Sigma公司;碱性磷酸酶购自美国NEB公司。
1.2 方法
1.2.1 DNA的提取与水解:体外培养乳腺癌MCF7细胞的DNA采用QIAamp DNABlood Mini Kit提取试剂盒(德国Qiagen公司)提取后,用终浓度为10μg/ml的RNA酶A去除含有的RNA;DNA水解过程参照已有文献[5]的方法:10μg DNA溶于10μl Tris-Cl(25mmol/L,pH8.0),10U的 DNA酶Ⅰ在 37℃处理1h,再100℃变性3min,迅速于冰上冷却10min,加入1μl核酸酶P1后放入37℃水浴中16h,然后加入碱性磷酸酶1.5U处理2h,于-20℃保存。1.2.2 高效液相色谱条件:戴安Ultimate3000型HPLC系统,色谱柱为戴安公司 Nano LC(75μm×15cm,5μm 粒径)和 Micro LC(1.0mm×15cm,5μm 粒径)的C18反相柱。流动相:A,去离子水;B,50mmol/L醋酸铵(pH5.0);C,甲醇。用于分离的梯度程序:0~20min 0~30%C(线性梯度),15%B;20~25min 30%C,15%B;25~50min 15%B,85%A(V/V)。柱温30℃。检测波长270nm。流速:Nano LC体系流速为 300nl/min,Micro LC体系流速为 40μl/min。进样量 0.2μl。
1.2.3 标准曲线的绘制:根据人类基因组DNA甲基化水平约为5%[6],因此将购买的标准品5mdC和dC按照质量浓度比1∶20配制。其中5mdC质量浓度系列为:1、2、4、8ng/μl,dC质量浓度系列为:20、40、80、160ng/μl。利用优化的实验条件,进样量为0.2μl。
1.3 数据处理
数据处理软件为Chromeleon Version 6.80(美国Dionex公司);采用外标法定量,以W(5mdC)/[W(dC)+W(5mdC)]100%(单位为 ng/ng DNA)的方式来表征DNA甲基化水平。根据标准品的标准曲线计算出样品中dC和5mdC各自的含量,然后通过公式:(5mdC)%=W(5mdC)/[W(dC)+W(5mdC)]100%,得出MCF7细胞系DNA甲基化水平。
2 结果
2.1 反相色谱分离条件的优化
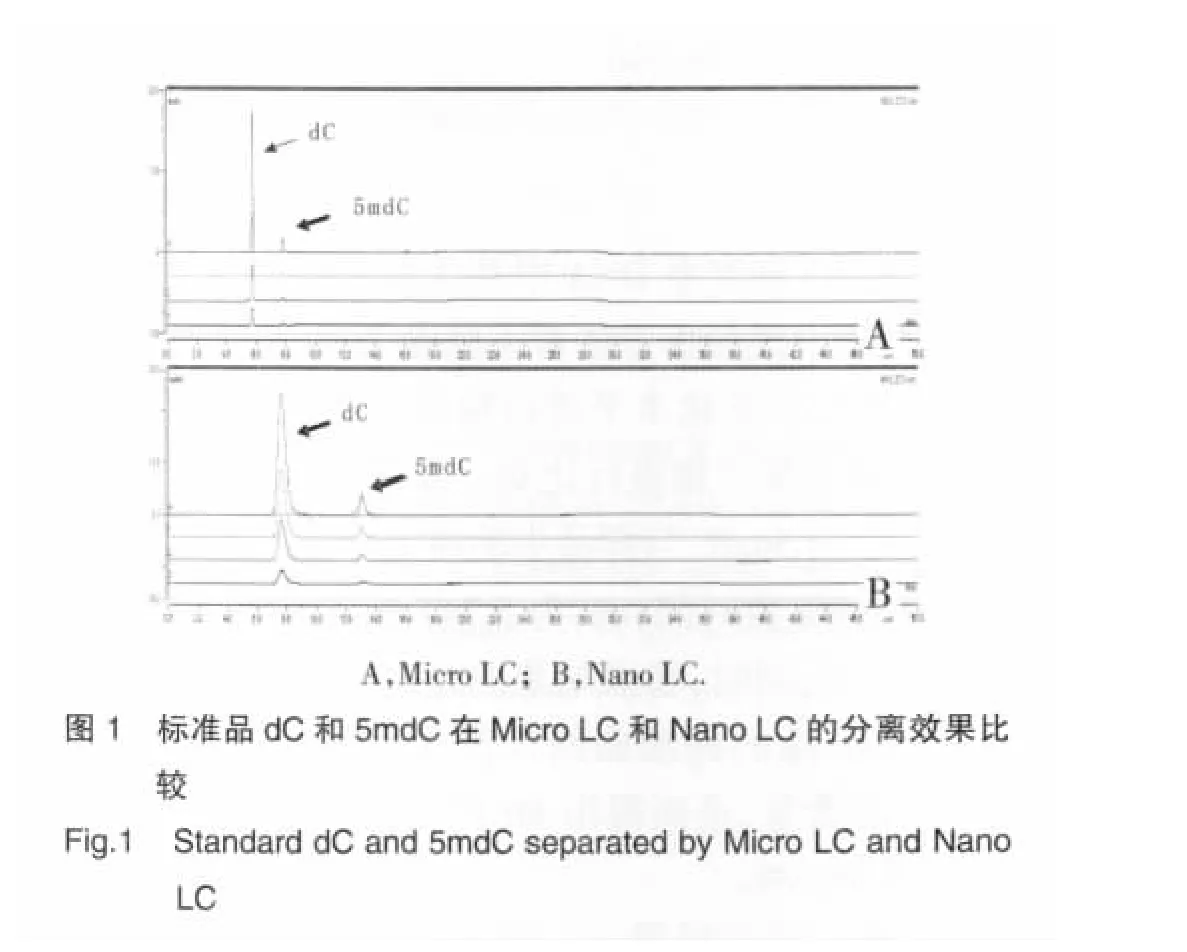
实验用的两个C18反相柱分别是戴安公司的Nano和Micro柱,即柱子的直径为75μm和1.0mm,已经测试合格,并具有较佳的柱效。对于流动相的选择,本研究用醋酸铵-甲醇、酸化甲醇-甲酸、醋酸铵-乙腈溶液等多种流动相进行试验,结果表明以醋酸铵-甲醇溶液得到的分离效果最好。将已稀释成不同浓度的标准品dC和5mdC为样品进行分离条件的优化,当线性梯度洗脱条件为:0~20min,0~30%甲醇,7.5%醋酸铵时,dC与5mdC可以达到完全分离(图1)。可以看出,采用Micro LC体系的分离图,峰宽较窄,分辨率较高,分离效果明显优于Nano LC体系,故以后实验采用Micro LC体系。
2.2 线性关系的考察
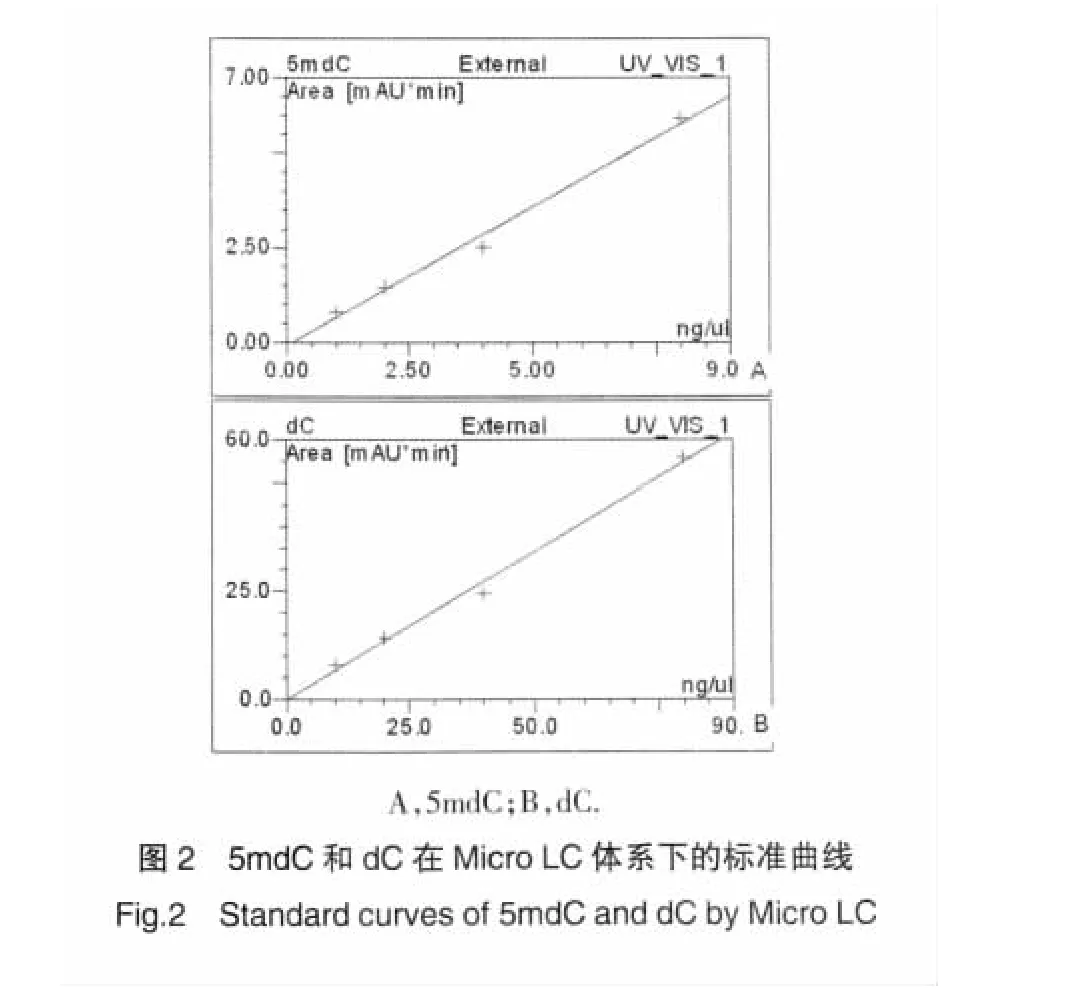
将5mdC和dC的母液按1∶20的质量比混合,再逐级稀释至浓度分别为 1、2、4、8ng/μl和 20、40、80、160ng/μl。实验采用 Micro LC体系,使用的定量方法为外标法。将各稀释液分别进样0.2μl进行检测,以浓度为横坐标,积分面积为纵坐标绘制标准曲线(图2),相关系数r分别为99.82%和99.89%。
2.3 方法的准确性与重复性
实验采用人乳腺癌细胞MCF7基因组DNA水解产物作为质量控制样品,对本方法的准确性和重复性进行了研究。取平行样品在同一天内不同时间以及同一样品在不同天数进行重复测试,结果表明5mdC和dC测定值平行样相对标准偏差、样品的日内偏差和日间偏差(RSD)范围在2%~5%(表1),表明本方法具有良好的重复性。

表1方法准确性和重复性实验结果Ta b.1Ac c u r a c y a n d r e p e a t a b i l i t y o f t h e me t h o d Result 5mdC(ng/μl) RSD%(n=4) dC(ng/μl) RSD%(n=4)Parallel sample 1.698±0.02 2.35 7.075±0.06 2.17Intra-day 1.665±0.03 3.38 7.112±0.025 3.21Inter-day 1.710±0.053 5.10 7.230±0.04 4.86RSD,relative standard deviation.Plus-minus values are means±SD.
2.4 MCF7细胞系DNA甲基化水平检测
利用上述分析方法对人乳腺癌MCF7细胞内基因组DNA甲基化水平进行检测,将标准品与样品MCF7的水解产物进行比对,发现标准品的两个峰图即dC与5mdC与样品中的两个峰匹配完好,可得到各自的保留时间:dC为5.747min,5mdC为7.753min(图3);同时,将该结果图整理得到表2。最后,根据标准品的标准曲线计算得出样品中的dC和5mdC的含量,进而得出MCF7细胞系DNA甲基化水平为19.3%。
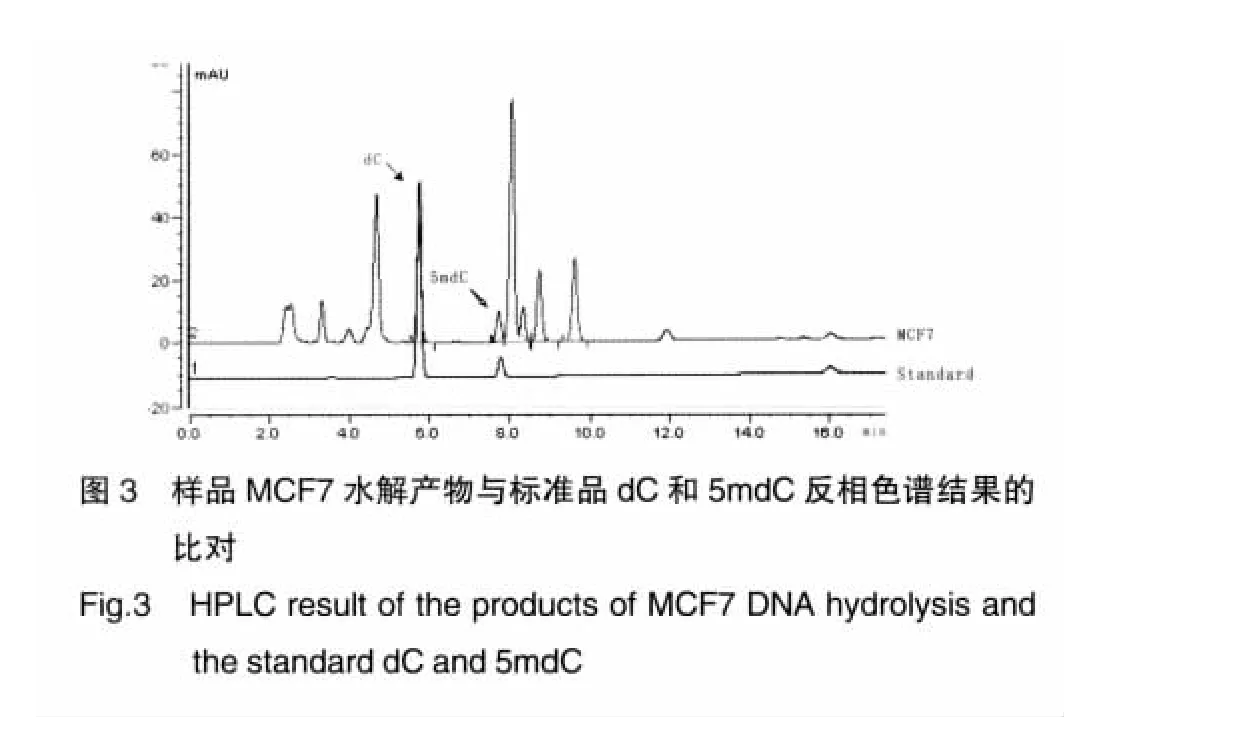

表2整理得到的样品峰图信息表Ta b.2In f o r ma t i o n s h e e t o f s a mp l e p e a k s No.Retention time(min)Peak NameHeight(mAU)Area(mAU·min)Relative area(%)Amoun(ng/μl)15.75 dC 37.778 4.744 18.46 7.08527.75 5mdC 9.018 1.162 4.52 1.692
3 讨论
近几年,表观遗传学改变的研究取得了长足进展,成为肿瘤临床基础研究的一个热点领域。表观遗传学是研究非DNA序列变化引起的基因表达的改变,是研究表达在时间和空间上的调控问题,其中最主要的研究内容就是DNA甲基化[7]。
DNA甲基化修饰是指在DNA甲基转移酶作用下,以S-腺苷甲硫氨酸为甲基供体,将甲基基团转移到胞嘧啶和鸟嘌呤(CpG)二核苷酸的胞嘧啶5位碳原子上,癌基因多为不充分甲基化,导致表达异常;抑癌基因多为过度甲基化,导致表达不足[8~10]。对肿瘤DNA甲基化的研究,应根据目的选择合适的方法,明确是研究整体水平的甲基化还是研究特定位点的甲基化。对DNA中CpG岛的甲基化分析涉及多种分子生物学方法,如甲基化敏感的限制性内切酶方法、NaHSO3法(包括BSP测序法、甲基化特异性PCR、荧光定量法)、芯片技术等。对DNA整体甲基化水平的研究,主要靠的是高效液相色谱、质谱等精密仪器[11]。可见,研究基因组DNA甲基化的方法多种多样,一方面说明甲基化研究难度大,另一方面也说明这些方法都存在着局限性。
本次研究通过采用反相色谱技术对乳腺癌细胞MCF7中基因组DNA甲基化水平的检测,成功将DNA水解产物进行分离、定量。通过研究发现,DNA水解产物对于C18反相柱为弱保留化合物,流动相选用甲醇-醋酸铵缓冲液较为合适;比较Micro LC和Nano LC的分离图谱,虽然二者都足以达到分离效果,但前者可以得到较窄的峰宽图,因此其分离效果要优于Nano LC;对MCF7细胞系甲基化的总体水平定量检测发现,反相HPLC可以得到较佳的分析重现性,样品定量结果表明,MCF7基因组甲基化水平高于正常细胞,推测该细胞系中的部分抑癌基因甲基化程度过度异常,导致抑癌基因表达下降,使肿瘤发生。
总之,建立基于高效液相色谱(HPLC)技术的DNA甲基化研究平台,可以为今后进一步研究某种基因甲基化的特定位点奠定基础,此外,深入研究DNA甲基化与基因表达的关系及作用机制为肿瘤的早期诊断与临床治疗提供了新的思路。
[1]Fieldes MA,Schaeffer SM,Krech MJ,et al.DNAhypomethylation in 5-azacytidine-induced early-flowering lines of flax [J].Theor Appl Genet,2005,111(1):136-149.
[2]Johnston JW,Harding K,Bremmer DH,et al.HPLCstudy of plant DNAmethylation:Astudy of critical methodological factors[J].Plant Physiol Biochem,2005,43(9):844-853.
[3]王超云,胡凤祖,师治贤.反相高效液相色谱法测定大鼠肝组织中 DNA的碱基含量[J].色谱,2002,20(4):348-349.
[4]Martens JW,Margossian AL,Schmitt M,et al.DNAmethylation as a biomarker in breast cancer [J].Future Oncol,2009,5(8):1245-1256.
[5]Liu ZF,Wu JJ,Xie ZL,et al.Quantification of regional DNAmethylation by liquid chromatography/tandem mass spectrometry[J].Anal Biochem,2009,391(2):106-113.
[6]Friso S,Choi SW,Dolnikowski GG,et al.Amethod to assess genomic DNAmethylation using high-performance liquid chromatography/electrospray ionization mass spectrometry [J].Anal Chem,2002,74(17):4526-4531.
[7]朱新江,孟春风,彭过,等.5-Aza-dC和TSA对胃癌细胞系p16和hMLH-1基因甲基化水平及表达的影响 [J].世界华人消化杂志,2008,16(17):1837-1841.
[8]Van der Auwera I,Yu W,Suo L,et al.Array-based DNAmethylation profiling for breast cancer subtype discrimination [J].PLoSOne,2010,5(9):e12616.
[9]张晔,曲秀娟,刘云鹏,等.3种胃癌细胞株中GSTP1基因表达及其启动子区甲基化状态的研究[J].中国医科大学学报,2008,37(6):724-726.
[10]Chuang CK,Chu DC,Tzou RD,et al.Hypermethylation of the CpGislands in the promoter region flanking GSTP1gene is a potential plasma DNAbiomarker for detecting prostate carcinoma[J].Cancer Detect Prev,2007,31(1):59-63.
[11]孙贝娜.DNA甲基化检测方法的研究进展[J].生命科学仪器,2009,7(4):11-14.
(编辑 裘孝琦,英文编辑 陈 姜)
Genomic DNAMethylation in Human Breast Cancer Cell Line MCF7Detected by Reversed-phase High Performance Liquid Chromatography
CAIXin-ze,QIAOYing,DUShu-yan,LIUNan,CHENDong,CHERui-chao,JIANGYi
(Central Laboratory,The First Hospital,China Medical University,Shenyang 110001,China)
ObjectiveTo detect the genomic DNAmethylation in human breast cancer cell line MCF7with reversed-phase high performance liquid chromatography(HPLC).MethodsThe products of genomic DNAhydrolysis in MCF7cells were chromatographed on Nano LC(75μm×15cm,5μm)and Micro LC(1.0mm×15cm,5μm)with ultraviolet detection at 270nm,and eluted by the mobile phase of MeOH-NH4Ac (pH5.0)at the flow rates of 300nl/min and 40μl/min under the condition of linear gradient program.The amounts of 2′-deoxycytidine (dC)and 5-methyl-2′-deoxycytidine (5mdC)were determined by external standard method.ResultsThe products of DNAhydrolysis were successfully separated by reversed-phase HPLCwith good repeatability and accuracy.The products were weakly retained and separated better by Micro LC.The level of global DNAmethylation in MCF7cells was 19.3%,which was higher than that in normal cells.ConclusionThe products of DNAhydrolysis in MCF7cells could be successfully separated by reversed-phase HPLC.Micro LCis superior to Nano LCin separating weak retention compounds of hydrolysis products.The level of DNAmethylation is higher in MCF7cells than in normal cells.
reversed-phase high performance liquid chromatography;genome;methylation level;breast cancer
O657.72;Q523
A
0258-4646(2010)11-0898-03
国家自然科学基金资助项目(30571701)
蔡鑫泽(1982-),男,助教,硕士.
姜奕,E-mail:yjiang58@hotmail.com
2010-07-30

