抗病毒固有免疫分子TRIM22 C端SPRY结构域对其转录、翻译及亚细胞定位的影响
高波,段志坚,徐薇,熊思东
复旦大学免疫生物研究所,复旦大学上海医学院免疫学系,上海200032
三结构域(tripartite motif,TRIM)蛋白家族均含有1个RING(really interesting new gene)结构域、1或2个B-box结构域和1个卷曲螺旋结构域(coiled-coil)。60%的TRIM蛋白在C端还有SPRY结构域[1]。TRIM家族蛋白与机体的多种生理功能有关,如细胞增殖、分化、发育、凋亡等[1,2]。尤为值得关注的是,近年研究发现多种TRIM家族蛋白在抗病毒免疫中起重要作用[3-7],提示TRIM家族蛋白是一类广泛存在的、新型的抗病毒固有免疫分子[8,9]。TRIM22是干扰素诱导蛋白,多项研究表明它可有效抑制人类免疫缺陷病毒1型(human immunodeficiency virus type 1, HIV-1)的复制,是干扰素抑制HIV-1复制的关键效应分子[5,10,11]。另外,在乙型肝炎病毒(hepatitis B virus,HBV)及丙型肝炎病毒(hepatitis C virus,HCV)感染猩猩的肝基因组学研究中发现,TRIM22与HBV及HCV的清除均有关[12],说明TRIM22亦可能在肝脏的抗病毒免疫中发挥作用。TRIM22的C端含有SPRY结构域。新近研究发现SPRY结构域在TRIM22的分子进化过程中起重要作用,并很可能与TRIM22对病毒的识别有关[13]。为研究SPRY结构域在TRIM22抗病毒免疫中的作用,本研究构建TRIM22的SPRY结构域缺失突变体,并对该突变体的功能进行初步研究。结果发现,SPRY结构域缺失突变体在转录及翻译水平上与野生型并无差异,但它完全丧失了核定位能力。
1 材料和方法
1.1 细胞系
人高分化的肝癌细胞系HepG2细胞株为本研究室保存。
1.2 试剂
真核表达质粒pcDNA3.1、TRIzol试剂购自Invitrogen公司。两步法反转录-聚合酶链反应(reverse transcriptase-polymerase chain reaction,RT-PCR)试剂盒为Fermentas公司产品。DNA聚合酶LaTaq、T4 DNA连接酶和限制性内切酶NheⅠ、HindⅢ均为TaKaRa公司产品。引物合成由上海生工生物工程技术服务有限公司完成。胶回收试剂盒购自上海华舜公司。质粒提取使用Qiagen公司的质粒中量抽提试剂盒。鼠抗c-myc抗体(克隆号9E10)、鼠抗人GAPDH抗体及FITC标记的羊抗鼠IgG二抗均购自Santa Cruz公司。辣根过氧化物酶(horseradish peroxidase,HRP)标记羊抗鼠IgG抗体购自上海康成生物工程有限公司。硝酸纤维素膜和ECL化学发光试剂盒为Amersham公司产品。
1.3 带Myc表位的TRIM22-ΔSPRY真核表达质粒的构建
以本实验室先期构建的野生型TRIM22真核表达质粒[14]为模板,PCR扩增C端带Myc表位的SPRY结构域缺失的TRIM22基因,上游引物:5′-CAGGCTAGCATGGATTTCTCAGTAAAGGTAGAC-3′(NheⅠ),下游引物:5′-CTGAAGCTTTCACAGGTCTTCTTCAGAGATCAGTTTCTGTTCA
GGTCAGTTAGCCTTGCCT-3′(HindⅢ),下划线为酶切位点序列。对PCR产物和pcDNA3.1分别用NheⅠ和HindⅢ进行双酶切后琼脂糖凝胶电泳回收目的片段,用T4 DNA连接酶在16 ℃将目的基因片段插入pcDNA3.1。连接产物转化DH5α感受态细胞,挑取可疑阳性克隆,进行菌落PCR及双酶切鉴定,并将阳性克隆送测序。
1.4 细胞培养及转染
HepG2细胞在37 ℃、5% CO2条件下培养于含10%胎牛血清、100 u/ml青霉素、100 u/ml链霉素及2 mmol/L谷氨酰胺的DMEM培养基中。利用磷酸钙沉淀法将目的质粒转染对数生长期的HepG2细胞。转染16 h后,用磷酸缓冲液(phosphate-buffered saline,PBS)洗2遍,然后加入新鲜培养基。
1.5 RT-PCR
收集HepG2细胞,用TRIzol试剂抽提细胞总RNA,并用DNaseⅠ处理1 h以清除残余DNA。取总RNA 1 μg进行反转录。反应条件:72 ℃ 5 min,42 ℃ 60 min,72 ℃ 5 min。采用Oligo(dT)18引物进行反转录,获得cDNA。PCR反应条件:94 ℃预变性4 min;94 ℃ 30 s,55 ℃退火30 s,72 ℃延伸90 s,共30个循环;然后72 ℃ 10 min终止反应。PCR产物经1%琼脂糖凝胶电泳后,在凝胶成像仪上拍摄成像并进行灰度扫描。
1.6 蛋白质免疫印迹法
收集转染后的HepG2细胞,置细胞裂解液中,经12%十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)分离。电泳后,采用湿法(300 mA,90 min)转移至硝酸纤维素膜。经封闭液(5%脱脂奶粉-TBST)封闭2 h后,分别加入1∶500稀释的鼠抗c-myc单克隆抗体和1∶1 000稀释的鼠抗人GAPDH单克隆抗体(用TBST稀释),4 ℃过夜,漂洗3次,每次10 min。加入羊抗鼠IgG-HRP(TBST稀释1∶500),室温作用2 h,漂洗3次,每次10 min。用ECL化学发光法成像和显影。
1.7 间接免疫荧光分析
将HepG2以1×106个/ml铺于放有玻璃盖片的6孔细胞培养板中,培养48 h后通过磷酸钙沉淀法将TRIM22真核表达质粒转染HepG2细胞。48 h后将玻璃盖片取出,用4%中性甲醛固定30 min,PBS洗3次;0.2% Triton X-100冰上透化10 min,PBS洗3次;1%牛血清白蛋白(bovine serum albumin,BSA)封闭1 h,PBS洗3次;与1∶500稀释的鼠抗c-myc单克隆抗体4 ℃孵育过夜,PBS洗3次;与1∶1 000稀释的FITC标记的羊抗鼠二抗室温避光孵育2 h,PBS洗3次;与1∶500稀释的DAPI避光作用10 min,PBS洗3次;甘油封片后镜检、拍照。
2 结果
2.1 TRIM22 SPRY结构域缺失突变体的构建与鉴定
为研究SPRY结构域在TRIM22抗病毒过程中的作用,以本实验室先期构建的野生型TRIM22真核表达质粒为模板,PCR扩增C端带Myc表位、SPRY结构域缺失的基因片段,再将其克隆入真核表达质粒pcDNA3.1中。SPRY结构域缺失突变体(ΔSPRY)的菌落PCR和酶切鉴定结果进一步证实缺失SPRY结构域的TRIM22真核表达质粒构建成功、读码框架正确无误(图1)。
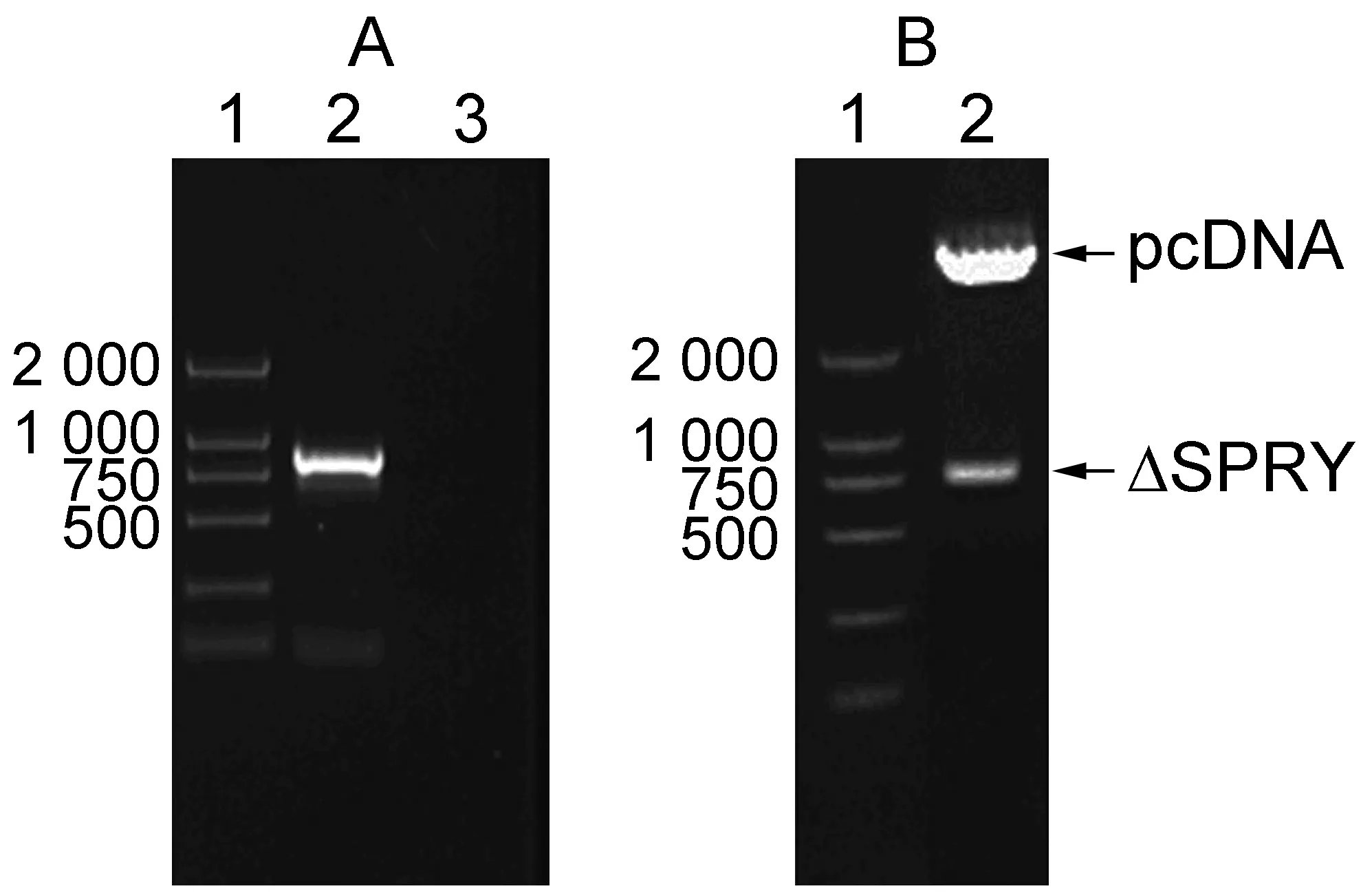
A: Identification of plasmid expressing TRIM22-ΔSPRY by colony PCR. 1, DNA marker; 2, TRIM22-ΔSPRY PCR products; 3, negative control. B: Identification of plasmid expressing TRIM22-ΔSPRY by restrict endonuclease digestion. 1, DNA marker; 2, pcDNA/TRIM22-ΔSPRY digested withNheⅠandHindⅢ.
图1TRIM22SPRY结构域突变体的构建与鉴定
Fig.1ConstructionofeukaryoticexpressionvectorforTRIM22-ΔSPRY
2.2 TRIM22的SPRY结构域对其转录的影响
为研究TRIM22的SPRY结构域对其转录的影响,将表达野生型TRIM22和SPRY结构域缺失的TRIM22真核表达质粒分别转染HepG2细胞,转染48 h后通过半定量RT-PCR检测mRNA表达水平。结果发现,突变型TRIM22在转录水平上与野生型TRIM22没有明显差异(图2)。

图2TRIM22的SPRY结构域对其转录的影响
Fig.2TheeffectoftheTRIM22SPRYdomainonitstrans-cription
2.3 TRIM22的SPRY结构域对其翻译的影响
为研究TRIM22的SPRY结构域对其翻译的影响,将带Myc表位的野生型和SPRY结构域缺失突变的TRIM22真核表达质粒转染HepG2细胞,转染48 h后利用抗Myc抗体进行蛋白质免疫印迹法检测。在约55×103和33×103处分别检测出野生型TRIM22和SPRY结构域缺失突变型TRIM22的表达,与预期的相对分子质量(Mr)相符,但两者在蛋白表达水平上并无明显差异(图3)。

图3TRIM22的SPRY结构域对其翻译的影响
Fig.3TheeffectofTRIM22SPRYdomainonitstranslation
2.4 TRIM22的SPRY结构域对其亚细胞定位的影响
为研究SPRY结构域对TRIM22在HepG2细胞中亚细胞定位的影响,将带Myc表位的野生型和SPRY结构域缺失突变的TRIM22真核表达质粒转染HepG2细胞,然后利用抗Myc抗体进行免疫荧光定位。结果发现野生型TRIM22主要定位在HepG2细胞核中,与我们以前所报道的在COS-7细胞中的结果相符[14],而SPRY结构域缺失的TRIM22则弥散分布在细胞质中(图4),提示SPRY结构域对TRIM22的亚细胞定位起关键作用。

A: Wild type TRIM22. B: SPRY domain deleted TRIM22.
图4TRIM22的SPRY结构域对其亚细胞定位的影响(×400)
Fig.4TheeffectofTRIM22SPRYonitssubcellularlocalization(×400)
3 讨论
TRIM蛋白属于RING蛋白家族,迄今已发现近70个TRIM家族成员都含有相似的RBCC结构序列,从蛋白肽链的N端到C端依次包含1个RING结构域、1或2个B-box结构域和1个卷曲螺旋结构域。60%的TRIM家族分子C端还有1个SPRY结构域[1]。许多TRIM家族分子在抗病毒固有免疫中起重要作用,研究最为深入的是TRIM5α,具有广泛的抗反转录病毒作用。恒河猴的TRIM5α分子可通过迅速降解病毒衣壳蛋白来抑制HIV-1的复制,从而抵抗HIV感染[3]。与TRIM5α在结构和功能上最为相似的是TRIM22。TRIM22基因的染色体定位于11p15,与TRIM5紧相邻,两者之间的距离仅为4.8 kb[15]。新近研究发现,TRIM22和TRIM5都表现出快速适应进化,即达尔文正向选择(positive selection)的特点,但由于两者具有极大的遗传相似性,所以不能同时进行达尔文正向选择[13]。多项研究表明,TRIM22亦可有效抑制HIV-1的复制,并与多种病毒感染有关,但TRIM22的抗病毒机制迄今还不清楚[5,10,11]。
60%的TRIM分子,包括TRIM22,在C端具有SPRY结构域。SPRY结构域的功能多样,与细胞因子信号转导的调节、RNA代谢及细胞内钙离子的释放等有关。在抗病毒免疫中,SPRY结构域可能与抗病毒特异性有关[16]。新近研究亦发现,TRIM5α和TRIM22分子进化的正选择氨基酸位点均位于SPRY结构域,提示SPRY结构域可能与TRIM22对病毒的识别有关[13]。为研究SPRY结构域在TRIM22抗病毒过程中发挥的作用,本研究对TRIM22的SPRY结构域进行缺失突变。由于基因突变可能影响其表达水平,我们将SPRY结构域缺失突变型和野生型TRIM22真核表达质粒转染HepG2细胞,然后比较其转录和翻译表达水平。结果显示,SPRY结构域的缺失并不影响TRIM22的转录和翻译水平,这为后续研究中排除基因表达水平差异的影响以及研究SPRY结构域在TRIM22抗病毒过程中的作用奠定基础。
研究表明多种TRIM分子以三聚体形式存在,TRIM蛋白的这种自我寡聚化能力有利于形成高Mr的复合物,从而构成显著的亚细胞结构,存在于胞核或胞质中[1],而TRIM分子的亚细胞定位往往与其功能的发挥密切相关。我们前期的研究发现,TRIM22主要定位于细胞核中,与其作为转录因子的功能相符。为研究SPRY结构域在TRIM22的亚细胞定位中所起的作用,我们将SPRY结构域缺失突变型和野生型TRIM22真核表达质粒转染HepG2细胞,利用免疫荧光技术进行亚细胞定位研究。结果发现,与定位在细胞核中的野生型TRIM22不同,SPRY结构域缺失突变体弥散分布在细胞质中,提示SPRY结构域对TRIM22的核内定位起关键作用,为进一步阐明该结构域在TRIM22抗病毒免疫中的作用和机制提供有益的启示。
[1] Reymond A, Meroni G, Fantozzi A, Merla G, Cairo S, Luzi L, Riganelli D, Zanaria E, Messali S, Cainarca S, Guffanti A, Minucci S, Pelicci PG, Ballabio A. The tripartite motif family identifies cell compartments [J]. EMBO J, 2001, 20(9): 2140-2151.
[2] Meroni G, Diez-Roux G. TRIM/RBCC, a novel class of ‘single protein RING finger’ E3 ubiquitin ligases [J]. Bioessays, 2005, 27(11): 1147-1157.
[3] Stremlau M, Owens CM, Perron MJ, Kiessling M, Autissier P, Sodroski J. The cytoplasmic body component TRIM5 alpha restricts HIV-1 infection in Old World monkeys [J]. Nature, 2004, 427(6977): 848-853.
[4] Regad T, Saib A, Lallemand-Breitenbach V, Pandolfi PP, de Thé H, Chelbi-Alix MK. PML mediates the interferon-induced antiviral state against a complex retrovirus via its association with the viral transactivator [J]. EMBO J, 2001, 20(13): 3495-3505.
[5] Barr SD, Smiley JR, Bushman FD. The interferon response inhibits HIV particle production by induction of TRIM22 [J]. PLoS Pathog, 2008, 4(2): e1000007.
[6] Gack MU, Shin YC, Joo CH, Urano T, Liang C, Sun L, Takeuchi O, Akira S, Chen Z, Inoue S, Jung JU. TRIM25 RING-finger E3 ubiquitin ligase is essential for RIG-I-mediated antiviral activity [J]. Nature, 2007, 446(7138): 916-920.
[7] Wolf D, Goff SP. TRIM28 mediates primer binding site-targeted silencing of murine leukemia virus in embryonic cells [J]. Cell, 2007, 131(1): 46-57.
[8] Nisole S, Stoye JP, Saïb A. TRIM family proteins: retroviral restriction and antiviral defence [J]. Nat Rev Microbiol, 2005, 3(10): 799-808.
[9] Ozato K, Shin DM, Chang TH, Morse HC 3rd. TRIM family proteins and their emerging roles in innate immunity [J]. Nat Rev Immunol, 2008 8(11): 849-860.
[10] Tissot C, Mechti N. Molecular cloning of a new interferon-induced factor that represses human immunodeficiency virus type 1 long terminal repeat expression [J]. J Biol Chem, 1995, 270(25): 14891-14898.
[11] Bouazzaoui A, Kreutz M, Eisert V, Dinauer N, Heinzelmann A, Hallenberger S, Strayle J, Walker R, Rübsamen-Waigmann H, Andreesen R, von Briesen H. Stimulated trans-acting factor of 50 kDa (Staf50) inhibits HIV-1 replication in human monocyte-derived macrophages [J]. Virology, 2006, 356(1-2): 79-94.
[12] Wieland S, Thimme R, Purcell RH, Chisari FV. Genomic analysis of the host response to hepatitis B virus infection [J]. Proc Nat Acad Sci USA, 2004, 101(17): 6669-6674.
[13] Sawyer SL, Emerman M, Malik HS. Discordant evolution of the adjacent antiretroviral genes TRIM22 and TRIM5 in mammals [J]. PLoS Pathog, 2007, 3(12): e197.
[14] Duan Z, Gao B, Xu W, Xiong S. Identification of TRIM22 as a RING finger E3 ubiquitin ligase [J]. Biochem Biophys Res Commun, 2008, 374(3): 502-506.
[15] Tissot C, Taviaux SA, Diriong S, Mechti N. Localization of Staf50, a member of the Ring finger family, to 11p15 by fluorescence in situ hybridization [J]. Genomics, 1996, 34(1): 151-153.
[16] Yap MW, Nisole S, Stoye JP. A single amino acid change in the SPRY domain of human Trim5 alpha leads to HIV-1 restriction [J]. Curr Biol, 2005, 15(1): 73-78.

