酵母菌SRAP-PCR反应体系的优化及遗传多样性分析
蒋冬花,徐晓波,叶莹莹,嵇 豪,蔡琪敏
(浙江师范大学化学与生命科学学院,浙江金华 321004)
酵母菌是一类重要的微生物,对其分类与鉴定是人类开发利用的基础。传统的酵母菌鉴定方法所依赖的形态和生理生化特征均是表型性状,会随着环境的变化有所改变,这样就会造成菌株鉴定结果的不稳定性。随着DNA分子标记技术的引入给微生物分类鉴定带来了积极的推动作用,使分类鉴定工作由一般的表型特征鉴定深化到分子水平和遗传特征的鉴定。SRAP是一种基于PCR技术的新的DNA分子标记,由Li和Quiros[1]于2001年提出,具有简便、稳定、中等产率和容易得到选择条带序列的特点。SRAP利用其独特的引物设计对基因组的开放阅读框(ORFs)进行特异扩增,上游引物长17 bp,对外显子进行特异扩增,下游引物长18 bp,对内含子区域、启动子区域进行特异性扩增,因个体不同以及物种的内含子、启动子和间隔序列的不同,产生基于内含子和外显子的SRAP多态性[2]。用于SRAP实验的引物包括1个含17个碱基的正向引物(F-pr imer)和1个含18个碱基的反向引物(R-primer),正向引物含有一段14个碱基的核心序列,5′端前10个为填充序列TGAGTCCAAA,无任何特异组成,接着为CCGG序列,随后3′端为3个不同的选择性碱基,与相同的核心序列组成一套正向引物。反向引物的5′端前11个为填充序列GACTGCGTACG,接着为AATT,随后3′端同样为3个不同的选择性碱基[3-4]。SRAP标记已经在马铃薯、水稻、苹果、柑橘类果树、樱桃、梅子、油菜、大蒜、莴苣和棉花等[5-9]植物研究中得到了应用,具有简便、稳定、中等产率的特点,在基因组中分布均匀,适合基因定位、基因克隆等分子生物学研究。但是在真菌中的应用很少,尤其是在酵母菌中的应用未见报道。本研究应用SRAP标记对实验室保藏的19株待鉴定的酵母进行分子标记鉴定,检测这种分子标记技术用于酵母菌菌种鉴别的可行性,为酵母菌的遗传分析和分类研究提供DNA水平的证据。
1 材料与方法
1.1 材料
1.1.1 菌株 从梨、苹果、葡萄、猕猴桃等水果表皮不同部位,果园土、泡菜、酒曲等场所分离筛选纯化得到18株酵母菌菌株,分别编号为Y-2~Y-19;浙大紫金港微生物实验室提供标准啤酒酵母(Saccharom yces cerevisiae)菌株作为参照菌株,并将其编号为Y-1。
1.1.2 试剂 用于SRAP-PCR反应的dNTPs、TaqDNA聚合酶及DL 2000TMDNA Marker均购自宝生物工程(大连)有限公司。本实验使用的引物包括8个正向引物和11个反向引物,共组合成88个引物对,其序列见表1,引物由上海生工生物技术有限公司合成。

表1 SRAP实验的引物序列Table 1 Sequences of primers used in SRAP analysis
1.2 方法
1.2.1 基因组DNA的提取 使用T IANamp YeastDNA Kit提取酵母基因组DNA,试剂盒购于北京科百奥生物科技有限责任公司。用0.8%的琼脂糖电泳检测DNA质量,通过紫外分光光度计测定DNA的浓度和纯度,并将DNA浓度稀释为50 ng/μL。
1.2.2 引物的筛选 通过组合8对正向引物和11对反向引物得到88对引物组合,利用这些引物组合对酵母Y-1进行扩增实验,从中筛选出带形清晰,多态性丰富的引物。扩增反应体系总体积为25μL,含50 ng模板DNA,50 ng引物,1.5 μL dNTPs(2.5 mmol/L),2.0μLMgCl2(25 mmol/L),1.0 UTaqDNA聚合酶,1×PCR缓冲液。反应程序为:94℃预变性5 min;94℃变性1 min,35℃退火1 min,72℃延伸1 min,扩增5个循环;94℃变性1 min,50℃退火1 min,72℃延伸1 min,扩增35个循环;72℃延伸10 min。
1.2.3 PCR正交设计试验[10-11]为确定PCR反应中各因素最佳水平,以酵母Y-1的DNA为模板,使用1.2.2所得结果的引物组合,采用L16(45)正交法,对影响SRAP扩增的主要因素Mg2+、dNTPs、DNA模板、引物、TaqDNA聚合酶设计5因素4水平的正交试验,以确定PCR电泳分析的最佳条件。正交设计中的16个处理重复3次,PCR反应各因素水平见表2和表3。
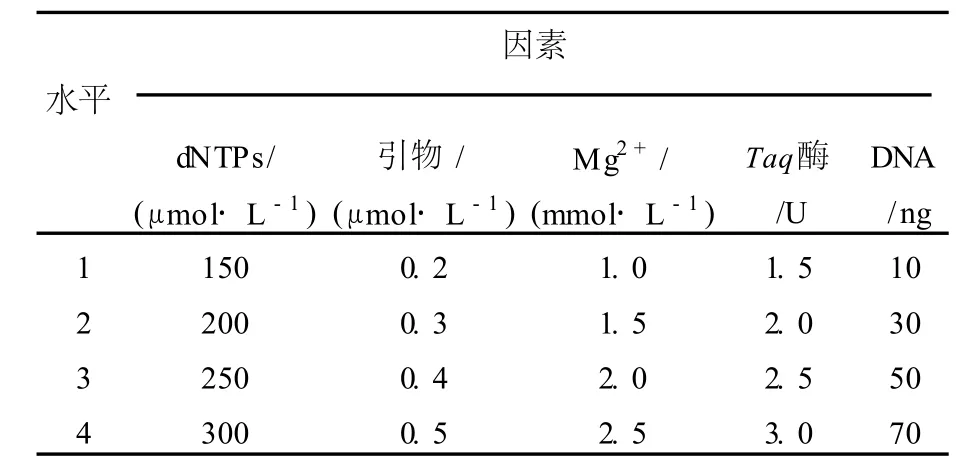
表2 SRAP-PCR体系因素-水平Table 2 SRAP-PCR factors-levels

表3 SRAP-PCR正交试验设计(L16(45))Table 3 SRAP-PCR orthogonal design(L16(45))

续表
1.2.4 DNA多样性聚类分析 在最适条件下进行PCR扩增,PCR扩增产物在琼脂糖凝胶电泳中分离获得扩增图谱,根据同一位点带的有无编成1,0矩阵输入计算机,用NTSYSpc软件中的S IMQUA I程序计算菌株间的相似系数,然后用UPG MA法进行聚类分析,构建相似聚类树状图。
2 结 果
2.1 引物筛选结果
用88对SRAP引物对酵母Y-1进行PCR扩增,其中me2-em7、me2-em10、me3-em4、me3-em6、me3-em8、me4-em2、me4-em5、me4-em9、me4-em10、me5-em7、me5-em8、me5-em10、me6-em8、me8-em5共14对引物扩增产物的条带清晰、重复性和稳定性好且多态性条带相对较多,因此选用上述14对引物进行19个供试菌株的SRAP扩增实验(图1)。

图1 部分SRAP引物筛选结果Fig.1 Result of primers screening for SRAP
2.2 PCR正交试验设计的结果分析
按表3设计的16个处理进行PCR试验,电泳检测结果见图2,使用正交设计助手Ⅱ进行结果分析,极差分析结果见表4。依据琼脂糖电泳条带的强弱和杂带的多少进行直观分析。PCR的扩增结果是dNTPs、引物、DNA模板、Mg2+和Taq酶等因素综合作用的结果。从表4中可以看出,极差R值越大,说明该因素对PCR扩增结果影响越大,因此5个因素水平变化对SRAP-PCR扩增结果的影响从大到小依次为dNTPs浓度>引物浓度>DNA模板浓度>Mg2+浓度>Taq酶浓度,即dNTPs浓度的影响最为显著。
2.2.1 dNTPs浓度对于PCR扩增结果的影响dNTPs浓度在150μmol/L时扩增效果最好,之后呈现直线下降趋势,每2个水平之间差异都达到显著水平。由关系曲线的波动幅度可知(图3),dNTPs浓度对PCR结果影响较大。本实验选择峰值150μmol/L为最佳反应水平。
2.2.2 引物浓度对PCR扩增结果的影响 引物浓度为0.2μmol/L和0.3μmol/L在水平间表现无显著差异,但是两者均与0.4μmol/L、0.5 μmol/L在水平间差异显著。引物的浓度高低会影响产物的特异性,最佳引物浓度一般在0.1~0.5μmol/L,以最低引物量产生所需要的结果为最佳,引物浓度偏高会引起错配和非特异性扩增,且会增加引物之间形成二聚体的机会。因此,应选择拐点0.3μmol/L为最佳水平(图3)。
2.2.3 模板DNA浓度对PCR扩增结果的影响模板DNA的浓度和纯度对PCR扩增结果都会有一定的影响,DNA浓度过低,可能导致扩增结果不稳定及扩增条带模糊;浓度过高,则可能使引物或dNTPs过早耗尽,底物过量扩增,过早进入线性阶段,出现扩增结果不稳定的假象。如图3所示,当DNA取峰值30 ng/25μL时,反应体系稳定,在保证扩增效果的前提下,应尽量降低模板DNA的用量,因此,最终选定30 ng/25μL作为本实验的最佳反应水平。

2.2.4 Mg2+浓度对PCR扩增结果的影响Mg2+浓度影响PCR扩增的多个方面,如DNA聚合酶的活性,这会影响PCR扩增的产量;再如引物退火,这会影响扩增产物的特异性。dNTPs和模板DNA同Mg2+结合,降低了酶活性所需要的游离Mg2+的量。最佳的Mg2+浓度对于不同的引物和模板都不同。如图3所示,Mg2+浓度在2.0 mmol/L与2.5 mmol/L水平间差异不显著,与其他两个浓度在水平间差异显著。较高的游离Mg2+浓度会增加非特异性扩增,降低忠实性;Mg2+浓度过低会降低Taq酶的活性,使反应产物减少。综合各因素最终确定2.0 mmol/L为最佳水平。
2.2.5 Taq酶浓度对PCR扩增结果的影响 在100μL的典型PCR反应体系中,Taq聚合酶的用量一般为1~2.5单位,Taq聚合酶含量偏少时PCR产物相应减少,而Taq聚合酶含量过高时则会增加非特异性扩增的机会。如图3所示,Taq聚合酶各水平间差异均不明显,说明Taq聚合酶在本实验所选梯度范围内对PCR扩增反应结果影响较小。由图2可知,当Taq聚合酶浓度为1.5 U时,反应体系不稳定。因此,本实验最终选定Taq聚合酶浓度2.5 U/25μL作为最佳反应水平。
2.2.6 SRAP-PCR扩增最佳反应体系 通过正交设计实验,得到用于酵母菌种分类的SRAPPCR扩增反应的最佳体系。25μL反应体系包括:dNTPs 150μmol/L,引物0.3μmol/L,模板DNA 30 ng,Mg2+2.0 mmol/L,Taq聚合酶2.5 U。

表4 SRAP-PCR正交试验设计极差分析/L16(45)Table 4 The range analysis of SRAP-PCR orthogonal design/L16(45)
2.3 SRAP扩增结果
在最适的SRAP扩增条件下,对供试的19株酵母菌的DNA进行了14对引物的SRAP扩增实验,经重复试验(3次)扩增结果稳定。部分引物组合对供试菌株的扩增结果见图4~图8。

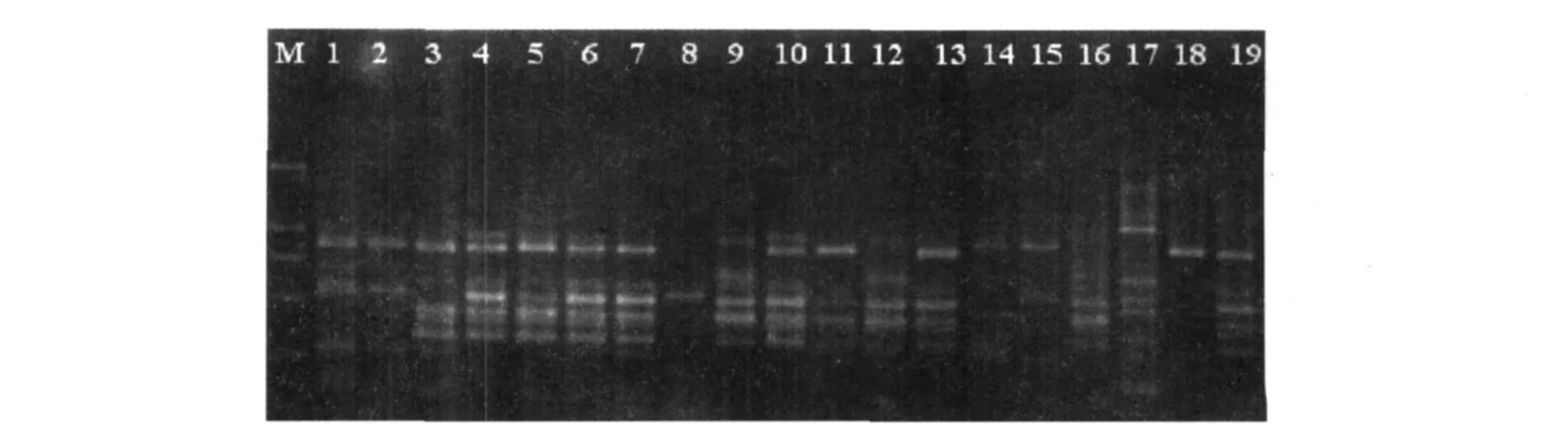
图8 引物me4-em9对供试菌株的扩增结果Fig.8 Amplification products generated from tested strainswith primerme4-em9
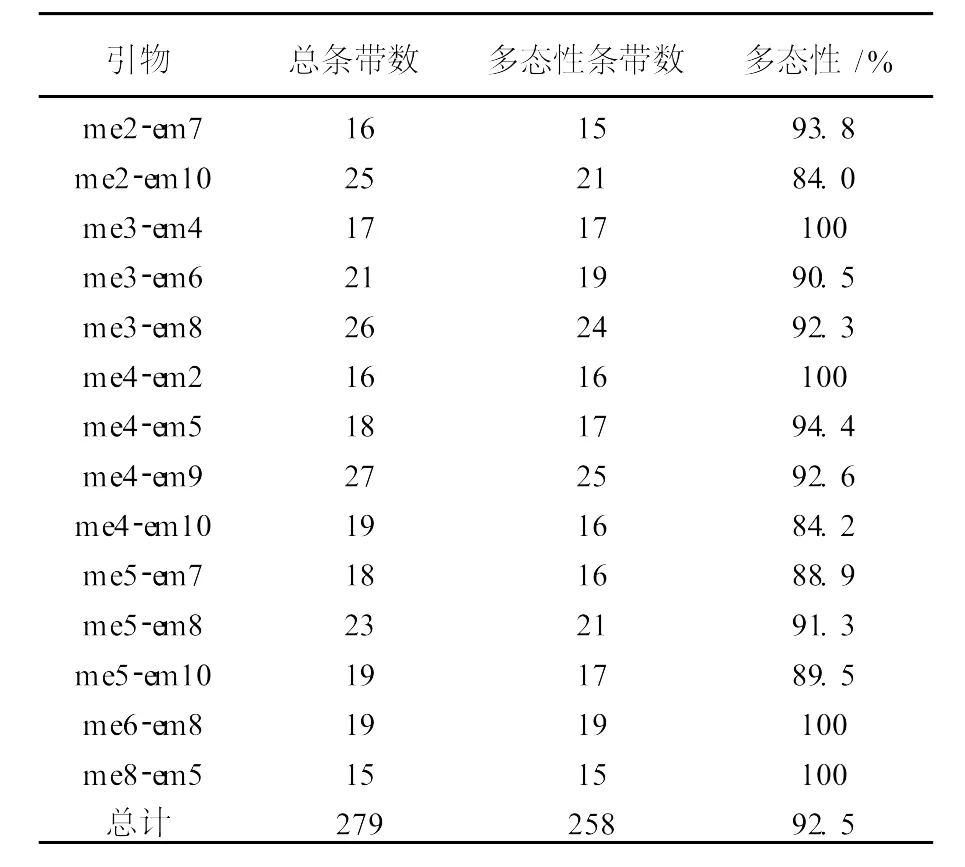
表5 19株酵母菌株SRAP扩增的总条带数和多态性条带数Table 5 Total bands and polymorphic bands resulting from SRAP
14对引物共扩增出279条带,其中92.5%呈多态性(见表5),每对引物平均扩增出20条带。引物组合me4-em9扩增出来的总条带最多为27条,多态性条带为25条。引物组合me3-em4、me4-em2、me6-em8和me8-em5扩增出来的多态性条带比例最高,都达到了100%,可以将19株酵母菌株完全区别开来。以上述14对引物扩增的产物图谱为依据,对供试的19株酵母菌株进行聚类分析。
2.4 SRAP扩增产物聚类分析
采用NTSYSpc软件中的S IMQUA I程序进行数据分析计算两两菌株间的遗传相似系数。供试的19个酵母菌株相似系数范围为0.60~1.00。与ISSR的鉴定结果类似,Y-12酵母与其他酵母的相似系数较低。相似系数最高的菌株是Y-2和Y-4,两者的相似系数达到了1.00。

图9 SRAP聚类分析树形图Fig.9 Dendrogram based on SRAP fingerprints
如图9所示,在相似水平为71%处时,供试的19株酵母菌株被划分为4个类群。第Ⅰ群:Y-6、Y-18、Y-14、Y-7、Y-2、Y-4;第Ⅱ群:Y-12;第Ⅲ群:Y-3、Y-11、Y-16、Y-5、Y-8、Y-13、Y-17、Y-1、Y-19、Y-10、Y-9;第Ⅳ群:Y-15。
3 讨 论
SRAP是一种新型的分子标记技术,已经成功地应用在多种植物的研究中,它适于在不同作物上用于各种目的的研究[12]。对于SRAP分子标记技术而言,不同的材料所需要的PCR反应体系是不同的,为了让所选菌种扩增出理想的产物,必须优化PCR反应体系[13]。模板浓度、dNTPs浓度、Mg2+浓度和退火温度等因素都会对扩增结果产生影响,本次试验中dNTPs浓度和引物浓度是主要影响因素。SRAP扩增获得的条带十分丰富,该分子标记技术的引物扩增产物为基因区域,这意味着将不能揭示非基因区域的遗传多样性[12]。形态上有明显差异的菌株,SRAP扩增条带也有明显区别,但是,形态上相似的菌株之间谱带差异很小,种内菌株之间几乎是没有差异的。对于SRAP分子标记技术而言,引物组合的选择是关键,正向引物和反向引物的自由组合不仅增加了引物的多样性,而且节省了引物合成的成本,本文中使用的SRAP引物是目前文献中报道的部分标准引物序列[13-14]。本研究结果显示,不同的SRAP引物组合对不同酵母菌种间的区分度有所不同。在利用该标记技术进行分类鉴定及亲缘关系研究时,应尽量选用多种引物进行扩增反应,在综合大量谱带信息的基础上在进行聚类分析。
本研究利用SRAP分子标记获得的多态性条带,对实验室分离纯化得到的19株酵母菌在一定的程度上进行区分,比较适合于酵母菌的种间鉴别。要将这19株酵母菌准确鉴定到种的水平,还需结合其他辅助手段一起进行,如26S r DNA D1/D2区序列分析和5.8S rDNA-ITS区域的PCRRFLP分析等,每种技术手段都有各自的优缺点,多种技术手段的结合才能大大提高酵母菌种鉴定的准确性。本研究对SRAP分子标记技术的成功应用为今后酵母菌属真菌的种系鉴定、遗传多样性研究、指纹图谱构建以及遗传连锁图谱构建等奠定了重要基础。
[1] Li G,Quiros C F.Sequence related amplified polymorphism(SRAP),A new marker system based on simple PCR reaction:its application to mapping and genetagging in Brossica[J].Theor.Appl.Genet.,2001,103:455-461.
[2] Ferrlol M,Pico B,Nuez F.Genetic diversity of a germplasm collection of Cucurbita pepousing SRAP and AFLP markes[J].Theor.Appl.Genet.,2003,107(2):271-282.
[3] 林忠旭,张献龙,聂以春,等.新型标记SRAP在棉花F2分离群体及遗传多样性评价中的适用性分析[J].遗传学报,2004,31(6):622-626.
[4] 林忠旭,张献龙,聂以春,等.棉花SRAP遗传连锁图构建[J].科学通报,2003,48(15):1676-1679.
[5] 张安世,邢智峰,刘永英,等.SRAP分子标记及其应用[J].安徽农业科学,2007,35(9):2562-2563.
[6] 应正河,吴小平,谢宝贵,等.香菇SRAP反应体系的优化[J].食用菌学报,2006,13(4):1-5.
[7] 董星光,樊丽,王志刚,等.梨SRAP体系的正交优化研究[J].江苏农业科学,2009,2:51-53.
[8] 李晓慧,田朝阳,王从彦.SRAP分子标记分析西瓜遗传多态性[J].生物技术,2007,617(3):23-26.
[9] 李翠翠,郭立忠,卢伟东,等.RAPD和SRAP分子标记在真姬菇菌种鉴定中的应用[J].食用菌报,2009,16(1):21-25.
[10] 杨水云,李续娥,吴明宇,等.正交实验法在PCR反应条件优化中的应用[J].生物数学学报,2005,20(2):202-206.
[11] 沙伟,滕兆岩,倪红伟.正交设计优化星星草ISSR-PCR反应体系研究[J].中国草地学报,2006,28(6):52-55.
[12] 杨迎花,李先信,曾柏全,等.新型分子标记SRAP的原理及其研究进展[J].湖南农业科学,2009,(5):15-17,20.
[13] 谭碧玥,王源秀,徐立安.杨树基因组SRAP扩增体系的建立与优化[J].林业科技开发,2009,23(2):25-29.
[14] RiazA,Li G,Quresh Z,et al.Genetic diversity of oilseed Brassica napus inbred lines based on sequence-related amplified polymorphis m(SRAP)and its relation to hybrid performance[J].Plant Breeding,2001,120(5):411-415.

