利用种特异性PCR快速鉴定新疆酸马奶优势菌群
赵潞,张列兵,张世湘,郝彦玲
(中国农业大学 食品科学与营养工程学院,北京 100083)
利用种特异性PCR快速鉴定新疆酸马奶优势菌群
赵潞,张列兵,张世湘,郝彦玲
(中国农业大学 食品科学与营养工程学院,北京 100083)
建立了一种快速准确鉴定酸马奶中乳酸菌优势菌群的种特异性PCR技术。据资料报道,酸马奶中已知的6种优势菌主要集中在植物乳杆菌、干酪乳杆菌和嗜酸乳杆菌3大类群。首先,针对属于同一类群内的两种优势菌设计2对种特异性引物,采用3组PCR实验验证引物的特异性。然后以酸马奶中提取的总DNA为模板,利用经验证的6对种特异性引物鉴定2份新疆酸马奶样品中的优势菌群。结果表明,新疆酸马奶中含有Lactobacillus(L.)helveticus,L.pentosus,L.plantarum,L.acidophilus和L.paracasei,与前人报道结果一致。 本研究建立的种特异性PCR技术不仅能够快速鉴定酸马奶中乳酸菌优势菌群组成,同时可为其它发酵制品中3大类群乳酸菌鉴定提供有效的方法。
种特异性PCR;酸马奶;乳酸菌;优势菌群
0 引 言
酸马奶是以鲜马奶为原料,经乳酸菌和酵母菌等微生物共同发酵而成的乳饮料,主要饮用于新疆和内蒙古等地。现代医学证实,酸马奶具有降血压、降血脂和调节胃肠道等功能[1]。其中,优势乳酸菌菌群在其医疗保健功能中发挥关键作用。因此,酸马奶中乳酸菌多样性成为目前研究热点[2,3]。
目前,乳制品中乳酸菌多样性研究可分为依赖培养的传统方法和非培养的分子方法。依赖培养的传统方法仅对可培养的活菌进行分离,而且纯化及生理生化鉴定费时费力,且结果易与样品本身乳酸菌多样性存在偏差[4]。与之相比,作为非培养方法的种特异性PCR技术不仅快速准确,而且容易普及。
据资料报道,酸马奶中优势菌主要集中在16S rRNA序列无法鉴定的植物乳杆菌,干酪乳杆菌和嗜酸乳杆菌类群[5,6]。因此,为了准确区分每个类群中乳酸菌,本研究设计6对种特异性引物,以酸马奶中提取总DNA为模板,建立了一种快速鉴定酸马奶优势菌群的种特异性PCR方法。
1 实 验
1.1 材料
(1) 菌 株 来 源 :L.plantarum 05-19,L.paracasei E4213,L.helveticus AS1.1877T,L.acidophilus La3。L.casei.subsp.casei ATCC393,L.pentosus ATCC8041。
(2)酸马奶样品:实验所检测的两份酸马奶样品均采自新疆伊犁地区新源县那拉提夏牧场的哈萨克牧民家庭。
(3)试剂和仪器:细菌基因组DNA提取试剂盒以及PCR相关试剂。PCR仪(PTC-200型)。
1.2 方法
1.2.1 乳酸菌基因组DNA提取
将乳酸菌以质量浓度为10 g/L接种量接种于MRS培养基中,37℃培养OD600至0.6~0.8,离心收集菌体,按照试剂盒说明书的步骤进行提取,采用质量浓度为10 g/L琼脂糖凝胶电泳进行检测。
1.2.2 酸马奶样品细菌总DNA提取
酸马奶样品总DNA提取参照Baradei等[7]方法,并进行改进。5 mL酸马奶样品与5 mL质量浓度为20 g/L柠檬酸钠溶液振荡混匀后,加10 mg链霉蛋白酶和20 μL β-巯基乙醇,50℃水浴3 h。菌体经洗涤后,利用溶菌酶结合SDS变性剂裂解菌体,采用酚仿和异丙醇分离纯化总DNA,经琼脂糖电泳检测后,于-20℃保存备用。
1.2.3 种特异性PCR引物及扩增条件
本实验采用的6种乳酸菌种特异性PCR引物及扩增条件如表1所示。
1.2.4 PCR验证及优势菌群的鉴定

表1 种特异性PCR引物及扩增条件
为了考察每一类群中文献推荐引物的特异性,设置3组种特异性引物的PCR实验进行验证。首先以目标菌种基因组为模板(阳性对照)和属于同一类群的菌种基因组为模板(阴性对照)两组PCR验证引物特异性。其次,以同一种群内的两菌种的混合基因组为模板进行PCR(各0.5 ng),验证在同类群菌的干扰下,引物的种特异性。最后,以酸马奶样品总DNA为模板,分别采用植物乳杆菌类群、干酪乳杆菌类群和嗜酸乳杆菌类群的6对特异性引物进行扩增,利用质量浓度为20 g/L琼脂糖凝胶电泳检测酸马奶中优势菌群的组成。
2 结果与分析
2.1 种特异性引物的PCR验证
2.1.1 植物乳杆菌类群的种特异性PCR验证
为了验证植物乳杆菌类群中引物的特异性。首先以植物乳杆菌为阴性对照,以戊糖乳杆菌基因组和混合基因组为阳性对照,3组PCR验证戊糖乳杆菌的引物特异性,结果如图1所示。由图1可以看出,阳性对照泳道2和4出现与预期片段218 bp一致的条带,而阴性对照未出现相应条带;其次以戊糖乳杆菌基因组为阴性对照,以植物乳杆菌基因组和混合基因组为阳性对照,3组PCR验证植物乳杆菌的引物特异性,从图1可见,阳性对照泳道6和8出现与预期片段318 bp一致的条带,而阴性对照未出现相应条带。结果表明,所用两对引物能很好的区分同属于植物乳杆菌类群内的植物乳杆菌和戊糖乳杆菌。

图1 植物乳杆菌类群特异性检测结果
图1中,M为DL2000 Marker(下同);1~4为戊糖乳杆菌特异性PCR;模板分别:1为ddH2O;2为戊糖乳杆菌基因组;3为植物乳杆菌基因组;4为植物乳杆菌和戊糖乳杆菌混合基因组;5~8为植物乳杆菌特异性PCR;5为ddH2O;6为植物乳杆菌基因组;7为戊糖乳杆菌基因组;8为植物乳杆菌和戊糖乳杆菌混合基因组。
2.1.2 干酪乳杆菌类群的特异性验证
为了验证干酪乳杆菌类群中引物的特异性,实验设计思路如2.1.1所述。从图2可见,阳性对照泳道2和4、6和8分别出现与干酪乳杆菌和副干酪乳杆菌预期大小一致的条带,均为290 bp,而阴性对照均未出现相应条带,实验结果说明所用的两对引物能很好的区分同属干酪乳杆菌类群中的干酪乳杆菌和副干酪乳杆菌。
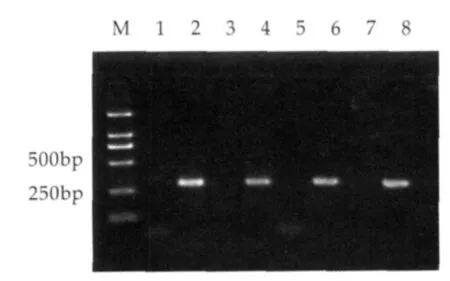
图2 干酪乳杆菌类群特异性检测结果
图2中,1~4为干酪乳杆菌特异性PCR;模板分别:1为ddH2O;2为干酪乳杆菌基因组;3为副干酪乳杆菌基因组;4为干酪乳杆菌和副干酪乳杆菌混合基因组;5~8为副干酪乳杆菌特异性PCR;模板分别:5为ddH2O;6为副干酪乳杆菌基因组;7为干酪乳杆菌基因组;8为副干酪乳杆菌和干酪乳杆菌混合基因组。
2.1.3 嗜酸乳杆菌类群特异性验证
为了验证嗜酸乳杆菌类群中引物的特异性,实验设计思路如2.1.1所述。图3为干酪乳杆菌类群特异性检测结果。由图3可以看出,阳性对照泳道2,4,6和8分别出现与瑞士乳杆菌和嗜酸乳杆菌预期大小一致的条带,分别为524 bp和740 bp,而阴性对照均未出现相应条带,实验结果说明所用的两对引物能很好的区分同属嗜酸乳杆菌类群中的嗜酸乳杆菌和瑞士乳杆菌。

图3 干酪乳杆菌类群特异性检测结果
图3中,1~4为 瑞 士 乳 杆 菌 特 异 性PCR;1为ddH2O;2为瑞士乳杆菌基因组;3为嗜酸乳杆菌基因组;4为瑞士乳杆菌和嗜酸乳杆菌混合基因组;5~8为嗜酸乳杆菌特异性PCR;5为ddH2O;6为 嗜酸乳杆菌基因组;7为瑞士乳杆菌基因组;8为瑞士乳杆菌和嗜酸乳杆菌混合基因组。
2.1.4 利用PCR方法鉴定酸马奶中的优势乳酸菌
以2份酸马奶总DNA为模板,进行3大类群的特异性PCR扩增。图4为种特异性PCR方法鉴定1号酸马奶样品中优势乳酸菌。由图4可以看出,1号样品中含有L.pentosus,L.plantarum,L.helveticus和L.acidophilus。 图5为种特异性PCR方法鉴定2号酸马奶样品中优势乳酸菌。由图5可以看出,2号样品中含有L.pentosus,L.helveticus,L.paracasei。 孙天松等[2-3,5]用传统分离鉴定方法对酸马奶中的乳酸菌组成进行研究,结果表明优势菌主要有L.acidophilus,L.plantarum,L.pentosus,L.casei和L.helveticus,这与本研究结果一致。因此,本研究建立的种特异性PCR技术不仅能够快速鉴定酸马奶中乳酸菌优势菌群组成,同时可为其他发酵制品中3大类群乳酸菌的鉴定提供行之有效的方法。

图4 PCR方法鉴定1号酸马奶样品中优势乳酸菌

图5 PCR方法鉴定2号酸马奶样品中优势乳酸菌
图4中,特异性引物分别:1为戊糖乳杆菌;2为植物乳杆菌;3为干酪乳杆菌;4为副干酪乳杆菌;5为瑞士乳杆菌;6为嗜酸乳杆菌。图5中,特异性引物分别:1为戊糖乳杆菌;2为植物乳杆菌;3为干酪乳杆菌;4为副干酪乳杆菌;5为瑞士乳杆菌;6为嗜酸乳杆菌。
3 结果与讨论
目前,酸马奶中乳酸菌生物多样性的研究皆采用依赖培养的传统方法,资料报道酸马奶中乳酸菌的6种优势菌分别属于乳酸菌的3个类群,其中,L.acidophilus和L.helveticus属于嗜酸乳杆菌类群,L.plantarum和L.pentosus属于植物乳杆菌类群,L.casei和L.paracasei属于干酪乳杆菌类群[12]。由于三大类群内菌种之间的生理生化特性和16S rRNA基因序列都高度相似,因此无法精确鉴定类群内菌种[10]。
种特异性PCR技术是根据每物种rDNA上可变区或编码基因的特有序列设计引物,进行物种鉴定的方法,此方法的关键是引物的选取。本研究建立的种特异性PCR方法可用于鉴定培养方法分离的3大类群中的菌株,是对传统乳酸菌鉴定方法的一个重要补充。此外,与依赖培养的传统方法相比,原来需要几周完成的菌种鉴定工作,种特异PCR技术在12 h内即可结束,是一种简单,快速的检测手段。
变性梯度凝胶电泳(DGGE)技术是一种不依赖于培养过程而对样品中微生物组成直接进行鉴定的分子生物学技术,其理论依据为片段大小相同、碱基组成不同的双链DNA分子,在变性凝胶梯度电泳时因具有不同的解链温度而滞留于凝胶的不同位置,最终形成相互分开的谱带[13]。目前,DGGE技术常用的靶基因是16S rRNA V3区。但由于3大类群内菌株V3区序列高度相似,DGGE时会发生共迁移现象,导致无法根据Marker比对和测序的方法鉴定菌种,需要通过种特异性PCR方法进一步鉴定[4]。因此,本研究建立的种特异性PCR方法也是DGGE技术鉴定共迁移菌种的一个重要环节。
[1]哈斯苏荣,阿木古楞,芒来.酸马奶及其医学价值[J].中国中药杂志,2003,28(1):11-14.
[2]孙天松,王俊国,张列兵,等.中国新疆地区酸马奶中乳酸菌生物多样性研究[J].微生物学通报,2007,34(3):451-454.
[3]孟和毕力格,乌日娜,王立平,等.不同地区酸马奶中乳杆菌的分离及其生物学特性的研究[J].中国乳品工业,2004,32(11):6-11.
[4]OGIER J C,LAFARGE V,GIRARD V,et al.Molecular Fingerprinting of Dairy Microbial Ecosystems by Use of Temporal Temperature and Denaturing Gradient Gel Electrophoresis[J].Applied Environmental Microbiology,2004,70:5628–5643.
[5]ISHII S,KIKUCHI M,TAKAO S.Isolation and Identification of Lactic Bacteria and Yeasts from“Chigo”in Inner Mongolia,China[J].Animal Science and Technology,1997,68:325-329.
[6]SINGH S,GOSWAMI P,SINGH R,et al.Application of Molecular Identification Tools for Lactobacillus,with a Focus on Discrimination between Closely Related Species:A Review[J].LWT-Food Science and Technology,2009,42:448-457.
[7]EL-BARADEI G,DELACROIX-BUCHET A,OGIER J C.Bacterial Biodiversity of Traditional Zabady Fermented Milk[J].International Journal of Food Microbiology,2008,121:295-301.
[8]TORRIANI S,FELIS G E,DELLAGLIO F.Differentiation of Lactobacillus plantarum,L.pentosus and L.paraplantarum by recA Gene Sequence Analysis and Multiplex PCR Assay with recA Gene-Derived Primers[J].Applied Environmental Microbiology,2001,67:3450–3454.
[9]FORTINA M G,RICCI G,MORA D,et al.Specific Identification of Lactobacillus helveticus by PCR with pepC,pepN and htrA Targeted Primers[J].FEMS Microbiology Letters,2001,198:85-89.
[10]WALTER J,TANNOCK G W,TILSALA-TIMISJARVI A,et al.Detection and Identification of Gastrointestinal Lactobacillus species by Using Denaturing Gradient Gel Electrophoresis and Species-specific PCR Primers[J].Applied Environmental Microbiology,2000,66:297–303.
[11]WARD L J H,TIMMINS M J.Differentiation of Lactobacillus casei,Lactobacillus paracasei and Lactobacillus rhamnosus by Polymerase Chain Reaction[J].Letters in Applied Microbiology,1999,29:90–92.
[12]马凯,李春玲,程池.Lactobacillus的系统发育分析及分群[J].食品与发酵工业,2007,33(2):27-35.
[13]马俊孝,孔健.PCR-DGGE技术在乳酸菌检测中的条件研究[J].食品科学,2007,28(1):225-229.
Rapid identification of the dominant LAB in fermented Koumiss produced in Xinjiang using species-specific PCR
ZHAO Lu,ZHANG Lie-bing,ZHANG Shi-xiang,HAO Yan-ling
(College of Food Science and Nutritional Engineering,China Agricultural University,Beijing100083,China)
In order to identify the dominant lactic acid bacteria(LAB)in koumiss rapidly and precisely,a species-specific PCR method was established.It was reported that six dominant LAB species existed in koumiss,which belong to L.plantarum group,L.casei group and L.acidophilus group.First,two pairs of species-specific primers were designed to distinguish the dominant strains belonging to the same group,and their specificity was respectively determined by three PCR tests.Then,the total DNA extracted from samples was used as template,and the dominant LAB of two koumiss products from Xinjiang were identified by the six pairs of species-specific primers.The results showed that L.helveticus,L.pentosus,L.plantarum,L.acidophilus and L.paracasei were presented,which was consisted with previously reports.In addition to analyze the dominant LAB in koumiss rapidly,this species-specific PCR method also can identify the LAB belonging to the three groups in other fermented products.
species-specific PCR;Koumiss;LAB;dominant bacteria
Q93-331
A
1001-2230(2010)01-0012-03
2009-07-16
国家自然科学基金项目(30671483),国家高技术研究与发展计划(863计划)项目(2007AA10Z354,2006AA10Z317)。
赵潞 (1985-),男,硕士研究生,研究方向为乳品微生物。
郝彦玲

